最近涉及到了磁共振图像MRI的重建,网络上相关的实现比较少,因此进行实现记录。
磁共振图像MRI重建实现
1.配置代码环境
这里介绍一个很好的开源项目,git为: https://github.com/NKI-AI/direct.git。
安装环境为:
(1)CUDA ≥ 10.2 supported GPU.
(2)Linux with Python ≥ 3.8
(3)PyTorch ≥ 1.6
可以使用Docker或Conda配置环境,这里以为Conda为例,很慢的话,可以-i 清湖镜像源:
conda create -n myenv python=3.9
conda activate myenv
pip3 install torch torchvision torchaudio --extra-index-url https://download.pytorch.org/whl/cu116
git clone https://github.com/NKI-AI/direct.git
cd direct
python3 -m pip install -e ".[dev]"2.MRI数据集处理
这里使用公开数据集,常用的为The Multi-coil Calgary-Campinas数据集,链接为: https://sites.google.com/view/calgary-campinas-dataset/download;以及FastMRI数据集,链接为: https://fastmri.med.nyu.edu/。
这里我以为FastMRI数据集中的Knee MRI为例,数据集很大,因此我下载val为例。
下载后,val文件夹一共199个.h5文件。将其划分training和validation文件夹。
下面提供一个 划分的代码。
javascript
import os
import shutil
import random
def split_data(source_dir, train_dir, val_dir, split_ratio=0.8):
# 确保源目录存在
if not os.path.exists(source_dir):
print(f"Source directory {source_dir} does not exist.")
return
# 创建训练和验证目录
os.makedirs(train_dir, exist_ok=True)
os.makedirs(val_dir, exist_ok=True)
# 获取所有.h5文件
files = [file for file in os.listdir(source_dir) if file.endswith('.h5')]
random.shuffle(files) # 打乱文件顺序
# 计算划分点
split_point = int(len(files) * split_ratio)
# 划分文件
train_files = files[:split_point]
val_files = files[split_point:]
# 复制文件到新的目录
for file in train_files:
shutil.copy(os.path.join(source_dir, file), train_dir)
for file in val_files:
shutil.copy(os.path.join(source_dir, file), val_dir)
print(f"Files split into {len(train_files)} training and {len(val_files)} validation.")
# 路径配置
source_directory = 'singlecoil_val' # 源文件夹路径
training_directory = os.path.join(os.path.dirname(source_directory), 'training') # 训练集文件夹路径
validation_directory = os.path.join(os.path.dirname(source_directory), 'validation') # 验证集文件夹路径
# 执行数据划分
split_data(source_directory, training_directory, validation_directory)将singlecoil_val文件夹进行了training和validation文件夹的划分。
3.配置数据集以及模型文件
划分好数据集后,进行config.py文件的配置。
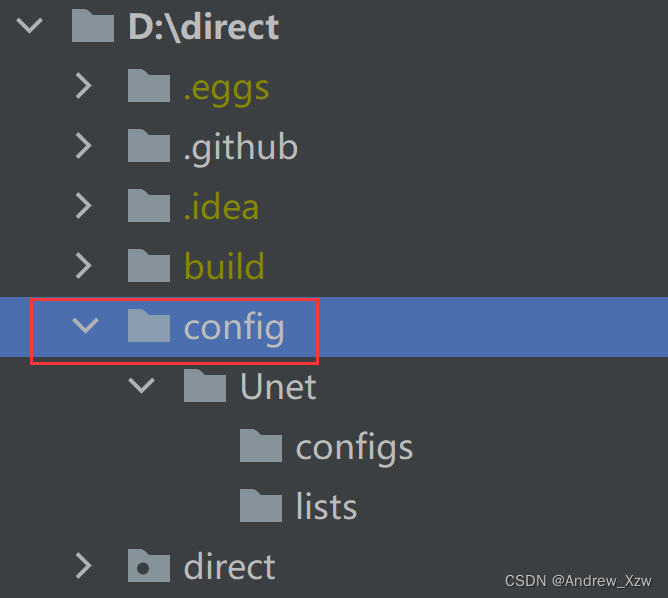
在代码根路径下新建一个config文件夹,该文件夹下以选用的模型为文件夹名进行独立配置,这里以UNet为为例。
随后,UNet下新建configs文件夹(用于存放实例化Unet模型以及数据集的配置文件)以及lists文件夹(用于存放划分好的文件夹的名字,类似于目标检测中Yolo系列的训练)。
新建好,文件夹层级关系如下:
接下来,在configs文件下新建一个Unet的 base_unet.yaml文件。这里我附上我自己(其余的模型可以参考代码根路径下的projects文件夹,不同的数据集以及模型的配置文件均不同。)
这里的字典形式的yaml文件,先找training和validation;
然后更改datasets对应的name以及filenames_lists和masking对应的name;
由于我是用的是FastMRI数据集,所以datasets对应的name更改为:FastMRI;masking对应的name更改为:FastMRIRandom。
javascript
physics:
forward_operator: fft2
backward_operator: ifft2
training:
datasets:
- name: FastMRI
filenames_lists:
- ../lists/train.lst
transforms:
cropping:
crop: null
sensitivity_map_estimation:
estimate_sensitivity_maps: true # Estimate the sensitivity map on the ACS
normalization:
scaling_key: masked_kspace # Compute the image normalization based on the masked_kspace maximum
masking:
name: FastMRIRandom
accelerations: [4]
center_fractions: [0.08]
batch_size: 1 # This is the batch size per GPU!
optimizer: Adam
lr: 0.002
weight_decay: 0.0
lr_step_size: 30000
lr_gamma: 0.2
lr_warmup_iter: 100
num_iterations: 150000
gradient_steps: 1
gradient_clipping: 0.0
gradient_debug: false
checkpointer:
checkpoint_steps: 500
validation_steps: 2500
loss:
crop: header
losses:
- function: l1_loss
multiplier: 1.0
- function: ssim_loss
multiplier: 1.0
validation:
datasets:
# Twice the same dataset but a different acceleration factor
- name: FastMRI
filenames_lists:
- ../lists/val.lst
transforms:
cropping:
crop: null
sensitivity_map_estimation:
estimate_sensitivity_maps: true # Estimate the sensitivity map on the ACS
normalization:
scaling_key: masked_kspace
masking:
name: FastMRIRandom
accelerations: [4]
center_fractions: [0.08]
text_description: 4x # Description for logging
crop: header # This sets the cropping for the DoIterationOutput
metrics: # These are obtained from direct.functionals
- fastmri_psnr
- fastmri_ssim
model:
model_name: unet.unet_2d.Unet2d
num_filters: 32
image_initialization: SENSE
additional_models:
sensitivity_model:
model_name: unet.unet_2d.UnetModel2d
in_channels: 2
out_channels: 2
num_filters: 8
num_pool_layers: 4
dropout_probability: 0.0
logging:
tensorboard:
num_images: 4这里重点要说的是filenames_lists,这个.lst类似于.txt文件,里面存放一些划分好的文件名。这里附上读取划分好数据集的文件夹来生成对应的.lst文件。
javascript
import os
def save_folder_names_to_file(directory, output_file):
# 检查指定的目录是否存在
if not os.path.exists(directory):
print("指定的目录不存在")
return
# 打开输出文件,准备写入
with open(output_file, 'w') as file:
# 使用os.walk遍历目录
for folder_name in os.listdir(directory):
# 将文件夹名写入文件,每个名字一行
file.write(folder_name + '\n')
# 调用函数,你需要替换'directory_path'和'output_file_path'为你的实际路径
save_folder_names_to_file(r'D:\direct\MRI_dataset\training', r'D:\direct\config\Unet\lists\train.lst')
save_folder_names_to_file(r'D:\direct\MRI_dataset\validation', r'D:\direct\config\Unet\lists\val.lst')处理好的文件夹如下:
其中,lists文件夹的.lst文件可视化为:

5.训练
下面是 训练的脚本。
direct train <experiment_directory> --training-root <training_data_root> --validation-root <validation_data_root> \
--num-gpus <number_of_gpus> --cfg <path_or_url_to_yaml_file> [--other-flags]<experiment_directory>可以在根目录下新建一个output文件夹,用于存放模型的配置以及训练的日志;
<training_data_root> 就是指定到我们上面划分好的数据集的training的路径;
<validation_data_root> 就是指定到我们上面划分好的数据集的validation的路径;
<number_of_gpus>根据自己的算力资源进行调度分配,eg.1;
<path_or_url_to_yaml_file>就是指定到我们上面配置好的模型的yaml文件的路径;
--other-flags\]没有特殊需求,可省略。