小罗碎碎念
文献日推主题:人工智能在肿瘤亚型分类领域的研究进展
昨天晚上在研究鼻咽癌的病理学诊断指南,看到了下面这段话的时候,我问了自己一个问题------通过AI识别出肿瘤亚型的根本目的是什么?可以衔接哪些具体的下游任务?

为了解决我自己提出的问题,于是便有了今天这期推文。
AI识别肿瘤亚型的用途
现在回答开头我提出的问题------癌症区分不同亚型的意义在于,它有助于实现对癌症的精准诊断、预后分层、肿瘤分期、指导治疗、复发监控及药物研发。
肿瘤分子分型(molecular classification)通过分子分析技术为肿瘤进行分类,使肿瘤分类从传统的形态学转向以分子特征为基础的分子分型。这种分型方式可以深入剖析肿瘤分子水平的异质性,从而实现针对不同亚型的精准治疗 。
例如,在肺癌领域 ,2015年世界卫生组织(WHO)的肺肿瘤分类,与2004年版相比,发生了较大的变化,整合了肿瘤学、分子生物学、病理学、放射学和外科学等各个领域的研究成果,使得病理学分类能够更好地服务于临床实践及临床/基础研究 。在乳腺癌方面,分子亚型直接决定了患者后续的用药方案,例如HER2阳性患者可通过HER2信号阻断治疗,HR阳性患者可通过内分泌阻断和细胞周期阻断治疗,三阴性乳腺癌可考虑PARP抑制剂或免疫治疗 。
此外,肿瘤分子分型还有助于识别肿瘤的遗传性、个体差异性和分子机制的复杂性,为个体化的靶向治疗提供了基础。通过基因芯片、二代测序等分子生物学技术及系统生物学的发展,为形态学分型向更为精准的分子分型转变提供了技术支持 。
一、基于隐藏基因组分类器的肝内胆管癌精准医学研究

一作&通讯
| 作者类型 | 姓名 | 单位 | 单位中文翻译 |
|---|---|---|---|
| 第一作者 | Yi Song | Department of Surgery, Memorial Sloan Kettering Cancer Center, New York, New York. | 纪念斯隆-凯特琳癌症中心外科部门,纽约,纽约州 |
| 通讯作者 | William Jarnagin | Department of Surgery, Memorial Sloan Kettering Cancer Center, 1275 York Avenue,C-891, New York,NY 10065. | 纪念斯隆-凯特琳癌症中心外科部门,纽约,纽约州 |
文献概述
这篇文章是关于一种新方法来量化肝内胆管癌(Intrahepatic Cholangiocarcinoma, IHC)的异质性,该方法使用了一种名为"hidden-genome classifier"的监督式机器学习算法来提高肿瘤分类的准确性。
这篇论文介绍了一种新的方法来量化肝内胆管癌(IHC)的异质性,称为"隐藏基因组分类器"。
-
研究背景:
- 问题:肝内胆管癌(IHC)是一种异质性肿瘤,其复杂的遗传、组织学和临床特征使得治疗和临床试验的解释面临挑战。
- 难点:IHC的异质性导致其治疗反应差异大,难以通过传统的组织学分类来准确预测患者的预后。
- 相关工作:现有研究主要集中在IHC的罕见遗传变异上,未能全面捕捉其基因组特征。需要一种新的方法来整合和分类这些复杂的遗传数据。
-
研究方法:
- 本研究采用回顾性分析,纳入了1370例IHC、肝外胆管癌(EHC)、胆囊癌(GBC)、肝细胞癌(HCC)或双表型肿瘤患者。
- 使用隐藏基因组模型,基于遗传相似性将527例IHC分类为与EHC/GBC或HCC相似的类别。
- 隐藏基因组算法整合了包括常见和罕见遗传变异、突变背景和拷贝数变异(CNV)在内的遗传元特征数据,并通过机器学习进行分类。
- 具体步骤包括:从MSK-IMPACT平台获取基因组数据,使用lasso回归选择活跃的遗传预测因子,通过主成分分析和Uniform Manifold Approximation and Projection (UMAP)降维技术进行可视化。
-
实验设计:
- 患者队列包括2003年至2022年在纪念斯隆-凯特琳癌症中心接受MSK-IMPACT靶向肿瘤测序的患者。
- 数据包括人口统计学、临床病理学和结果数据,从前瞻性维护的数据库中获取,并通过医学记录审查补充。
- 遗传元特征数据从cBioPortal数据库中检索。
-
结果与分析:
- 研究发现,78%的IHC(410例)与EHC/GBC有超过50%的遗传相似性,23%(122例)有超过90%的相似性("胆道类"),5.7%(30例)有超过90%的相似性("HCC类")。
- 胆道类IHC的中位总生存期(OS)为1年,而不适合手术切除的IHC为1.8年,适合手术切除的IHC为2.4年。
- HCC类IHC的中位OS为5.1年。
- 隐藏基因组分类器预测的OS独立于FGFR2和IDH1变异。相比之下,组织学亚型并不能预测OS。
-
总体结论:
- 这篇论文展示了隐藏基因组分类器在IHC分类中的首次应用,揭示了IHC的遗传特征从胆道样到HCC样的连续谱系。
- 该分类方案与组织学亚型和致癌驱动突变相关,并强烈预测临床结果。
- 这种新的基因组分类方法可以用于改善未来临床试验中的患者分层和指导治疗。
通过这篇论文,研究人员提供了一种新的工具来理解和分类IHC的遗传异质性,这对于改进治疗策略和提高患者预后具有重要意义。
重点关注
Figure 4 展示了肝内胆管癌(IHC)的组织学分析,特别是胆管类(biliary-class)和肝细胞癌类(HCC-class)IHC的代表性苏木精-伊红染色图像。
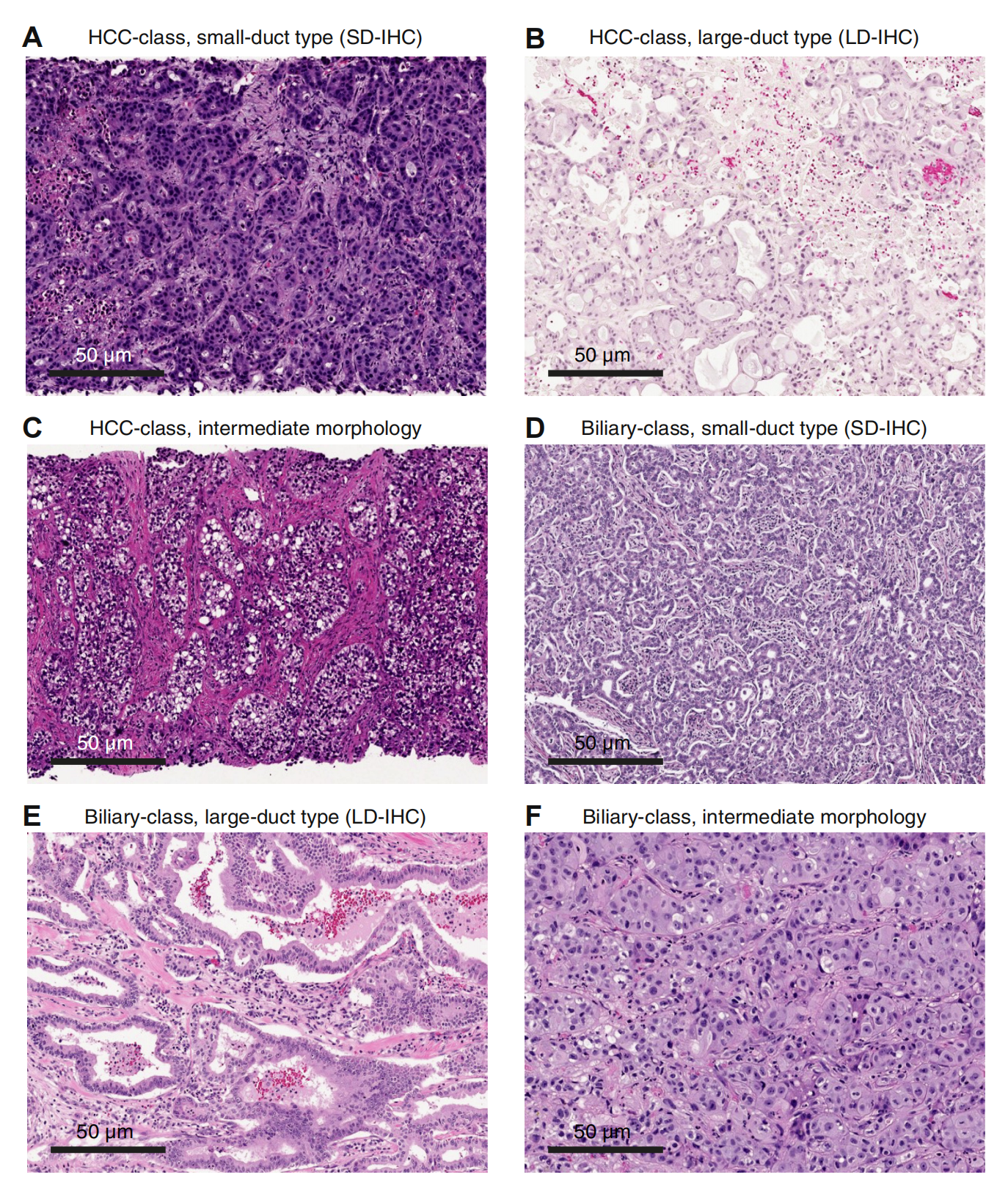
(A) HCC-class, SD-IHC (小胆管型): 这部分图像展示了小胆管型的肝细胞癌类IHC,其特征是存在立方状细胞(cuboidal cells)和相互连接的管状腺体(anastomosing tubular glands)。这种结构提示肿瘤可能起源于小胆管。
(B) HCC-class, LD-IHC (大胆管型): 这部分图像展示了大胆管型的肝细胞癌类IHC,可能偶尔出现小口径腺体轮廓,但更常见的是含有粘液的柱状细胞质(columnar mucin-containing cytoplasm)。这表明肿瘤可能具有更接近于大胆管的特征。
© HCC-class, 形态不明确: 这部分图像中的HCC类IHC缺乏足够的组织结构特征来区分是SD-IHC还是LD-IHC。这意味着在这些样本中,无法明确判断肿瘤是起源于小胆管还是大胆管。
(D) 胆管类, SD-IHC: 这部分图像与(A)类似,展示了胆管类IHC的小胆管型,特征是小口径的管状结构和立方状细胞。
(E) 胆管类, LD-IHC: 这部分图像展示了胆管类的大胆管型IHC,具有经典的大胆管和柱状上皮细胞(columnar epithelium)。这表明肿瘤可能起源于肝内大胆管。
(F) 胆管类, 形态不明确: 这部分图像中的胆管类IHC具有多边形细胞(polygonal cells)和不足以确定亚型的腺体形成不足(insufficient gland formation)。这同样表明在这些样本中,无法明确判断肿瘤的起源。
这些图像的分析表明,IHC在组织学上具有异质性,可以从小胆管型到大胆管型不同,甚至有些样本难以确定具体的亚型。这种异质性可能与肿瘤的起源、发展以及对治疗的响应有关。通过这种组织学分析,研究人员可以更好地理解IHC的不同亚型,并可能为临床治疗提供指导。
二、等离子体增强型SERS传感器在结直肠癌早期诊断中的应用

一作&通讯
| 作者类型 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Kangseok Jo | 韩国材料科学研究所(Korea Institute of Materials Science, KIMS) |
| 第一作者 | Vo Thi Nhat Linh | 韩国材料科学研究所(Korea Institute of Materials Science, KIMS) |
| 通讯作者 | Su Woong Yoo | 全南国立大学生物医学科学研究生项目(Biomedical Science Graduate Program, Chonnam National University) |
| 通讯作者 | Ho Sang Jung | 韩国材料科学研究所(Korea Institute of Materials Science, KIMS) |
文献概述
这篇文章报道了一种基于表面增强拉曼散射(SERS)的无标记结直肠癌(CRC)检测技术,通过光纤内窥镜系统实现直接粘液取样和癌症诊断。
-
研究背景:
- 问题:结直肠癌(CRC)是一种常见且致命的癌症,早期和准确的检测对提高患者生存率至关重要。
- 难点:现有的诊断技术如结肠镜检查和活检具有侵入性,并且存在并发症风险,特别是对于小于1厘米的肿瘤,现有方法的诊断效果有限。
- 相关工作:目前,荧光内窥镜和肿瘤标志物检测等方法在一定程度上改善了CRC的诊断,但这些方法在图像质量、帧率、电池寿命以及自动化异常检测等方面存在局限性。
-
研究方法:
- 开发了一种基于等离子体金纳米多面体(AuNH)涂层的针式表面增强拉曼散射(SERS)传感器,结合内窥镜进行直接粘液采样和无标记CRC检测。
- 使用聚多巴胺(PD)作为粘附层和还原剂,在针表面形成AuNP种子,并通过表面定向还原生长成高密度的AuNHs。
- 通过扫描电子显微镜(SEM)、聚焦离子束-透射电子显微镜(FIB-TEM)、X射线衍射(XRD)和电子能量损失谱(EELS)分析,详细研究了AuNHs的形成机制和PNS传感器的层状结构。
- 使用有限差分时域(FDTD)模拟验证了PNS传感器的强场增强效应,主要集中在AuNHs的多边形边缘和纳米间隙处。
-
实验设计:
- 利用小鼠模型进行体内粘液采样和SERS测量,验证了PNS传感器结合内窥镜系统的可行性。
- 通过正交小鼠结肠肿瘤模型,使用内窥镜针注射法将CT26小鼠结肠癌细胞注入结肠壁,建立肿瘤模型。
- 在内窥镜过程中,PNS传感器被预插入内窥镜管中,确保安全有效的导航。通过白光内窥镜引导,精确可视化并减少组织损伤,PNS传感器在目标区域缓慢而小心地暴露并轻轻擦拭结肠表面,直接收集粘液样本。
-
结果与分析:
- PNS传感器展示了高灵敏度和均匀性,能够检测到低至20 nM的孔雀绿(MG),计算得出的检测限为11.7 nM。
- 通过逻辑回归(LR)机器学习方法,成功区分了CRC和正常小鼠的粘液样本,实现了100%的敏感性、93.33%的特异性和96.67%的准确性。
- 进行了代谢物分析和相关性分析,发现了潜在的CRC生物标志物,如α-羟基苯乙酸(α-HPA)、亮氨酸、牛磺酸、丙氨酸、胸腺嘧啶、棕榈酸和N1,N12-二乙酰精胺(DAS)。
-
总体结论:
- 该系统有望成为非侵入性CRC筛查和早期诊断的有力工具,提升患者护理水平。
- 尽管当前系统分为两步进行(体内粘液样本采集和体外拉曼光谱分析),但对于高风险患者,该系统可用于初步检测和评估,后续再进行彻底评估和治疗决策。
- 未来可以将PNS传感器与光纤拉曼光极结合,实现实时监测和原位CRC检测。
这篇论文展示了PNS传感器结合内窥镜和机器学习技术在CRC无标记诊断中的巨大潜力。
重点关注
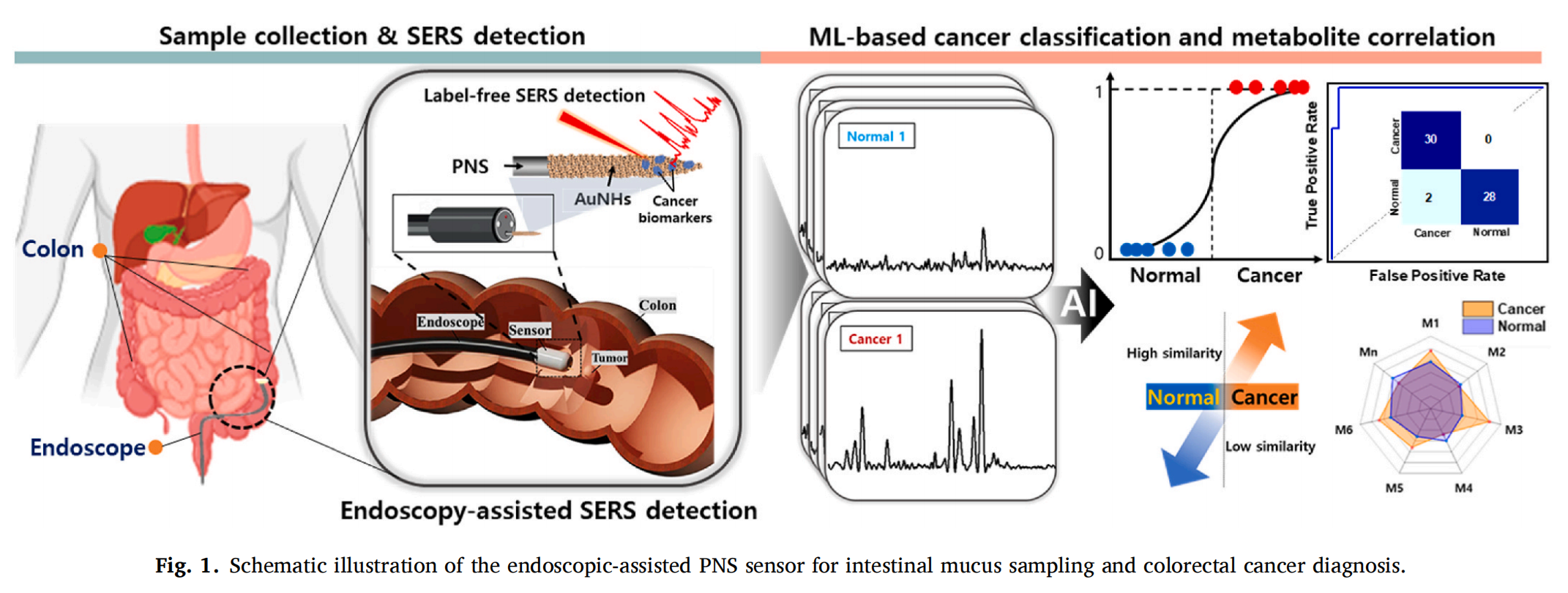
Fig. 1 提供了一种内窥镜辅助的PNS(Plasmonic Needle-SERS)传感器用于肠道粘液取样和结直肠癌诊断的示意图。
-
内窥镜集成: 图中展示了PNS传感器如何集成到内窥镜系统中。传感器是一根细长的针,可以插入内窥镜的管道中,以便在内窥镜检查过程中直接接触并取样肠道粘膜。
-
粘液取样: PNS传感器的表面被设计为可以与肠道粘液接触并吸附粘液,这些粘液可能包含与结直肠癌相关的生物标志物。
-
SERS检测机制: 在取样后,PNS传感器将利用表面增强拉曼散射技术来检测粘液中的分子。SERS是一种能够显著增强拉曼信号的技术,使得检测更为灵敏。
-
金纳米多面体(AuNH): PNS传感器的表面覆盖有AuNHs,这些特殊的纳米结构能够提供强烈的局部电磁场增强效应,这是SERS效应的关键。
-
癌症诊断: 通过分析SERS信号,可以区分正常和癌症状态下的粘液样本,实现结直肠癌的无标记诊断。
-
非侵入性: 整个取样和检测过程是微创的,减少了对患者的侵入性,提高了患者接受度。
-
实时潜力: 尽管文中没有明确指出,但这种集成的系统有潜力实现实时或近实时的癌症检测,因为它允许在内窥镜检查过程中直接取样和分析。
-
机器学习应用: 根据文中描述,这种技术还可能结合机器学习算法来分析SERS数据,进一步提高诊断的准确性。
Fig. 1 通过直观的方式展示了PNS传感器的工作原理和其在结直肠癌诊断中的应用潜力。
三、APOLLO平台:基于拉曼光谱和机器学习的胶质瘤亚型分类器

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Adrian Lita | 美国国立卫生研究院国家癌症研究所神经肿瘤科 |
| 通讯作者1 | Mioara Larion | 美国国立卫生研究院国家癌症研究所神经肿瘤科 |
| 通讯作者2 | Ion Petre | 芬兰图尔库大学数学与统计系 罗马尼亚布加勒斯特大学数学与计算机科学学院 罗马尼亚生物科学国家研究与发展部生物信息学系 |
文献概述
这篇文章报道了一个基于拉曼光谱和机器学习技术的新型平台APOLLO,能够从常规的石蜡包埋组织切片中准确区分不同亚型的胶质瘤,并揭示了它们独特的代谢特征。
-
研究背景:
- 问题:这篇文章旨在解决FFPE组织切片在拉曼光谱分析中的局限性,特别是由于嵌入介质引起的背景干扰。
- 难点:该问题的研究难点在于如何在不影响样本的情况下,从FFPE组织中提取有意义的生物化学信息,并准确区分不同类型的胶质瘤。
- 相关工作:现有工作主要集中在使用新鲜或冷冻组织进行拉曼光谱分析,而不是FFPE组织,因为FFPE组织的高背景干扰了光谱分析的准确性。
-
研究方法:
- 使用自发拉曼光谱对46例已知甲基化亚型的FFPE组织样本进行分子指纹分析。具体来说,使用ThermoFisher DXR2xi Raman显微镜采集光谱数据,激光波长为532 nm,功率为10 mW,曝光时间为0.25秒,扫描5次。
- 数据预处理包括去除沉默区域、基线校正和归一化。使用airPLS算法进行基线校正,并将每个光谱的强度除以其L2范数以实现归一化。
- 使用DBSCAN算法和Mini-batch K-means算法进行聚类分析,自动分离肿瘤和非肿瘤组织。
- 训练随机森林和支持向量机分类器,用于区分IDH1WT与IDH1mut肿瘤,以及G-CIMP-high与G-CIMP-low亚型。采用5折交叉验证设计,确保模型的泛化能力。
-
实验设计:
- 数据集来自46例患者的FFPE样本,这些样本的肿瘤已被分类为不同的甲基化亚型。每个样本有平行切片用于H&E染色确认肿瘤细胞的存在。
- 在每个样本的59个选定区域内记录了2116到14945个光谱,每个区域约300μm²,平均每个样本记录了300,506个拉曼光谱。
- 使用DBSCAN算法对光谱进行聚类,识别肿瘤和非肿瘤区域。通过PCA和t-SNE分析评估聚类结果的质量。
-
结果与分析:
- APOLLO平台能够准确区分肿瘤和非肿瘤组织,IDH1mut与IDH1WT肿瘤,以及IDH1mut亚型中的G-CIMP-high与G-CIMP-low。具体来说,肿瘤与非肿瘤区域的分类ROC曲线下面积(AUC)为0.99。
- 通过随机森林和ANOVA方法识别出2883 cm⁻¹、1690 cm⁻¹、1607 cm⁻¹、1573 cm⁻¹、1401 cm⁻¹和1335 cm⁻¹等频率在区分肿瘤和非肿瘤组织中具有重要作用。
- 在IDH1mut与IDH1WT的区分中,胆固醇酯的水平在IDH1mut胶质瘤中显著高,2883 cm⁻¹频率的强度显著高于IDH1WT。
- 在G-CIMP-high与G-CIMP-low亚型的区分中,2887 cm⁻¹和2865 cm⁻¹等频率表现出显著的差异,模型在G-CIMP-low的分类中表现更好,平均精度为0.89,召回率为0.91。
-
总体结论:
- 这篇论文展示了无标记拉曼光谱在分类胶质瘤亚型和提取有意义生物信息方面的潜力,特别是在FFPE组织样本中的应用。APOLLO平台提供了一种新的代谢和生化研究途径,可以扩展到其他类型的FFPE组织或分类中。
重点关注
图1提供了APOLLO平台研究设计的概览,可以分为三个主要部分:
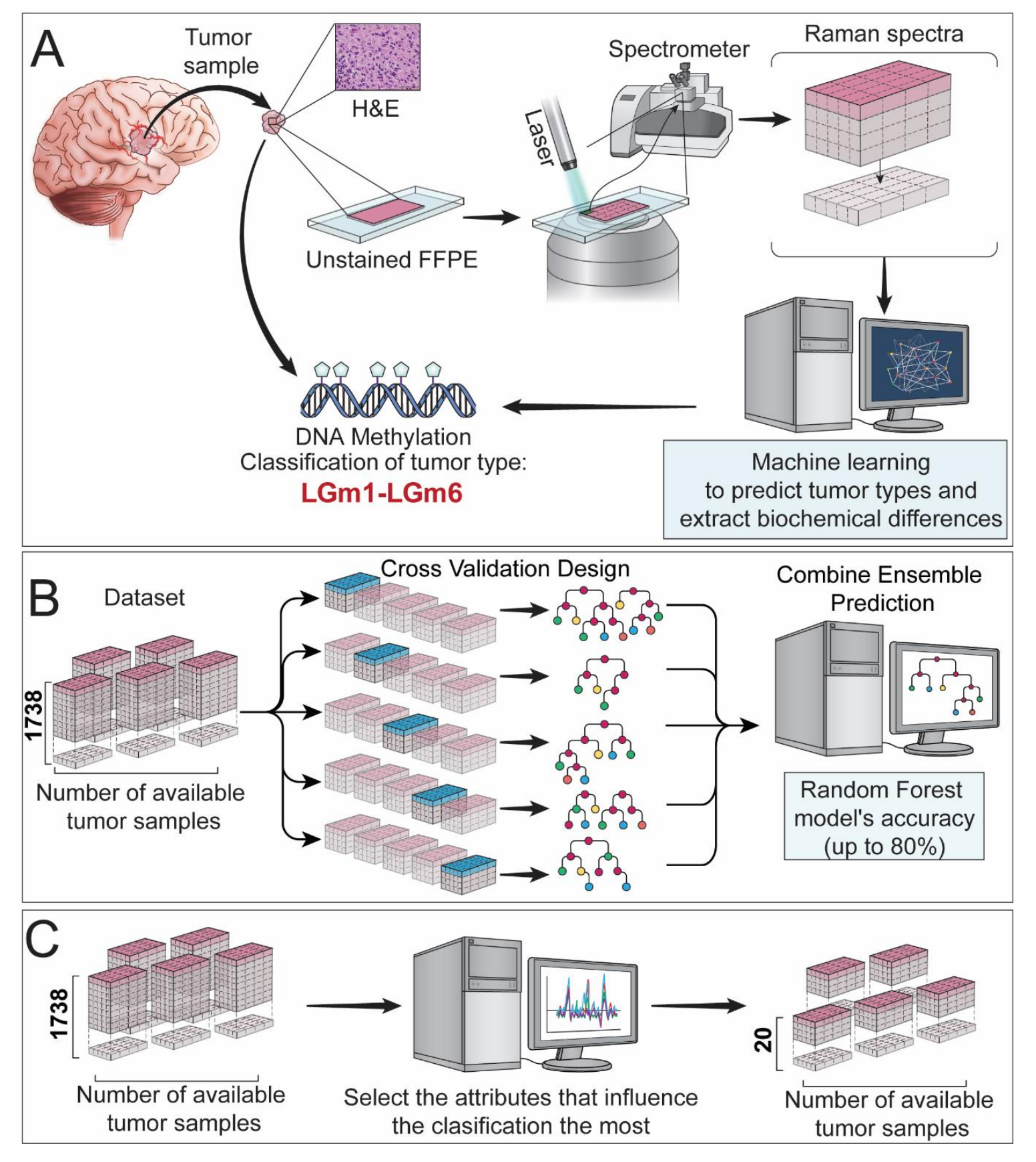
A. 研究设计:
- 对FFPE(甲醛固定,石蜡包埋)组织切片进行H&E(苏木精-伊红)染色,以确认感兴趣区域中的肿瘤。
- 确认肿瘤的甲基化亚型。
- 使用自发拉曼光谱分析样品。
B. 机器学习训练设计:
- 数据集由每个肿瘤斑点的拉曼光谱及其甲基化标签(IDH1突变型或野生型,LGm1或LGm2)组成。
- 由于数据通常不平衡,即一个类别的样本数量远多于另一个类别,研究者将数据分割成五个不相交的数据集,为机器学习模型的5折交叉验证训练做准备。
- 为了避免数据泄露,采用了肿瘤分层方法:一个样本的所有斑点都贡献给一个子集。
- 每个子集的数据分布大致遵循整个数据集的分布。
- 运行5折交叉验证,训练5个独立的随机森林模型,每个子集轮流作为验证集,其余四个作为训练集。
- 五个独立的随机森林模型的预测结果被合并为最终的随机森林模型。
C. 模型增强:
- 通过在20个最重要的拉曼频率上训练支持向量分类器,进一步增强模型的性能。
简而言之,图1展示了APOLLO平台如何结合拉曼光谱和机器学习技术来区分不同类型的胶质瘤,并通过交叉验证和特征重要性评估来优化其分类模型。
四、深度学习在黑色素细胞皮肤肿瘤全切片图像中的ROI检测与分类研究

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Yi Cui | 北卡罗来纳大学教堂山分校经济系 |
| 通讯作者 | Nancy E. Thomas | 北卡罗来纳大学教堂山分校医学院皮肤病学系、综合癌症中心 |
文献概述
这篇文章利用深度学习技术在黑色素细胞皮肤肿瘤的全切片图像中实现了高准确度的感兴趣区域检测和肿瘤类型分类。
-
研究背景:
- 问题:准确和及时的癌症诊断对于患者的治疗和预后至关重要。然而,传统的组织病理学诊断方法依赖于病理学家的视觉评估,存在诊断差异和高成本的问题。
- 难点:黑色素瘤和良性痣(nevus)的区分在临床上具有重要意义,但传统的诊断方法存在时间消耗和准确性不足的问题。此外,病理学家之间的诊断一致性较低,导致误诊和漏诊的风险增加。
- 相关工作:近年来,深度学习技术在医学图像分析中显示出巨大的潜力,已被应用于肿瘤分类、癌症分析和预测等领域。然而,现有的方法在黑色素瘤区域兴趣点(ROI)检测方面的准确性仍然有限。
-
研究方法:
- 使用UNC黑色素瘤数据集,包含86张黑色素瘤和74张良性痣的全视野图像(WSI)。数据集被随机分为80%的训练集(134张WSI)和20%的测试集(26张WSI)。
- 预处理步骤包括颜色归一化和数据增强,以确保图像在不同实验条件下的颜色一致性,并提高模型的鲁棒性。
- 提取图像补丁并进行标注,使用VGG16作为基础架构训练一个三类补丁分类模型(PCLA-3C),用于分类黑色素瘤、良性痣和其他类别。
- 通过多数投票法对整个WSI进行分类,并计算ROI检测的交并比(IoU)。使用OPTICS算法对高预测分数的补丁进行聚类,生成边界图和热图。
-
实验设计:
- 实验在UNC Longleaf Cluster上进行,使用NVIDIA GPU进行计算加速。实验包括对160张WSI进行随机分割,分别用于训练和测试。
- 通过不同的训练集比例(如80%、60%、40%和20%)训练模型,并在测试集上评估其性能,以验证模型的鲁棒性。
- 具体实验结果显示,使用80%的原始训练集时,模型在补丁分类准确率、切片分类准确率和IoU分别为0.885、0.885和0.371。
-
结果与分析:
- 在测试集上,PCLA-3C方法的切片分类准确率为92.3%,ROI检测的IoU为38.2%,显著优于CLAM方法的69.2%和11.2%。
- 混淆矩阵显示,PCLA-3C方法在识别良性痣和黑色素瘤方面具有高准确性和特异性,准确率为93.5%,敏感性为81.8%,特异性为100%。
- 只有两张良性痣的WSI被错误分类为黑色素瘤,主要原因是这些WSI中存在颜色较深的区域,导致模型误判。
-
总体结论:
- 该研究展示了一种高效的深度学习框架,能够自动检测皮肤肿瘤并预测肿瘤类型,具有较高的准确性和鲁棒性。
- 该方法不仅有助于区分黑色素瘤和良性痣,还可以扩展到其他类型的癌症诊断中,具有广泛的应用前景。
- 未来工作将包括结合基因表达和临床数据进一步提高检测和预测的准确性。
通过这篇论文,作者展示了深度学习技术在黑色素瘤和良性痣诊断中的巨大潜力,提出了一种高效且准确的自动检测方法,有望改进临床诊断流程。
重点关注
Figure 2 提供了所提出检测框架的概览,分析如下:
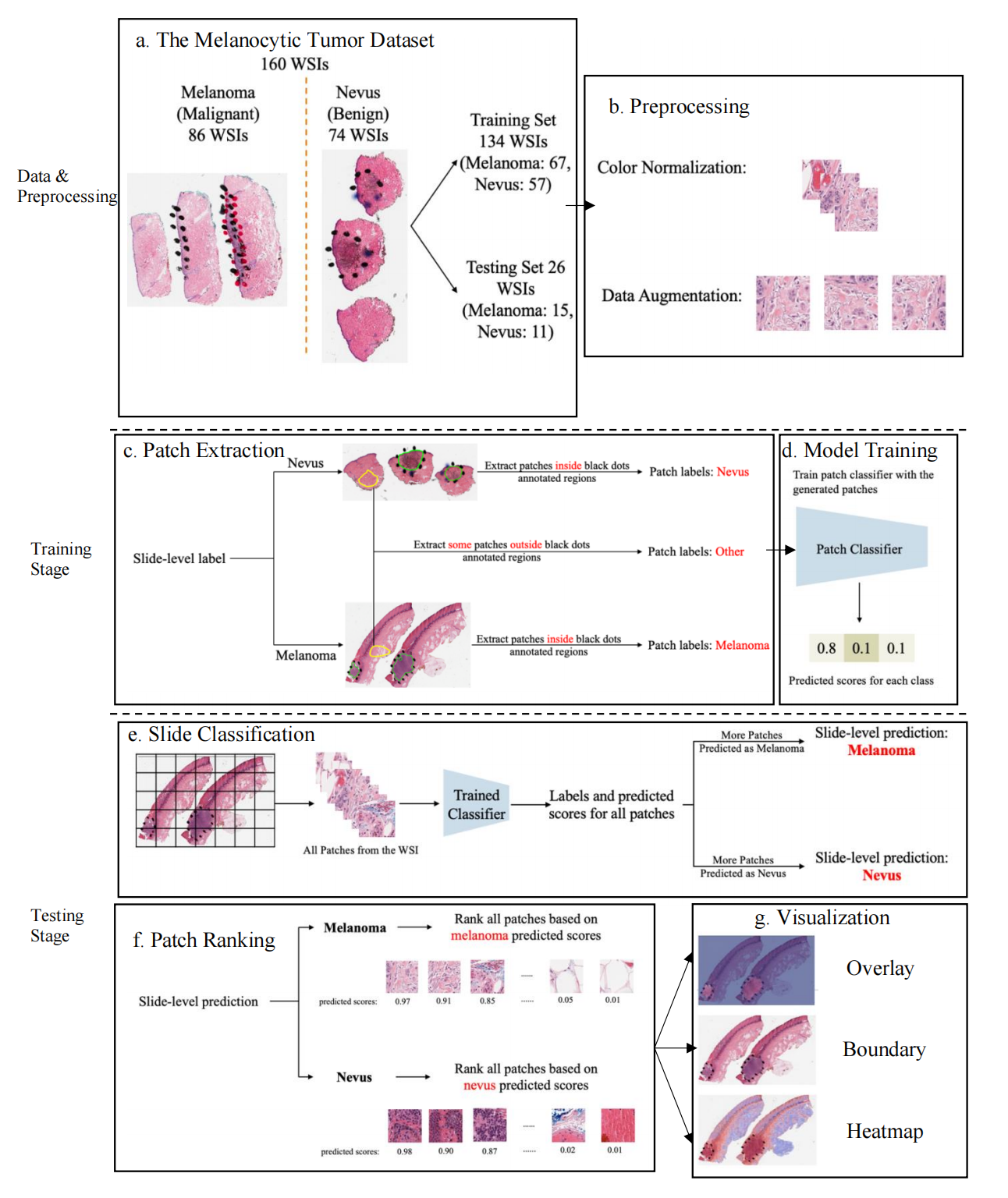
(a) 数据集划分:黑色素细胞肿瘤数据集共包含160个全切片图像(WSIs),其中80%(134个图像)被随机分配作为训练集,剩余20%(26个图像)作为测试集。
(b) 预处理:包括颜色标准化和数据增强。颜色标准化是为了减少不同实验室或不同时间扫描导致的色差对模型性能的影响。数据增强可能包括旋转、翻转、缩放等操作,以增加数据多样性并提高模型的泛化能力。
© 块提取:从训练数据中提取黑色素瘤、痣以及其他类型的组织块(patches)。这些块根据它们在图像中的标注被分类标签。
(d) 模型训练:基于提取的块,训练了一个三类(黑色素瘤、痣和其他)的块分类器。这里使用了VGG16作为基础架构进行模型训练。
(e) 切片分类:对于测试集中的每个切片,模型为所有块生成预测分数,并计算块和切片分类的准确性。
(f) 块排序:根据切片的分类结果,所有块根据相应的预测分数进行排序。如果是黑色素瘤,块将根据黑色素瘤预测分数排序;如果是痣,则按痣预测分数排序。
(g) 结果可视化:基于预测分数,生成可视化结果,可能包括不同类别块的分布图、预测感兴趣区域的边界图等,以直观展示模型的预测结果。
总的来说,Figure 2 描述了一个从数据准备到模型训练、分类、排序,最终结果可视化的完整流程,旨在自动化地检测和分类黑色素细胞皮肤肿瘤的全切片图像。
五、立体定向脑活检中AI驱动的脑肿瘤分类与分子亚型分析

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | David Reinecke | 德国科隆大学医学院及大学医院神经外科中心 |
| 通讯作者 | Volker Neuschmelting | 德国科隆大学医学院及大学医院神经外科中心、德国科隆大学医学院及大学医院肿瘤中心、德国科隆大学医学院及大学医院综合肿瘤中心 |
文献概述
这篇研究展示了使用人工智能和受激拉曼散射显微镜技术在立体定向脑活检中快速准确诊断脑肿瘤及其分子亚型的能力。
-
研究背景:
- 问题:这篇文章旨在解决立体定向脑活检中快速、准确地进行脑肿瘤分类和分子亚型分型的问题。
- 难点:该问题的研究难点在于传统冷冻切片分析需要较长时间,且对样本量要求较高,可能导致手术时间延长和样本信息的丢失。
- 相关工作:现有工作主要集中在利用人工智能算法辅助术中决策,但大多数研究基于微外科肿瘤切除样本,缺乏对小样本立体定向脑活检数据的验证。
-
研究方法:
- 研究设计:这是一项单中心的前瞻性研究,纳入了84名接受立体定向脑活检的患者,共收集了121张SRH图像。使用便携式光纤激光拉曼散射显微镜对未处理、无标记的样本进行成像。
- 深度学习模型:测试了三种深度学习模型:
- 第一个模型用于识别肿瘤和非肿瘤组织。
- 第二个模型用于将肿瘤进一步分类为高级别胶质瘤、低级别胶质瘤、转移瘤、淋巴瘤或胶质增生。
- 第三个模型用于成人型弥漫性胶质瘤的IDH和1p/19q状态的分子亚型分类。
- 数据处理:使用QuPath软件对SRH图像进行分割和拼接,计算可用于深度学习模型分析的样本面积。
-
实验设计:
- 样本收集:在手术过程中,收集了多个样本,每个患者至少获得一个样本用于FFPE诊断。新鲜的小样本(0.5-2毫米长,直径1毫米)用于SRH成像。
- 图像采集:使用不同的视野大小(1.7×1.8毫米、2.5×2.8毫米、4.2×4.7毫米)进行成像,平均成像时间为3.5分钟。
- 模型评估:通过逐步评估模型性能,确定了高质量图像补丁的数量阈值,以确保模型的高准确性。
-
结果与分析:
- 第一个模型识别肿瘤和非肿瘤组织的准确率为91.7%。
- 第二个模型在肿瘤亚分类中的准确率为73.9%,当SRH图像包含超过140个高质量补丁时,准确率提高到89.5%。
- 第三个模型在成人型弥漫性胶质瘤的分子亚型分类中的准确率为87%,当SRH图像包含超过140个高质量补丁时,准确率提高到93.9%。
- 与传统冷冻切片分析相比,SRH图像分析在样本量显著减少的情况下,诊断准确性相当甚至更高。冷冻切片分析的平均样本面积为16.7±8.2 mm²,而SRH图像的平均样本面积仅为4.1±2.5 mm²。
-
总体结论:
- AI辅助的SRH图像分析在小样本立体定向脑活检中是可行且有效的,能够在几分钟内实现脑肿瘤的诊断和分子亚型预测。
- 这种方法显著减少了所需的活检组织量,并允许在术中快速做出治疗决策。
- 未来的研究需要进一步优化模型性能,以更好地处理罕见诊断,并确定其在神经肿瘤学护理中的潜在常规作用。
这篇论文展示了AI和SRH技术在提高脑肿瘤术中诊断效率和准确性方面的巨大潜力。
重点关注
Figure 1展示了与传统的H&E快速冰冻切片组织病理学相比,基于AI的SRH图像分析流程的半自动化。

A部分:
- 描述了立体定向引导活检的轨迹规划过程,然后是如何准备新鲜小样本(0.5-2毫米)并挤压在亚克力/玻片上,接着使用基于光纤激光的SRH成像仪进行成像,生成类似H&E染色的虚拟图像(视野范围FOV为4.2 × 4.7毫米,组织区域为9.5平方毫米)。
B部分:
- 同时,一块较大的新鲜样本(5-10毫米)被运送到神经病理学单位进行H&E组织学分析(视野范围FOV为6 × 3.5毫米,组织区域为33.6平方毫米)。
- SRH图像被划分为300×300像素的小块,作为所有三个深度学习模型的特征提取输入。
- 第一个模型将图像块分类为三个类别:肿瘤(红色)、非肿瘤(绿色)和低质量(紫色)。
- 第二个模型使用推理算法,当预测为肿瘤(红色)时,进一步将图像块分为不同的诊断类别,如高级别胶质瘤、弥漫性低级别胶质瘤、淋巴瘤、转移瘤、胶质增生等。
- 非肿瘤组织(如白质、灰质和胶质增生)以绿色显示,低质量的图像块(如碎片)以紫色显示。
- 第三个模型结合了预训练的分子遗传数据和图像输入,通过两个嵌入空间集成到变换器编码器中,以实现成人型弥漫性胶质瘤的分子分类,包括IDH突变伴随1p/19q缺失(红色)、IDH野生型(绿色)和IDH突变不伴随1p/19q缺失(紫/蓝色)。
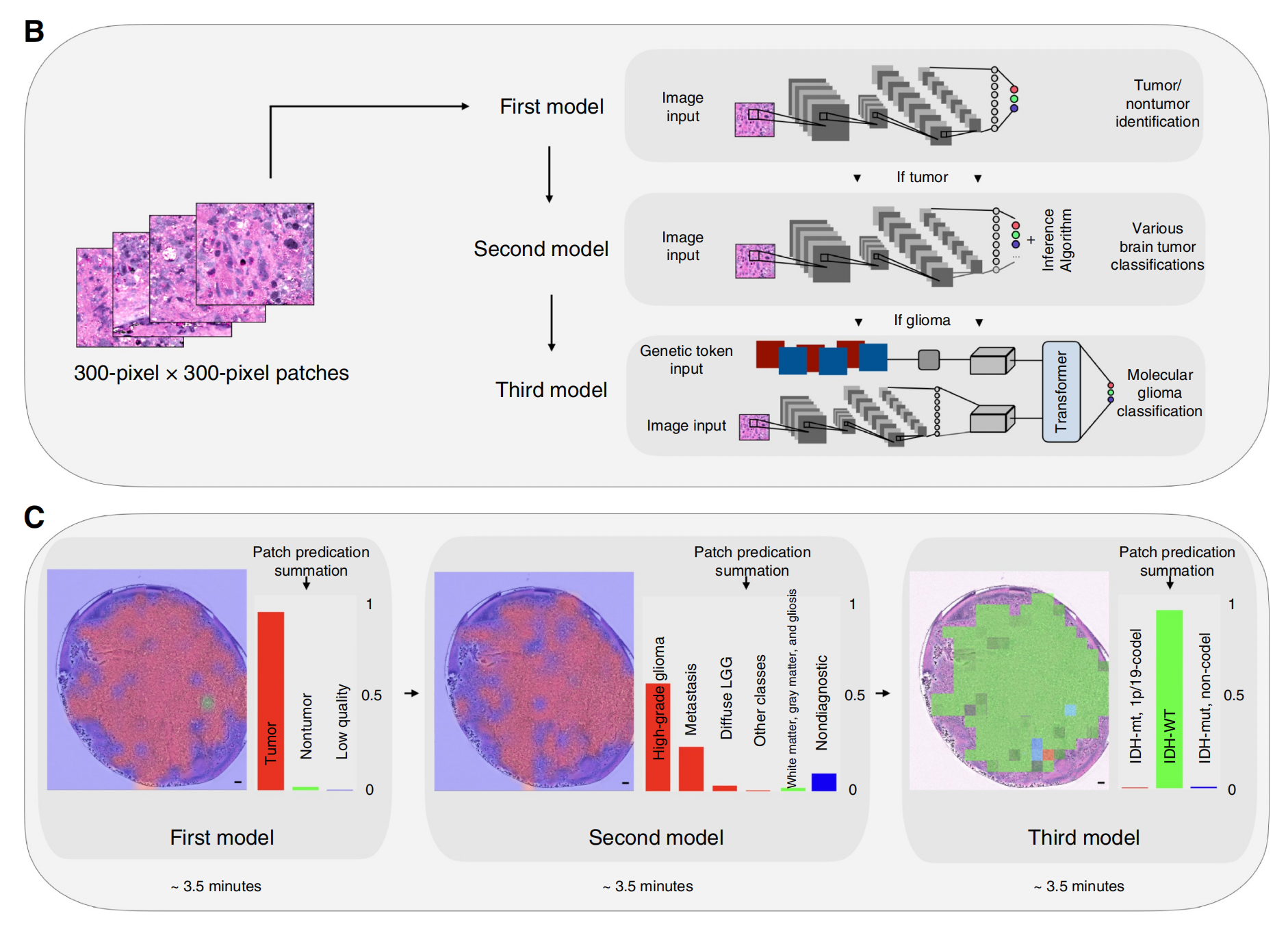
C部分:
- 将SRH图像的各个小块相加并重新标准化,以获得对诊断类别的整体预测分布。
- 此外,使用前馈窗口滑动技术对图像块进行语义分割,创建一个概率热图,作为实际SRH图像上的RGB叠加层,并为外科医生或神经病理学家提供基于颜色的可视化评估。
- 第一个模型将大部分SRH图像区域识别为肿瘤,第二个模型正确将其分类为高级别胶质瘤(CNS WHO 4级),第三个模型将其分类为IDH野生型胶质瘤。
- 图中还包含了比例尺,200微米。
整体而言,这个流程图说明了如何利用AI技术分析SRH图像,以辅助在脑肿瘤手术中进行快速和准确的诊断。
六、无监督互学习变换器:一种提升全切片图像分类性能的新算法

一作&通讯
| 角色 | 姓名 | 单位 | 单位(中文) |
|---|---|---|---|
| 第一作者 | Sajid Javed | Department of Computer Science, Khalifa University of Science and Technology, Abu Dhabi, P.O. Box 127788, United Arab Emirates | 计算机科学系,哈利法科学技术大学,阿布扎比,邮政信箱127788,阿拉伯联合酋长国 |
| 通讯作者 | Naoufel Werghi | Department of Computer Science, Khalifa University of Science and Technology, Abu Dhabi, P.O. Box 127788, United Arab Emirates | 计算机科学系,哈利法科学技术大学,阿布扎比,邮政信箱127788,阿拉伯联合酋长国 |
文献概述
这篇文章提出了一种基于互学习变换器的无监督算法,用于提高多吉咖级全切片图像分类的准确性,特别是在计算病理学中的癌症检测和细胞突变预测方面。
-
研究背景:
- 问题:癌症是全球主要的死亡原因之一,每年约有2000万新癌症病例报告,给医疗系统带来了巨大的负担。组织切片图像的视觉检查被认为是癌症诊断的金标准,但现代数字切片扫描仪可以生成高分辨率的多吉像素WSIs,直接应用机器学习方法面临挑战。
- 难点:WSIs的巨大尺寸使得标注工作既费时又昂贵,特别是区域级别的标注需要专家病理学家的参与。现有的弱监督方法虽然减少了标注成本,但仍需大规模标注数据集,且可能因标注不准确而导致模型性能下降。
- 相关工作:现有工作主要集中在有监督学习和弱监督学习上,有监督方法需要大量标注数据,而弱监督方法虽减少标注成本,但仍需专家检查。自监督学习方法通过数据自身生成标签,但在无监督WSIs分类中尚未得到广泛应用。
-
研究方法:
- 提出了一个基于互变压变的无监督WSIs分类算法,称为UMTL。该算法包括两个主要模块:变换器伪标签生成器和标签清洁网络。
- 变换器伪标签生成器通过将输入特征投影到潜在空间并逆投影回原始空间来生成伪标签。使用变换损失最小化原始特征和逆投影特征之间的差异。
- 标签清洁网络使用变换器模型清理噪声伪标签。清洁后的标签用于改进变换器伪标签生成器的性能。
- 引入了一个判别学习机制,通过在高斯噪声下测量变换损失来提高伪标签生成器的区分能力。
- 使用图平滑机制作为后处理步骤,抑制孤立的空间稀疏正标签。
-
实验设计:
- 在四个公开可用的WSIs数据集上评估所提出的算法,包括CAMELYON-16(乳腺癌)、TCGA肺癌、TCGA肾细胞癌和TCGA乳腺癌。
- 实验设置包括完全无监督、有限弱监督和下游分析任务训练。
- 在CAMELYON-16数据集上进行无监督病变分割和WSIs分类实验,使用0%标签或注释。
- 在TCGA-LC、TCGA-RCC和TCGA-BRCA数据集上进行完全无监督的WSIs分类实验,使用0%到100%的滑动级别标签。
- 在TCGA-LC数据集上进行弱监督的癌症亚型分类实验,使用LUAD vs. LUSC分类任务。
- 在TCGA-RCC数据集上进行弱监督的癌症亚型分类实验,使用KICH vs. KIRP vs. KIRC分类任务。
- 在TCGA-BRCA数据集上进行弱监督的HER2状态预测实验。
-
结果与分析:
- 在完全无监督设置下,UMTL在CAMELYON-16数据集上的AUC为0.844,在TCGA-LC数据集上的AUC为0.856,在TCGA-RCC数据集上的AUC为0.822。
- 在有限弱监督设置下,UMTL在CAMELYON-16数据集上的AUC为0.966,在TCGA-LC数据集上的AUC为0.966,在TCGA-RCC数据集上的AUC为0.972。
- 在下游分析任务中,D-UMTL在TCGA-LC数据集上的AUC为0.976,在TCGA-RCC数据集上的准确率为0.972,在TCGA-BRCA数据集上的AUC为0.791。
- 与现有的SOTA方法相比,UMTL在各种数据集上均表现出更好的性能。
-
总体结论:
- 提出了一种基于互变压变的无监督WSIs分类算法,能够在无需标注的情况下进行WSIs分类。
- 该算法在多个数据集上表现出色,并且在下游分析任务中也显示出良好的性能。
- 未来工作可以进一步研究使用该算法进行生存预测等临床任务。
重点关注
Fig. 1 展示了用于全切片图像(WSI)分类任务的不同类型的监督方法之间的比较。
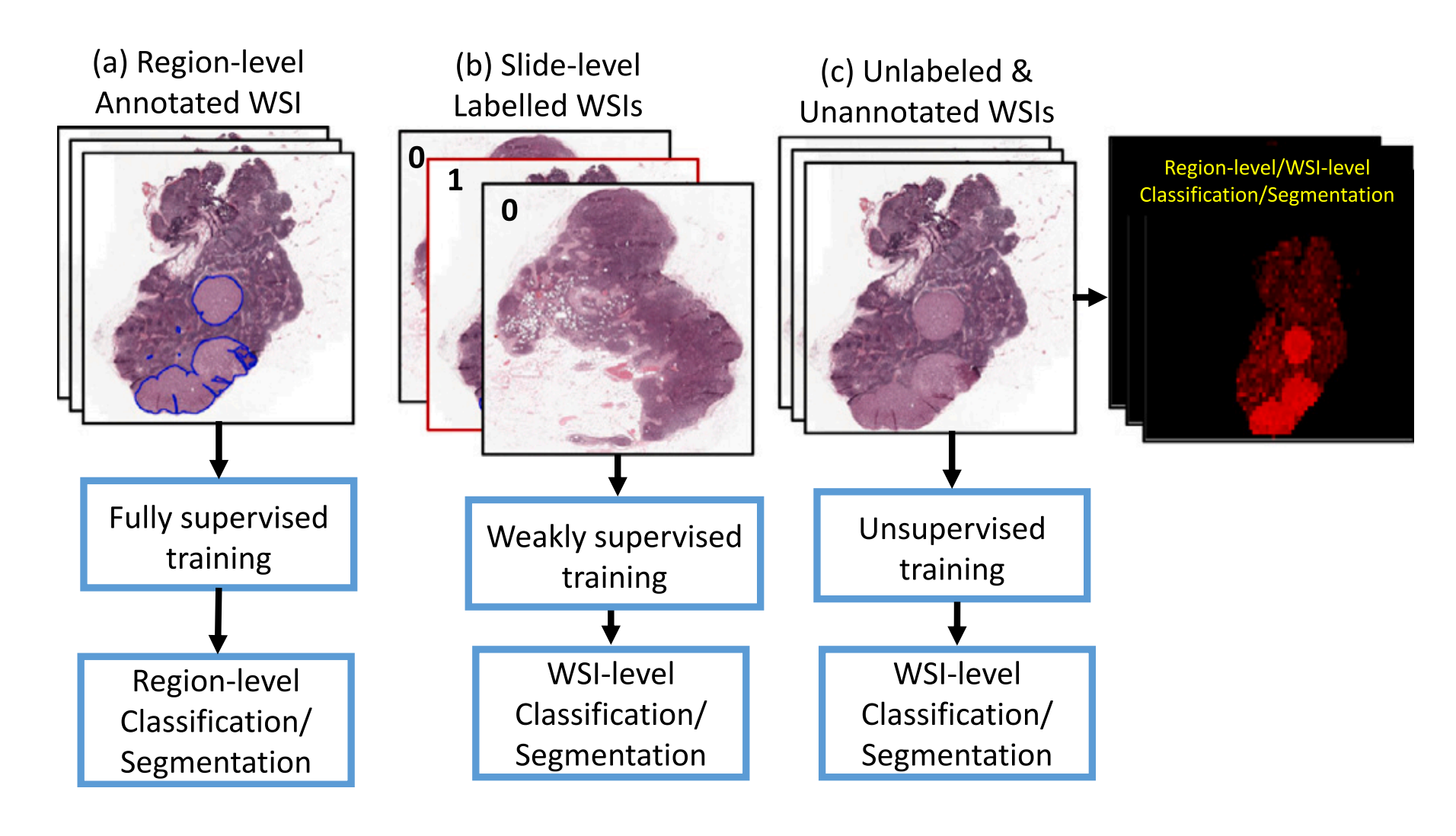
-
(a) 展示了全监督训练的情况,这要求有区域级别的正常或肿瘤的标注。这意味着在训练过程中,需要专家精确地标注出图像中每个区域是正常组织还是肿瘤组织。这种方法成本高且耗时,因为它需要病理学家对图像中的每个区域进行详细的检查和标注。
-
(b) 展示了弱监督训练的情况,这只需要切片级别的标签。与全监督训练相比,弱监督训练的标注要求较低,只需要对整个切片进行是否含有肿瘤的标注,而不需要具体到每个区域。这减少了工作量,但仍需要一定程度的专家介入。
-
© 展示了所提出的无监督训练方法,它既不需要区域级别的标注,也不需要切片级别的标签。这种训练方式完全不需要专家的手动标注,算法自行学习识别图像中的肿瘤区域。图中的红色区域表示使用所提出的无监督算法预测的肿瘤区域,这展示了算法在没有专家指导的情况下进行自动分类的能力。
无监督训练方法的优势在于能够显著减少病理学家的工作量,并可能降低成本和提高效率,同时仍然保持较高的分类准确性。这对于大规模的图像分类任务尤其有价值,因为它允许算法在缺乏详尽标注数据的情况下进行训练和预测。