✨作者主页 :IT研究室✨
个人简介:曾从事计算机专业培训教学,擅长Java、Python、微信小程序、Golang、安卓Android等项目实战。接项目定制开发、代码讲解、答辩教学、文档编写、降重等。
文章目录
一、前言
系统介绍
基于大数据的分化型甲状腺癌复发数据可视化分析系统是一个专门针对甲状腺癌患者临床数据进行深度分析的智能化平台。该系统采用Hadoop+Spark大数据架构,结合Django后端框架和Vue前端技术,构建了完整的数据处理与可视化分析流程。系统以383例分化型甲状腺癌患者的15年随访数据为基础,涵盖13个关键临床病理特征,包括患者基本信息、病理分期、治疗反应、甲状腺功能等多维度指标。通过Spark SQL进行大规模数据处理,运用Pandas和NumPy进行统计分析,采用ECharts实现交互式数据可视化,系统能够从患者人口统计学特征、临床病理核心特征、治疗效果指标、甲状腺功能状态等多个维度进行关联分析。平台提供了热力图关联性分析、风险分层可视化、复发预测模型等核心功能,支持医生快速识别影响甲状腺癌复发的关键因素,为临床决策提供数据支撑,同时为医学研究人员提供了便捷的数据探索工具。
选题背景
分化型甲状腺癌作为内分泌系统最常见的恶性肿瘤,其发病率在全球范围内呈现持续上升趋势,已成为严重威胁人类健康的重要疾病。尽管分化型甲状腺癌整体预后相对较好,但术后复发问题依然是临床关注的焦点,复发率可达10-30%,给患者带来沉重的身心负担和经济压力。传统的医疗数据分析方法在处理大规模、多维度的临床数据时存在明显局限性,医生往往只能基于有限的统计指标进行经验性判断,难以全面把握影响复发的复杂因素关系。随着医疗信息化的深入发展和临床数据的快速积累,如何运用先进的大数据技术挖掘隐藏在海量医疗数据中的有价值信息,识别影响甲状腺癌复发的关键因素模式,已经成为现代精准医学发展的迫切需求。
选题意义
本研究具有重要的理论价值和实践意义,能够为甲状腺癌的临床诊疗和医学研究提供有力支撑。从理论角度来看,该系统通过构建多维度数据分析模型,深入挖掘患者基本特征、病理参数、治疗反应等因素与复发风险之间的关联规律,丰富了甲状腺癌复发机制的理论认知,为建立更加科学的风险评估体系提供了数据基础。从实践应用层面来说,该系统能够辅助临床医生快速识别高风险患者群体,制定个性化的随访监测方案,提高早期发现复发的能力,降低治疗成本,改善患者预后质量。该平台还为医学研究人员提供了便捷的数据探索工具,支持大规模临床数据的统计分析和可视化展示,有助于开展循证医学研究,推动甲状腺癌诊疗规范的不断完善。虽然作为毕业设计项目,系统规模和复杂度相对有限,但其设计理念和技术方案为医疗大数据分析系统的开发提供了可行的参考模式。
二、开发环境
- 大数据框架:Hadoop+Spark(本次没用Hive,支持定制)
- 开发语言:Python+Java(两个版本都支持)
- 后端框架:Django+Spring Boot(Spring+SpringMVC+Mybatis)(两个版本都支持)
- 前端:Vue+ElementUI+Echarts+HTML+CSS+JavaScript+jQuery
- 详细技术点:Hadoop、HDFS、Spark、Spark SQL、Pandas、NumPy
- 数据库:MySQL
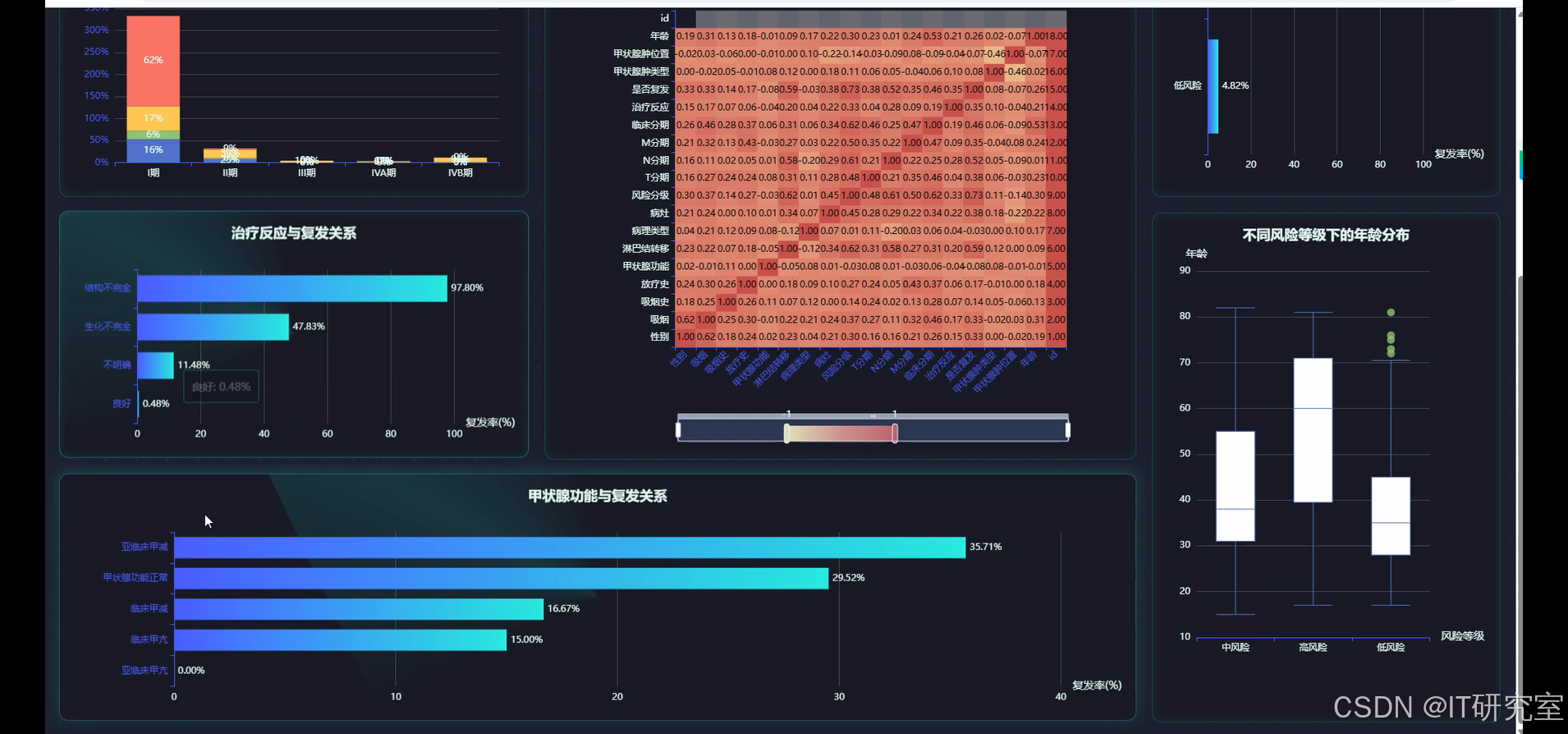
三、系统界面展示
- 基于大数据的分化型甲状腺癌复发数据可视化分析系统界面展示:
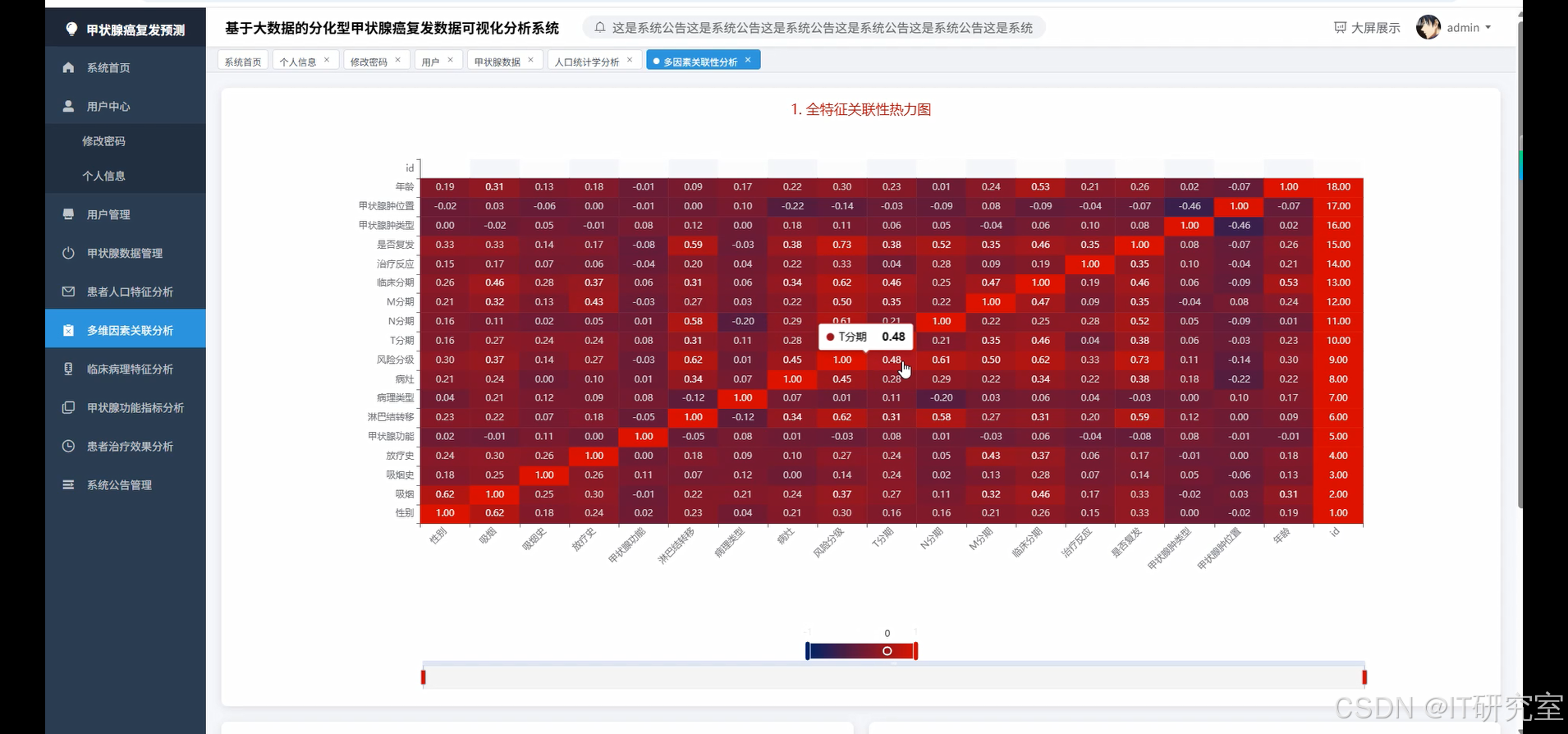
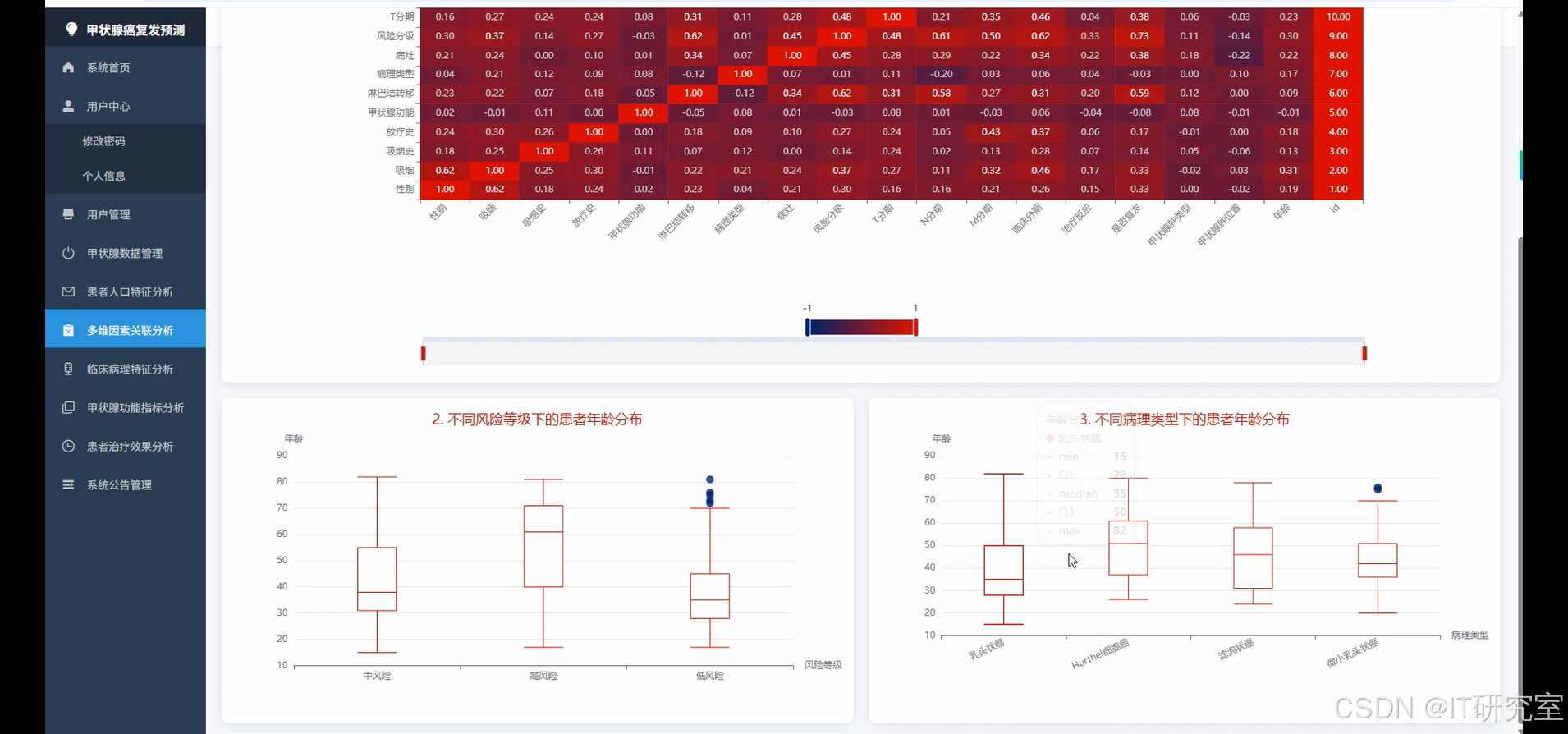
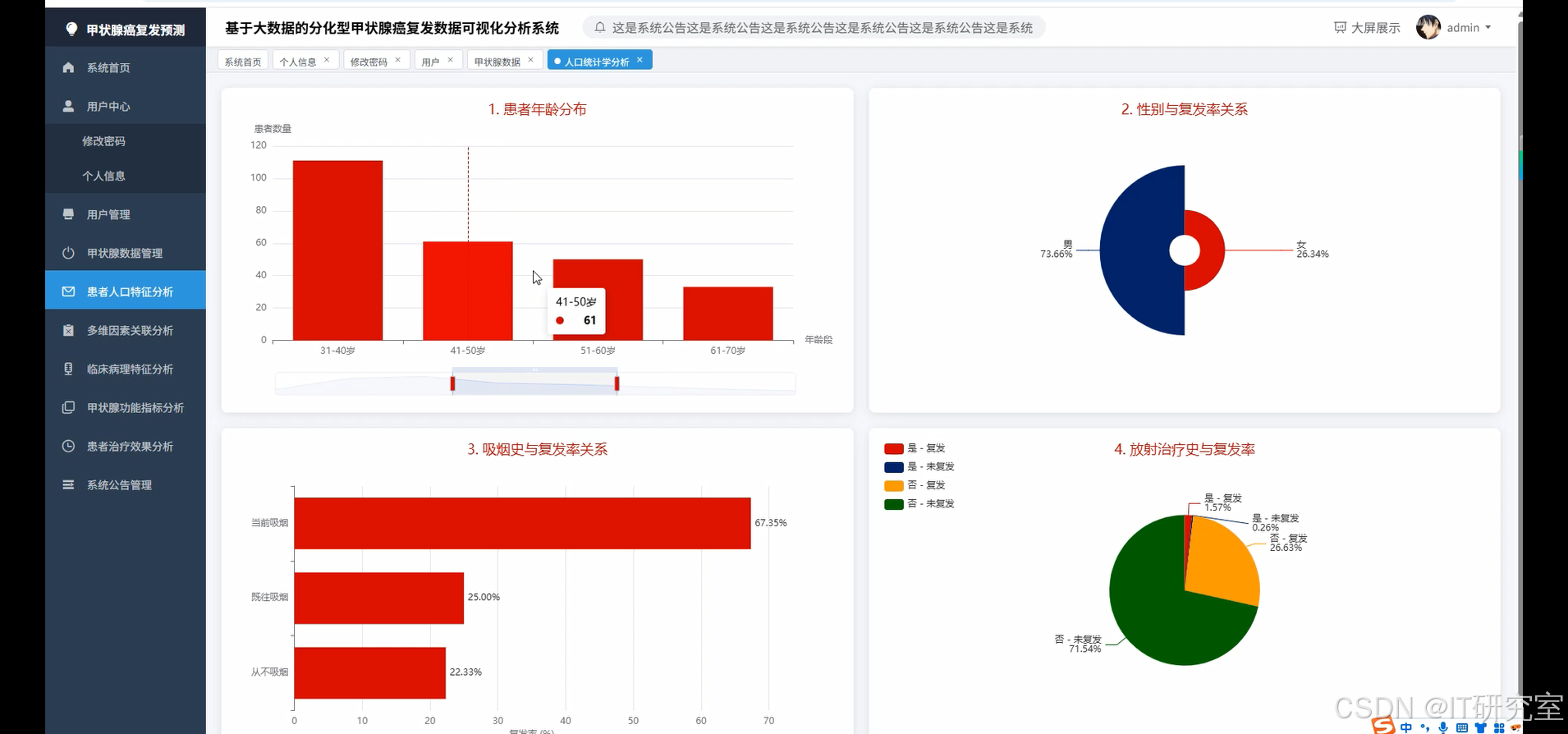
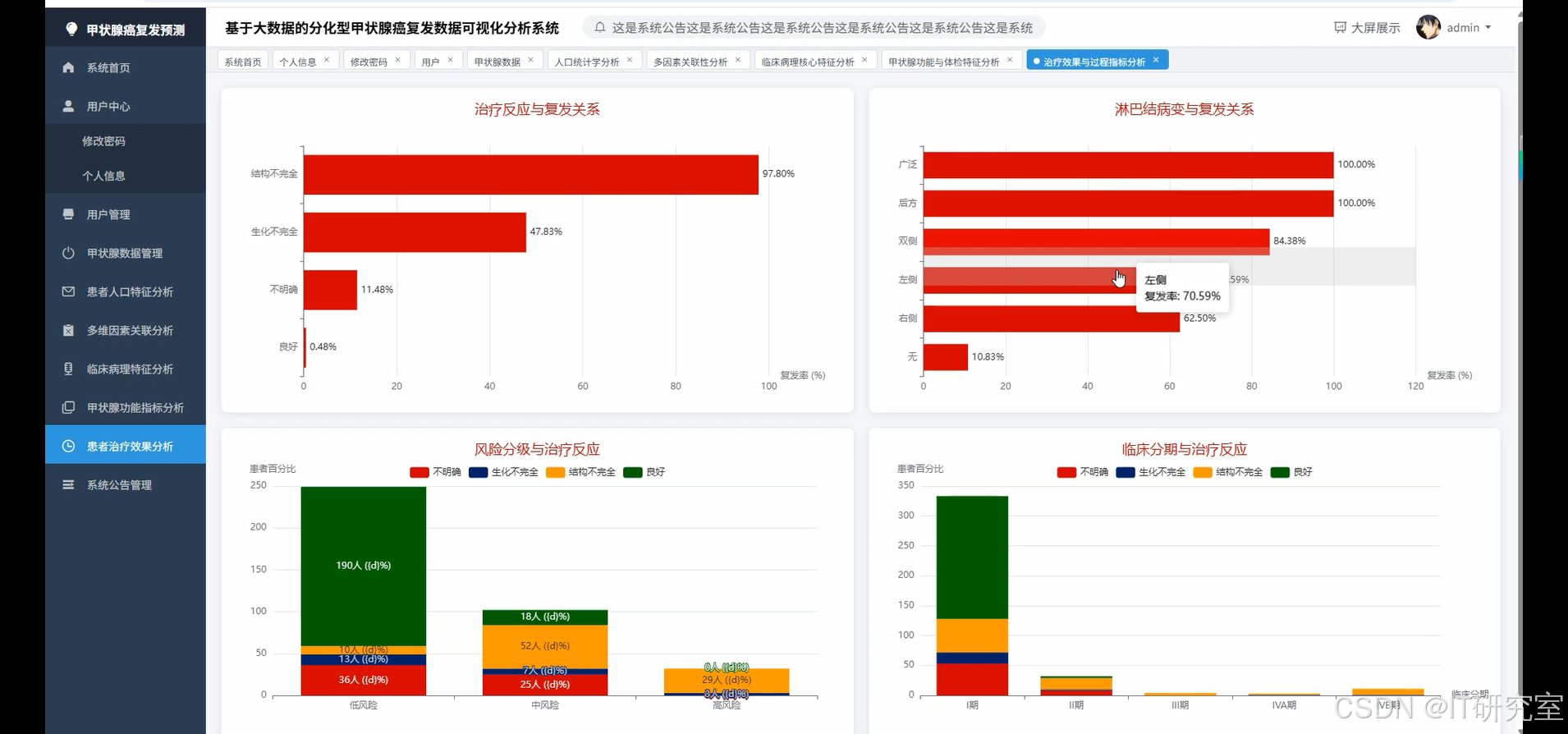
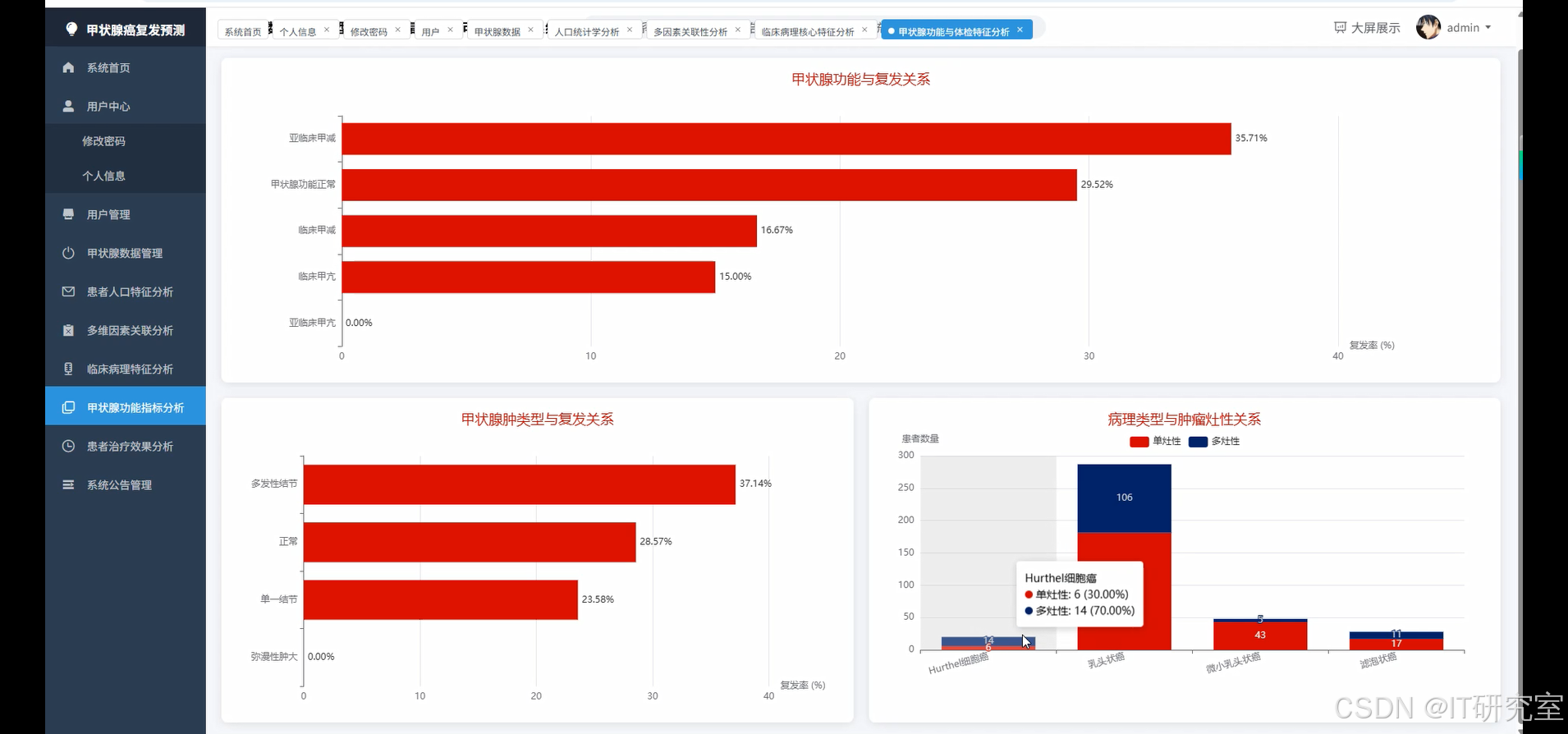
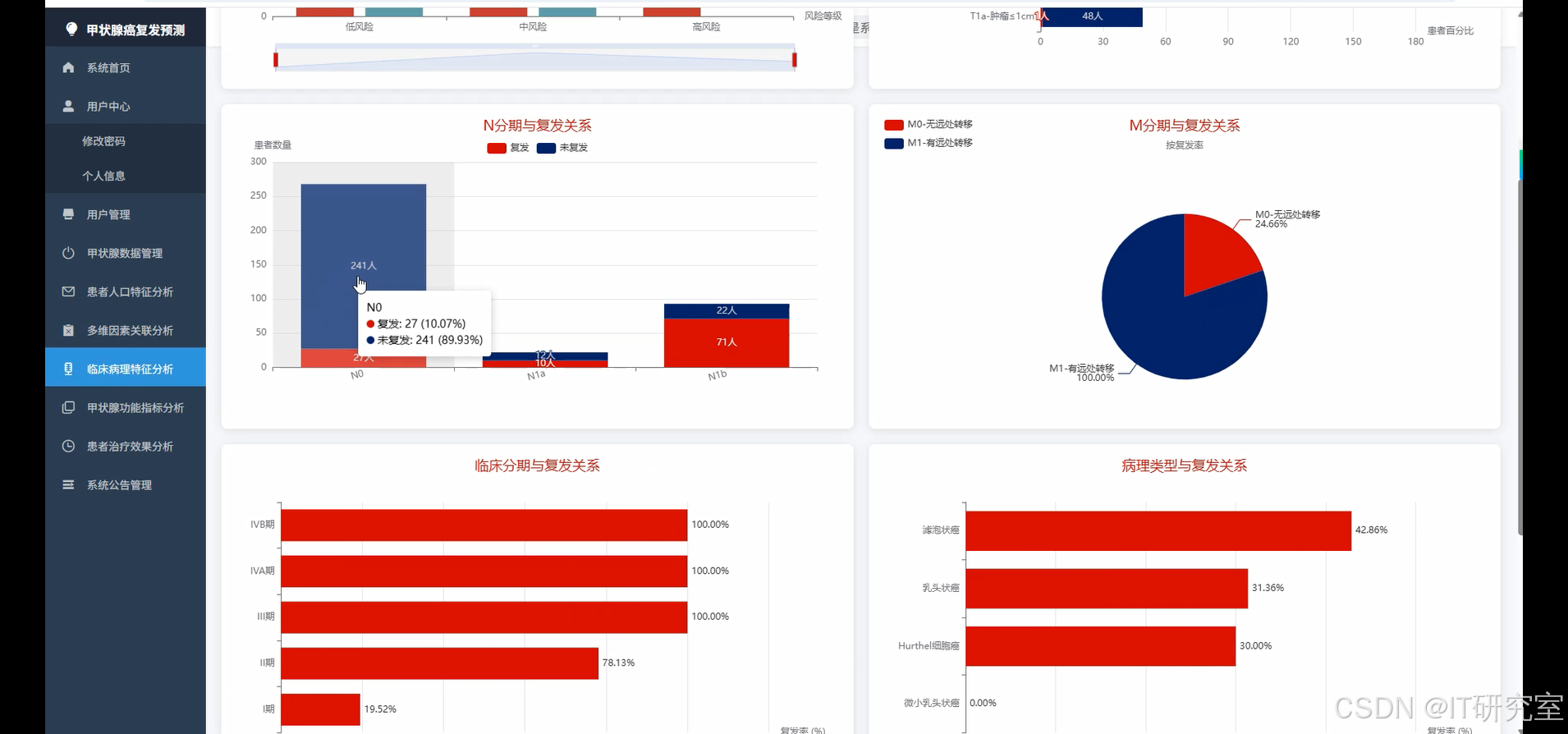
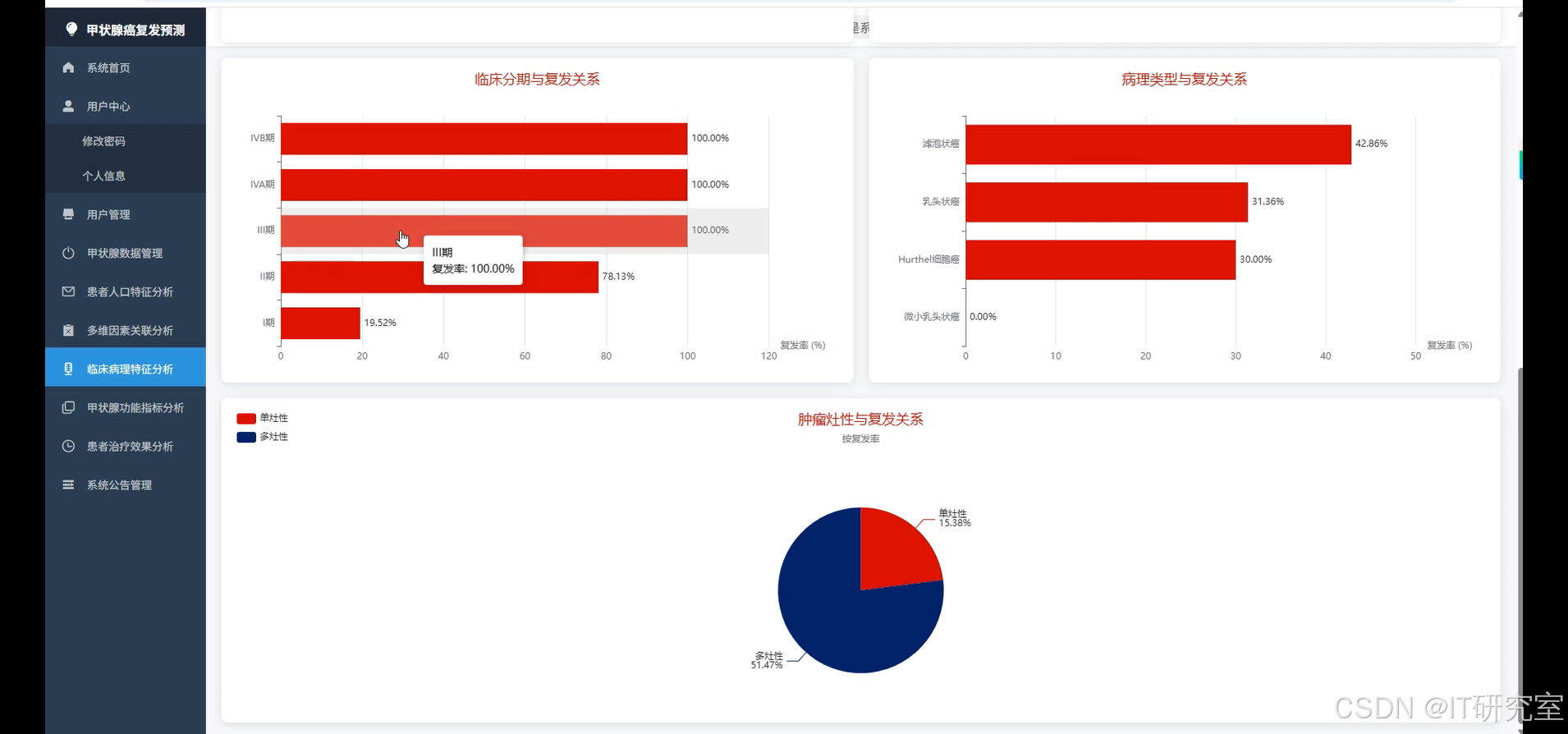
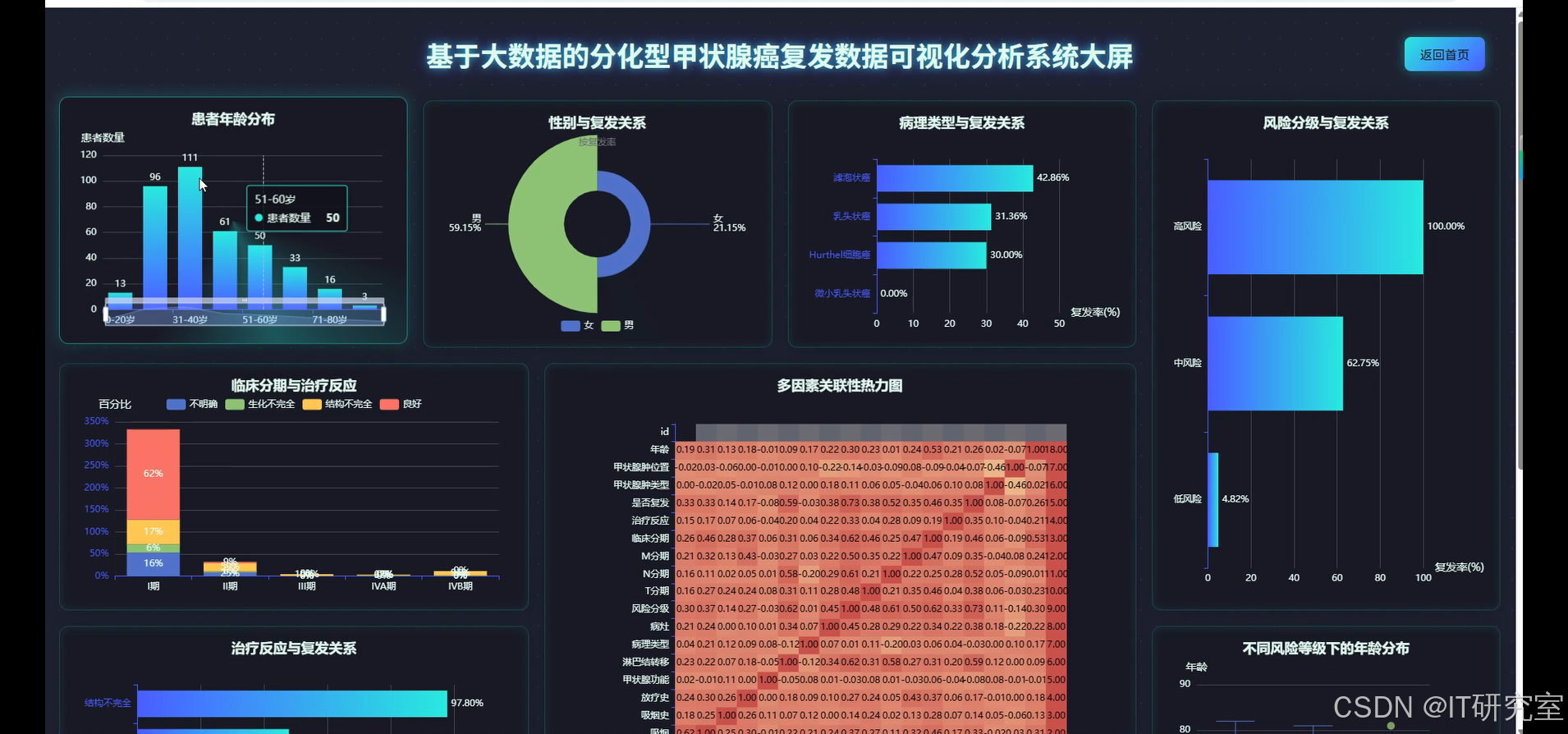
四、代码参考
- 项目实战代码参考:
java(贴上部分代码)
from pyspark.sql import SparkSession
from pyspark.sql.functions import col, when, count, avg, corr, collect_list
from pyspark.ml.feature import VectorAssembler
from pyspark.ml.stat import Correlation
import pandas as pd
import numpy as np
from django.http import JsonResponse
from django.views import View
import json
spark = SparkSession.builder.appName("ThyroidCancerAnalysis").config("spark.sql.adaptive.enabled", "true").getOrCreate()
class MultiFactorCorrelationAnalysis(View):
def post(self, request):
df = spark.read.format("jdbc").option("url", "jdbc:mysql://localhost:3306/thyroid_db").option("dbtable", "thyroid_data").option("user", "root").option("password", "password").load()
numeric_columns = ['Age', 'Gender_encoded', 'Smoking_encoded', 'Hx_Smoking_encoded', 'Hx_Radiothreapy_encoded', 'Thyroid_Function_encoded', 'Physical_Examination_encoded', 'Adenopathy_encoded', 'Pathology_encoded', 'Focality_encoded', 'Risk_encoded', 'T_encoded', 'N_encoded', 'M_encoded', 'Stage_encoded', 'Response_encoded', 'Recurred_encoded']
feature_assembler = VectorAssembler(inputCols=numeric_columns, outputCol="features")
feature_df = feature_assembler.transform(df)
correlation_matrix = Correlation.corr(feature_df, "features", "pearson").head()[0].toArray()
correlation_data = []
for i, col1 in enumerate(numeric_columns):
for j, col2 in enumerate(numeric_columns):
correlation_data.append({
'x_factor': col1,
'y_factor': col2,
'correlation_value': float(correlation_matrix[i][j])
})
recurrence_correlations = []
for i, column in enumerate(numeric_columns[:-1]):
corr_value = correlation_matrix[i][-1]
recurrence_correlations.append({
'factor_name': column,
'correlation_with_recurrence': float(corr_value),
'correlation_strength': 'strong' if abs(corr_value) > 0.5 else 'moderate' if abs(corr_value) > 0.3 else 'weak'
})
high_risk_factors = df.filter(col("Risk_encoded") == 2)
intermediate_risk_factors = df.filter(col("Risk_encoded") == 1)
low_risk_factors = df.filter(col("Risk_encoded") == 0)
risk_factor_analysis = {
'high_risk_recurrence_rate': high_risk_factors.filter(col("Recurred_encoded") == 1).count() / high_risk_factors.count(),
'intermediate_risk_recurrence_rate': intermediate_risk_factors.filter(col("Recurred_encoded") == 1).count() / intermediate_risk_factors.count(),
'low_risk_recurrence_rate': low_risk_factors.filter(col("Recurred_encoded") == 1).count() / low_risk_factors.count()
}
return JsonResponse({
'correlation_matrix': correlation_data,
'recurrence_correlations': recurrence_correlations,
'risk_factor_analysis': risk_factor_analysis,
'total_patients': df.count()
})
class PatientDemographicAnalysis(View):
def post(self, request):
df = spark.read.format("jdbc").option("url", "jdbc:mysql://localhost:3306/thyroid_db").option("dbtable", "thyroid_data").option("user", "root").option("password", "password").load()
age_distribution = df.withColumn("age_group", when(col("Age") < 30, "20-29").when(col("Age") < 40, "30-39").when(col("Age") < 50, "40-49").when(col("Age") < 60, "50-59").otherwise("60+")).groupBy("age_group").agg(count("*").alias("patient_count"), avg("Age").alias("avg_age")).collect()
gender_recurrence_analysis = df.groupBy("Gender").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
smoking_analysis = df.groupBy("Smoking", "Hx_Smoking").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
radiotherapy_analysis = df.groupBy("Hx_Radiothreapy").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
age_gender_cross_analysis = df.withColumn("age_group", when(col("Age") < 40, "Young").otherwise("Elder")).groupBy("age_group", "Gender").agg(count("*").alias("patient_count"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_count")).withColumn("recurrence_rate", col("recurred_count") / col("patient_count")).collect()
lifestyle_risk_factors = df.groupBy("Smoking", "Hx_Smoking", "Hx_Radiothreapy").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("combined_risk_score", when((col("Smoking") == "Yes") | (col("Hx_Smoking") == "Yes") | (col("Hx_Radiothreapy") == "Yes"), 1).otherwise(0)).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
demographic_summary = {
'total_patients': df.count(),
'average_age': df.agg(avg("Age")).collect()[0][0],
'gender_distribution': df.groupBy("Gender").count().collect(),
'overall_recurrence_rate': df.filter(col("Recurred") == "Yes").count() / df.count()
}
return JsonResponse({
'age_distribution': [row.asDict() for row in age_distribution],
'gender_recurrence_analysis': [row.asDict() for row in gender_recurrence_analysis],
'smoking_analysis': [row.asDict() for row in smoking_analysis],
'radiotherapy_analysis': [row.asDict() for row in radiotherapy_analysis],
'age_gender_cross_analysis': [row.asDict() for row in age_gender_cross_analysis],
'lifestyle_risk_factors': [row.asDict() for row in lifestyle_risk_factors],
'demographic_summary': demographic_summary
})
class ClinicalPathologyAnalysis(View):
def post(self, request):
df = spark.read.format("jdbc").option("url", "jdbc:mysql://localhost:3306/thyroid_db").option("dbtable", "thyroid_data").option("user", "root").option("password", "password").load()
tnm_staging_analysis = df.groupBy("T", "N", "M", "Stage").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).orderBy(col("recurrence_rate").desc()).collect()
pathology_type_analysis = df.groupBy("Pathology").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).orderBy(col("recurrence_rate").desc()).collect()
risk_stratification_analysis = df.groupBy("Risk").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
focality_analysis = df.groupBy("Focality").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
t_stage_detailed = df.groupBy("T").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("tumor_stage_risk", when(col("T").isin(["T3a", "T3b", "T4a", "T4b"]), "High").when(col("T").isin(["T2"]), "Intermediate").otherwise("Low")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
n_stage_detailed = df.groupBy("N").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("lymph_node_risk", when(col("N").isin(["N1a", "N1b"]), "High").otherwise("Low")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
pathology_focality_cross = df.groupBy("Pathology", "Focality").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
advanced_stage_analysis = df.filter(col("Stage").isin(["III", "IV", "IVA", "IVB"])).groupBy("Stage", "Pathology").agg(count("*").alias("total_patients"), count(when(col("Recurred") == "Yes", 1)).alias("recurred_patients")).withColumn("recurrence_rate", col("recurred_patients") / col("total_patients")).collect()
return JsonResponse({
'tnm_staging_analysis': [row.asDict() for row in tnm_staging_analysis],
'pathology_type_analysis': [row.asDict() for row in pathology_type_analysis],
'risk_stratification_analysis': [row.asDict() for row in risk_stratification_analysis],
'focality_analysis': [row.asDict() for row in focality_analysis],
't_stage_detailed': [row.asDict() for row in t_stage_detailed],
'n_stage_detailed': [row.asDict() for row in n_stage_detailed],
'pathology_focality_cross': [row.asDict() for row in pathology_focality_cross],
'advanced_stage_analysis': [row.asDict() for row in advanced_stage_analysis]
})五、系统视频
基于大数据的分化型甲状腺癌复发数据可视化分析系统项目视频:
大数据毕业设计选题推荐-基于大数据的分化型甲状腺癌复发数据可视化分析系统-Spark-Hadoop-Bigdata
结语
大数据毕业设计选题推荐-基于大数据的分化型甲状腺癌复发数据可视化分析系统-Spark-Hadoop-Bigdata
想看其他类型的计算机毕业设计作品也可以和我说~ 谢谢大家!
有技术这一块问题大家可以评论区交流或者私我~
大家可以帮忙点赞、收藏、关注、评论啦~
源码获取:⬇⬇⬇