做了转录组或者其他组学,有对应样本的多个特征数据,然后做相关性分析并绘制热图。示例热图见下图【数据是随机生成的,所以结果没几个显著相关的,实际数据过程中会比较多】:
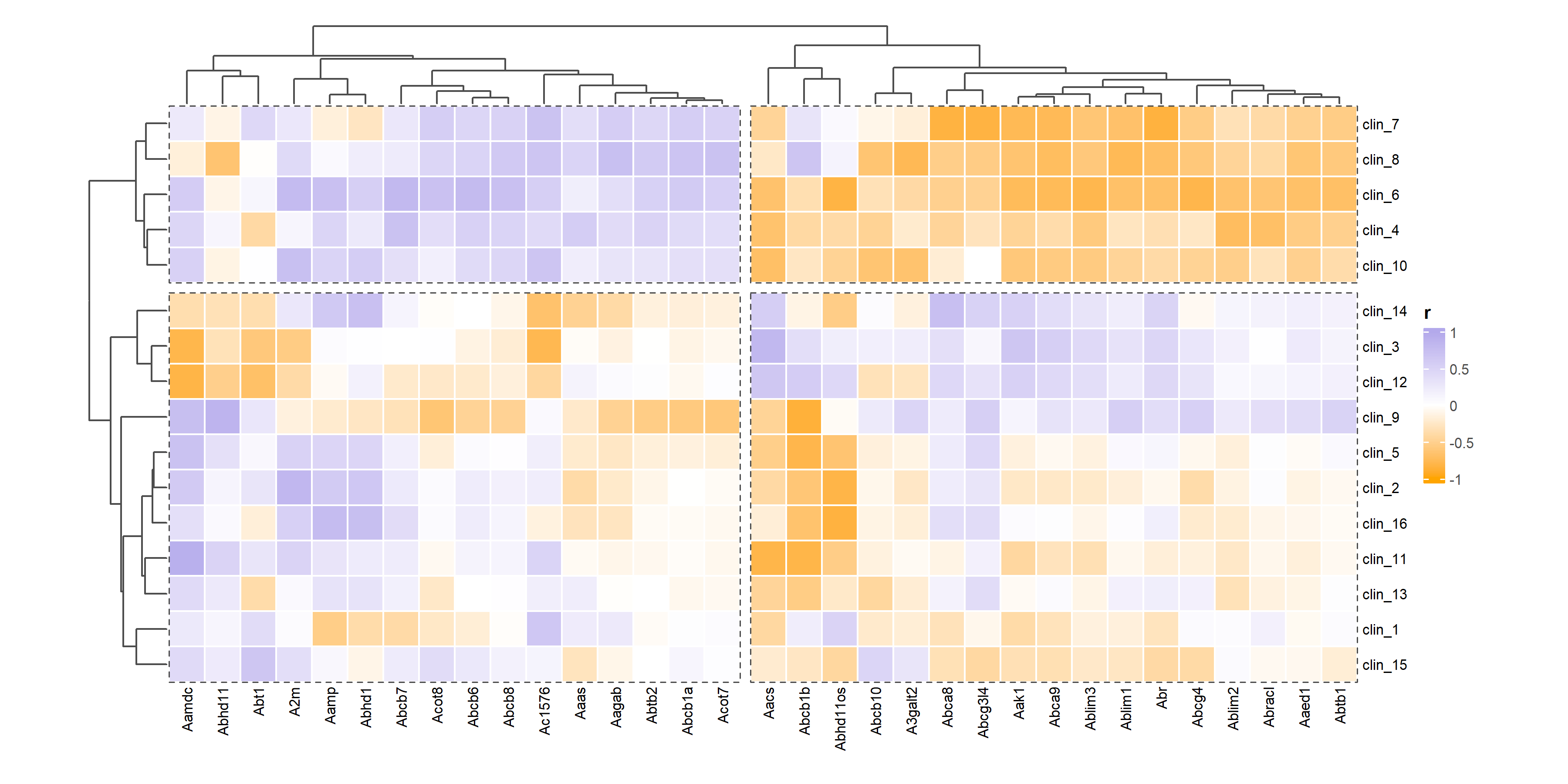
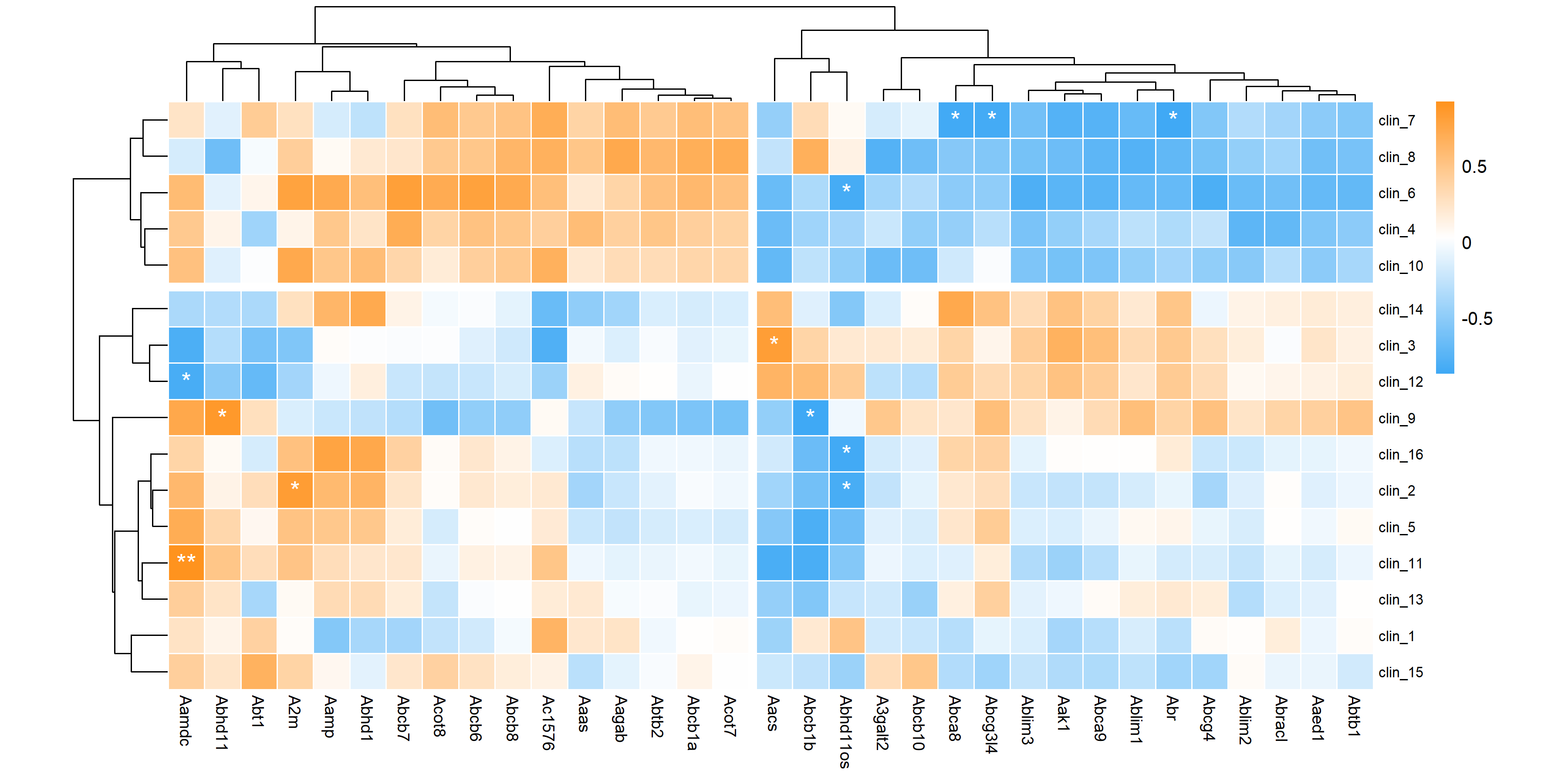
注:不适用于线性回归分析,后续会更新线性回归分析、WGCNA等更进一层次的脚本。
本脚本改一下对应的两个矩阵,换成你自己的就可以用了,示例数据在附件中,格式为第一列为基因,其余列为样本对应的数据;另一个 矩阵第一列为指标,其余列为第一个矩阵对应样本的指标。注:两个矩阵样本必须一致。
脚本如下:
R
# 基因表达与化学指标相关性分析及可视化脚本
# 功能:计算基因表达数据与化学指标间的相关性,输出结果并绘制热图
# 作者:未知(优化版)
# 日期:2025-10-05
#######################################
# 1. 环境准备与初始化
#######################################
# 清除当前环境中的所有变量,避免干扰
rm(list = ls())
# 设置工作目录(根据实际情况修改)
# setwd("/path/to/your/workdirectory")
# 安装必要的R包(首次运行时取消注释并安装)
# install.packages(c("openxlsx", "dplyr", "psych", "pheatmap"))
# if (!require("BiocManager", quietly = TRUE))
# install.packages("BiocManager")
# BiocManager::install(c("ComplexHeatmap", "circlize"))
# 加载所需R包
library(openxlsx) # 用于读取和写入Excel文件
library(dplyr) # 用于数据处理和转换
library(psych) # 用于相关性分析
library(ComplexHeatmap) # 用于绘制复杂热图
library(circlize) # 用于颜色映射
library(pheatmap) # 用于绘制热图
library(grid) # 用于绘图辅助
#######################################
# 2. 数据导入与预处理
#######################################
# 导入基因表达数据(TPM格式)
# 注意:确保"gene_TPM.xlsx"文件与脚本在同一目录或提供完整路径
exp <- read.xlsx("gene_TPM.xlsx", rowNames = TRUE)
# 预览数据(如需查看数据,取消下一行注释)
# View(exp)
# 导入临床化学指标数据
chem <- read.xlsx("clin.xlsx", rowNames = TRUE)
# 预览数据(如需查看数据,取消下一行注释)
# View(chem)
# 数据转置:使样本作为行,基因/化学指标作为列
# 这是因为相关性分析要求样本为行,变量为列
expt <- t(exp) # 转置基因表达数据
chemt <- t(chem) # 转置化学指标数据
#######################################
# 3. 相关性计算
#######################################
# 计算基因表达与化学指标之间的Pearson相关性
# 参数说明:
# method: 相关系数计算方法,可选"pearson"、"spearman"或"kendall"
# use: 缺失值处理方式,"pairwise"表示成对删除,"complete"表示完全删除
# adjust: p值校正方法,"fdr"表示控制错误发现率
ct <- corr.test(
x = expt, # 基因表达数据(行:样本,列:基因)
y = chemt, # 化学指标数据(行:样本,列:化学指标)
method = "pearson", # 使用Pearson相关系数
use = "pairwise", # 成对删除缺失值
adjust = "fdr" # 使用FDR校正p值
)
#######################################
# 4. 相关性结果提取与整理
#######################################
# 从相关性分析结果中提取关键矩阵
r_matrix <- ct$r # 相关系数矩阵
p_matrix <- ct$p # 原始p值矩阵
padj_matrix <- ct$p.adj # 校正后的p值矩阵
# 定义显著性星号标记函数
# 根据p值或校正后的p值返回对应的显著性星号
get_significance_stars <- function(p_values) {
case_when(
p_values < 0.001 ~ "***", # p < 0.001,极显著
p_values < 0.01 ~ "**", # 0.001 ≤ p < 0.01,高度显著
p_values < 0.05 ~ "*", # 0.01 ≤ p < 0.05,显著
TRUE ~ "" # p ≥ 0.05,不显著
)
}
#######################################
# 5. 结果输出到Excel文件
#######################################
# 1) 输出相关系数矩阵
write.xlsx(r_matrix, "1_correlation_coefficients.xlsx", rowNames = TRUE)
# 2) 输出原始p值矩阵
write.xlsx(p_matrix, "2_p_values.xlsx", rowNames = TRUE)
# 3) 输出校正后的p值矩阵
write.xlsx(padj_matrix, "3_adjusted_p_values.xlsx", rowNames = TRUE)
# 4) 输出带原始p值显著性星号的相关系数矩阵
r_with_p_stars <- r_matrix
for (i in seq_len(nrow(r_matrix))) {
for (j in seq_len(ncol(r_matrix))) {
r_with_p_stars[i, j] <- sprintf(
"%.3f%s",
r_matrix[i, j],
get_significance_stars(p_matrix[i, j])
)
}
}
write.xlsx(r_with_p_stars, "4_correlation_with_p_stars.xlsx", rowNames = TRUE)
# 5) 输出带校正p值显著性星号的相关系数矩阵
r_with_padj_stars <- r_matrix
for (i in seq_len(nrow(r_matrix))) {
for (j in seq_len(ncol(r_matrix))) {
r_with_padj_stars[i, j] <- sprintf(
"%.3f%s",
r_matrix[i, j],
get_significance_stars(padj_matrix[i, j])
)
}
}
write.xlsx(r_with_padj_stars, "5_correlation_with_padj_stars.xlsx", rowNames = TRUE)
#######################################
# 6. 可视化数据准备
#######################################
# 转置矩阵并格式化,为绘图做准备
r_values <- round(t(r_matrix), 3) # 转置并保留3位小数的相关系数
p_values <- round(t(p_matrix), 5) # 转置并保留5位小数的p值
padj_values <- round(t(padj_matrix), 5) # 转置并保留5位小数的校正p值
# 将p值转换为显著性星号(用于绘图)
p_stars <- p_values
p_stars[p_stars >= 0 & p_stars < 0.001] <- "***"
p_stars[p_stars >= 0.001 & p_stars < 0.01] <- "**"
p_stars[p_stars >= 0.01 & p_stars < 0.05] <- "*"
p_stars[p_stars >= 0.05] <- ""
# 将校正p值转换为显著性星号(用于绘图)
padj_stars <- padj_values
padj_stars[padj_stars >= 0 & padj_stars < 0.001] <- "***"
padj_stars[padj_stars >= 0.001 & padj_stars < 0.01] <- "**"
padj_stars[padj_stars >= 0.01 & padj_stars < 0.05] <- "*"
padj_stars[padj_stars >= 0.05] <- ""
#######################################
# 7. 热图绘制函数(复用函数)
#######################################
# 定义热图参数设置函数,避免代码重复
get_heatmap_parameters <- function(data_matrix) {
# 计算热图尺寸
cell_width <- 0.7
cell_height <- 0.7
col_num <- ncol(data_matrix)
row_num <- nrow(data_matrix)
list(
cellwidth = cell_width,
cellheight = cell_height,
width = unit(cell_width * col_num, "cm"),
height = unit(cell_height * row_num, "cm"),
# 颜色映射:从橙色到白色再到紫色,对应相关系数从-1到1
col_fun = colorRamp2(c(-1, 0, 1), c("#ffa500", "white", "#B3A9EB")),
# 聚类参数
row_split = 2,
column_split = 2,
# 文本样式
text_gp = gpar(fontsize = 8)
)
}
# 定义保存图形的函数
save_heatmap <- function(plot_object, filename_prefix, width = 12, height = 6) {
# 保存为PDF格式(矢量图,适合印刷)
pdf(paste0(filename_prefix, ".pdf"), width = width, height = height)
draw(plot_object)
dev.off()
# 保存为PNG格式(位图,适合展示)
png(
paste0(filename_prefix, ".png"),
width = width,
height = height,
units = "in",
res = 300 # 高分辨率
)
draw(plot_object)
dev.off()
}
#######################################
# 8. 基于p值的相关性热图绘制
#######################################
# 获取热图参数
heatmap_params <- get_heatmap_parameters(r_values)
# 1) 仅显示相关系数的热图
heatmap_p1 <- Heatmap(
matrix = r_values,
name = "r", # 图例名称
col = heatmap_params$col_fun, # 颜色映射
width = heatmap_params$width,
height = heatmap_params$height,
rect_gp = gpar(col = "white", lwd = 1.5), # 单元格边框
# 聚类树设置
column_dend_height = unit(1.5, "cm"),
row_dend_width = unit(1.5, "cm"),
column_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_dend_gp = gpar(col = "gray30", lwd = 1.4),
# 聚类拆分
row_split = heatmap_params$row_split,
column_split = heatmap_params$column_split,
# 标签设置
row_title = NULL,
column_title = NULL,
column_names_gp = heatmap_params$text_gp,
row_names_gp = heatmap_params$text_gp,
# 图例设置
heatmap_legend_param = list(
legend_height = unit(3, "cm"),
grid_width = unit(0.4, "cm"),
labels_gp = gpar(col = "gray30", fontsize = 8)
)
)
# 保存热图
save_heatmap(heatmap_p1, "correlation_heatmap_pvalue_based")
# 2) 显示相关系数和p值显著性星号的热图
heatmap_p2 <- Heatmap(
matrix = r_values,
name = "r",
col = heatmap_params$col_fun,
width = heatmap_params$width,
height = heatmap_params$height,
rect_gp = gpar(col = "white", lwd = 1.5),
border_gp = gpar(col = "gray30", lty = 2, lwd = 1.2), # 边框样式
# 聚类设置
column_dend_height = unit(1.5, "cm"),
row_dend_width = unit(1.5, "cm"),
column_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_split = heatmap_params$row_split,
column_split = heatmap_params$column_split,
row_gap = unit(2, "mm"),
column_gap = unit(2, "mm"),
# 标签设置
row_title = NULL,
column_title = NULL,
column_names_gp = heatmap_params$text_gp,
row_names_gp = heatmap_params$text_gp,
# 图例设置
heatmap_legend_param = list(
legend_height = unit(3, "cm"),
grid_width = unit(0.4, "cm"),
labels_gp = gpar(col = "gray30", fontsize = 8)
),
# 在单元格中添加显著性星号
cell_fun = function(j, i, x, y, width, height, fill) {
grid.text(
p_stars[i, j],
x, y,
vjust = 0.7, # 垂直调整
gp = gpar(fontsize = 13, col = "white") # 星号样式
)
}
)
# 保存热图
save_heatmap(heatmap_p2, "correlation_heatmap_with_pvalue_stars")
# 3) 使用pheatmap包绘制带p值星号的热图
# 定义颜色梯度
pheatmap_colors <- colorRampPalette(c("#3FA9F5", "white", "#FF931E"))(200)
# 绘制热图
pheatmap_p <- pheatmap(
mat = r_values,
scale = "none", # 不进行数据标准化
border_color = "white", # 单元格边框颜色
number_color = "white", # 数字颜色
fontsize_number = 14, # 数字字体大小
fontsize_row = 8, # 行名字体大小
fontsize_col = 9, # 列名字体大小
cellwidth = 20, # 单元格宽度
cellheight = 20, # 单元格高度
cutree_rows = 2, # 行聚类数量
cutree_cols = 2, # 列聚类数量
cluster_rows = TRUE, # 行聚类
cluster_cols = TRUE, # 列聚类
color = pheatmap_colors, # 颜色方案
display_numbers = p_stars,# 显示显著性星号
show_rownames = TRUE # 显示行名
)
# 保存pheatmap绘制的热图
pdf("correlation_pheatmap_pvalue_based.pdf", width = 12, height = 6)
grid.draw(pheatmap_p$gtable)
dev.off()
png(
"correlation_pheatmap_pvalue_based.png",
width = 12,
height = 6,
units = "in",
res = 300
)
grid.draw(pheatmap_p$gtable)
dev.off()
#######################################
# 9. 基于校正p值的相关性热图绘制
#######################################
# 1) 仅显示相关系数的热图(基于校正p值)
heatmap_padj1 <- Heatmap(
matrix = r_values,
name = "r",
col = heatmap_params$col_fun,
width = heatmap_params$width,
height = heatmap_params$height,
rect_gp = gpar(col = "white", lwd = 1.5),
# 聚类设置
column_dend_height = unit(1.5, "cm"),
row_dend_width = unit(1.5, "cm"),
column_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_split = heatmap_params$row_split,
column_split = heatmap_params$column_split,
# 标签设置
row_title = NULL,
column_title = NULL,
column_names_gp = heatmap_params$text_gp,
row_names_gp = heatmap_params$text_gp,
# 图例设置
heatmap_legend_param = list(
legend_height = unit(3, "cm"),
grid_width = unit(0.4, "cm"),
labels_gp = gpar(col = "gray30", fontsize = 8)
)
)
# 保存热图
save_heatmap(heatmap_padj1, "correlation_heatmap_padj_based")
# 2) 显示相关系数和校正p值显著性星号的热图
heatmap_padj2 <- Heatmap(
matrix = r_values,
name = "r",
col = heatmap_params$col_fun,
width = heatmap_params$width,
height = heatmap_params$height,
rect_gp = gpar(col = "white", lwd = 1.5),
border_gp = gpar(col = "gray30", lty = 2, lwd = 1.2),
# 聚类设置
column_dend_height = unit(1.5, "cm"),
row_dend_width = unit(1.5, "cm"),
column_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_dend_gp = gpar(col = "gray30", lwd = 1.4),
row_split = heatmap_params$row_split,
column_split = heatmap_params$column_split,
row_gap = unit(2, "mm"),
column_gap = unit(2, "mm"),
# 标签设置
row_title = NULL,
column_title = NULL,
column_names_gp = heatmap_params$text_gp,
row_names_gp = heatmap_params$text_gp,
# 图例设置
heatmap_legend_param = list(
legend_height = unit(3, "cm"),
grid_width = unit(0.4, "cm"),
labels_gp = gpar(col = "gray30", fontsize = 8)
),
# 在单元格中添加校正p值的显著性星号
cell_fun = function(j, i, x, y, width, height, fill) {
grid.text(
padj_stars[i, j],
x, y,
vjust = 0.7,
gp = gpar(fontsize = 13, col = "white")
)
}
)
# 保存热图
save_heatmap(heatmap_padj2, "correlation_heatmap_with_padj_stars")
# 3) 使用pheatmap包绘制带校正p值星号的热图
pheatmap_padj <- pheatmap(
mat = r_values,
scale = "none",
border_color = "white",
number_color = "white",
fontsize_number = 14,
fontsize_row = 8,
fontsize_col = 9,
cellwidth = 20,
cellheight = 20,
cutree_rows = 2,
cutree_cols = 2,
cluster_rows = TRUE,
cluster_cols = TRUE,
color = pheatmap_colors,
display_numbers = padj_stars, # 显示校正p值的显著性星号
show_rownames = TRUE
)
# 保存pheatmap绘制的热图
pdf("correlation_pheatmap_padj_based.pdf", width = 12, height = 6)
grid.draw(pheatmap_padj$gtable)
dev.off()
png(
"correlation_pheatmap_padj_based.png",
width = 12,
height = 6,
units = "in",
res = 300
)
grid.draw(pheatmap_padj$gtable)
dev.off()
# 分析完成提示
cat("分析完成!结果文件已保存到工作目录。\n")
cat("Excel结果文件包括:相关系数、p值、校正p值及带显著性星号的结果。\n")
cat("热图文件包括:基于p值和校正p值的多种热图(PDF和PNG格式)。\n")