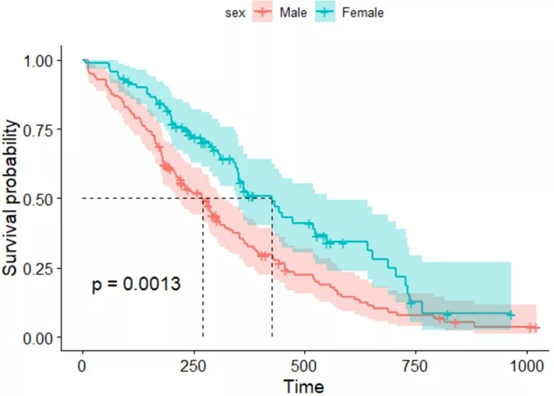
应粉丝要求,需要手写一个计算生存分析C指数(C-index)的函数。我在既往文章有介绍:C指数是评估生存模型预测性能的核心指标,其思想源自大神Harrell's C-statistic。它衡量的是:模型对任意两个个体发生事件风险的排序能力。
- 核心思想
对于一对可比较的个体 (i, j):
若模型预测的风险 risk_i > risk_j,
且实际观察到 time_i < time_j(即个体 i 比 j 更早发生事件),
则这对个体被认为是"一致的"(concordant)。
C指数就是所有可比较对中,"一致对"的比例。

用通俗的话来说,咱们以死亡举例子,比如咱们预测A的死亡率高于B,那么A的生存时间是不是要短于B?,要是咱们计算A和B的时候,A死亡率高B,而且A的生存时间短于B,那么咱们就说A和B具有一致性。因此,咱们需要的参数有:死亡结局,生存时间和预测的死亡率。
概念很简单,但实际操作还是有许多情况要考虑的,
比如:
1.在A和B都挂了,就直接比较AB的生存时间
2.A挂了,B还没挂,这个需要份两个情况,A风险大于B和A风险小于B
3.A没挂,B挂了,也是要分两种情况,A风险大于B和A风险小于B
下面导入个实际数据来演示一下:自用的是survival包自带的癌症数据
r
library(survival)
library(rms)
bc<-cancer
bc$status<-ifelse(bc$status==1,0,1)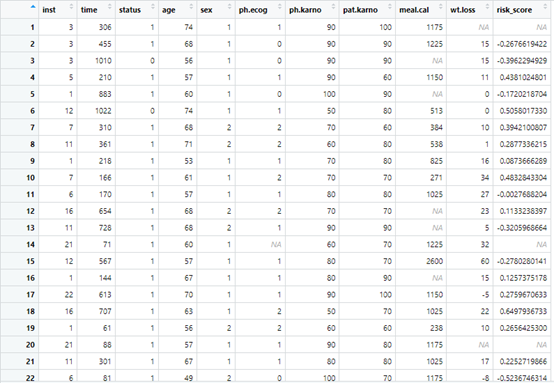
Status是结局指标,time是时间,其他是协变量
刚才咱们知道:咱们需要的参数有:死亡结局,生存时间和预测的死亡率。因此先组建模型
r
dc<-datadist(bc)
options(datadist="dc")
f <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss,
x=T, y=T, surv=T, data=cancer, time.inc=36)生成预测概率,这个预测概率就是咱们的三要素中的预测的死亡率
r
bc$risk_score<-predict(f)既然三个指标都有了,咱们就可以开干计算了
先生成所有的基础指标
r
n <- length(time)
concordant <- 0
discordant <- 0
tied_risk <- 0
total_pairs <- 0 写个循环,要考虑上面三个情况
r
for (i in 1:(n-1)) {
for (j in (i+1):n) {
if (time[i] != time[j] && (status[i] == 1 || status[j] == 1)) {
total_pairs <- total_pairs + 1
if (time[i] < time[j]) {
if (risk[i] > risk[j]) {
concordant <- concordant + 1
} else if (risk[i] < risk[j]) {
discordant <- discordant + 1
} else {
tied_risk <- tied_risk + 1
}
} else {
if (risk[j] > risk[i]) {
concordant <- concordant + 1
} else if (risk[j] < risk[i]) {
discordant <- discordant + 1
} else {
tied_risk <- tied_risk + 1
}
}
}
}
}最后进行计算
r
c_index <- (concordant + 0.5 * tied_risk) / total_pairs咱们把上面步骤封装成一个函数,进行计算一下,约等于0.658

那这个结果对不对呢,咱们使用RMS包自带的rcorrcens函数和survival包的concordance函数验证一下
r
concordance(f)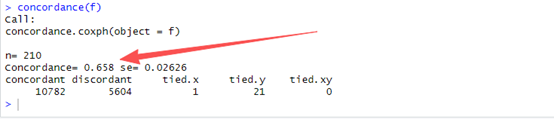
也是0.658,结果一模一样,再试下RMS包自带的rcorrcens函数
r
rcorrcens(Surv(time, status) ~ predict(f), data = bc)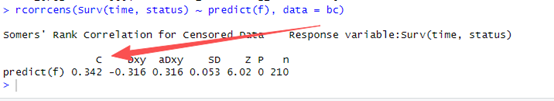
1-0.342=0.658,因此,三个函数计算的结果都是一模一样,咱们计算完全没有问题,玩美复现了生存分析C指数计算,但是我这个走的是for循环,数据量大的话可能要等一等。
新的问题来复杂加权数据也就nhanes数据的生存分析C指数怎么算?了解了原理,计算也就非常容易了,下次再介绍。