承接上一篇博客:https://blog.csdn.net/weixin_62528784/article/details/155135250?spm=1001.2014.3001.5502
本篇博客主要是我参与学习EMBL-EBI关于Alphafold培训课程的最后一篇笔记,
官网相关教程信息,参考:https://www.ebi.ac.uk/training/online/courses/alphafold/
最后也是完整结束了整个培训课程!
文章目录
- [一,AlphaFold 3 和 AlphaFold 服务器](#一,AlphaFold 3 和 AlphaFold 服务器)
-
- [1,介绍 AlphaFold 3](#1,介绍 AlphaFold 3)
-
- [扩展 AlphaFold 的应用范围](#扩展 AlphaFold 的应用范围)
- [AlphaFold 3 输入概述](#AlphaFold 3 输入概述)
- 如何访问AlphaFold3?
- [2,AlphaFold 3 是如何工作的?](#2,AlphaFold 3 是如何工作的?)
-
- [Tokenisation 分词](#Tokenisation 分词)
- [Diffusion 扩散](#Diffusion 扩散)
- [AlphaFold 3 输出概述](#AlphaFold 3 输出概述)
- [3,AlphaFold 3 面临的问题](#3,AlphaFold 3 面临的问题)
- [4,我应该使用 AlphaFold 2 还是 AlphaFold 3?](#4,我应该使用 AlphaFold 2 还是 AlphaFold 3?)
- [5,AlphaFold 3 的预测结果是如何得到验证的?](#5,AlphaFold 3 的预测结果是如何得到验证的?)
- [6,AlphaFold 服务器:通往 AlphaFold 3 的入口](#6,AlphaFold 服务器:通往 AlphaFold 3 的入口)
-
- [AlphaFold Server 能做什么?](#AlphaFold Server 能做什么?)
- [AlphaFold Server 不能做什么?](#AlphaFold Server 不能做什么?)
- [7,使用 AlphaFold Server 生成预测结果的逐步指南](#7,使用 AlphaFold Server 生成预测结果的逐步指南)
- [8,AlphaFold Server 的高级功能](#8,AlphaFold Server 的高级功能)
-
- [JSON 任务提交](#JSON 任务提交)
- 采样多个种子
- 复现任务(seed)
- [9,解读AlphaFold Server输出的结果](#9,解读AlphaFold Server输出的结果)
-
- [AlphaFold Server 提供的输出](#AlphaFold Server 提供的输出)
- 置信度指标
- [10,如何评估 AlphaFold 3 预测的质量](#10,如何评估 AlphaFold 3 预测的质量)
-
- [AlphaFold 3 中的 pLDDT 分数](#AlphaFold 3 中的 pLDDT 分数)
- [AlphaFold 3 中的 PAE 分数](#AlphaFold 3 中的 PAE 分数)
- [pTM 和 ipTM,每链评分和成对评分](#pTM 和 ipTM,每链评分和成对评分)
- [11,使用 AlphaFold 3 源代码](#11,使用 AlphaFold 3 源代码)
-
- 初始结构预测的注意事项
- [AlphaFold 3 输入格式](#AlphaFold 3 输入格式)
- [与 AlphaFold Server JSON 兼容](#与 AlphaFold Server JSON 兼容)
- 特殊考虑
- 12,总结
- 13,课程ppt下载
- 二,术语表
一,AlphaFold 3 和 AlphaFold 服务器
现在,我们终于来到了AlphaFold3,前面讲了那么多的AlphaFold2系列工具以及工作。

从本节开始,我们需要掌握一定的AlphaFold2前置知识,也就是前面我两篇博客记录的笔记。
包括对置信度分数、优势及局限性的理解,以及对底层技术的掌握,因为AlphaFold2和3底层的东西是一样的。
同样的,本节培训的目标如下:

1,介绍 AlphaFold 3

扩展 AlphaFold 的应用范围



官网所给出的这几个例子,都是前面AlphaFold2时代局限性,但是我们又不得不考虑并且更新迭代完善所需要的功能,
比如说前面multimer才能做的复合物建模,现在我们3也能做了,我们会考虑蛋白质和DNA等非常复杂的互作关系。
还有化学修饰,比如说是在蛋白质中非常广泛的翻译后修饰。


统一泛化的AlphaFold3。

还有一个RNA结构预测建模的问题:

官网培训教程给出的一些例子:在多模态分子结构建模方面(核酸、蛋白质模态)
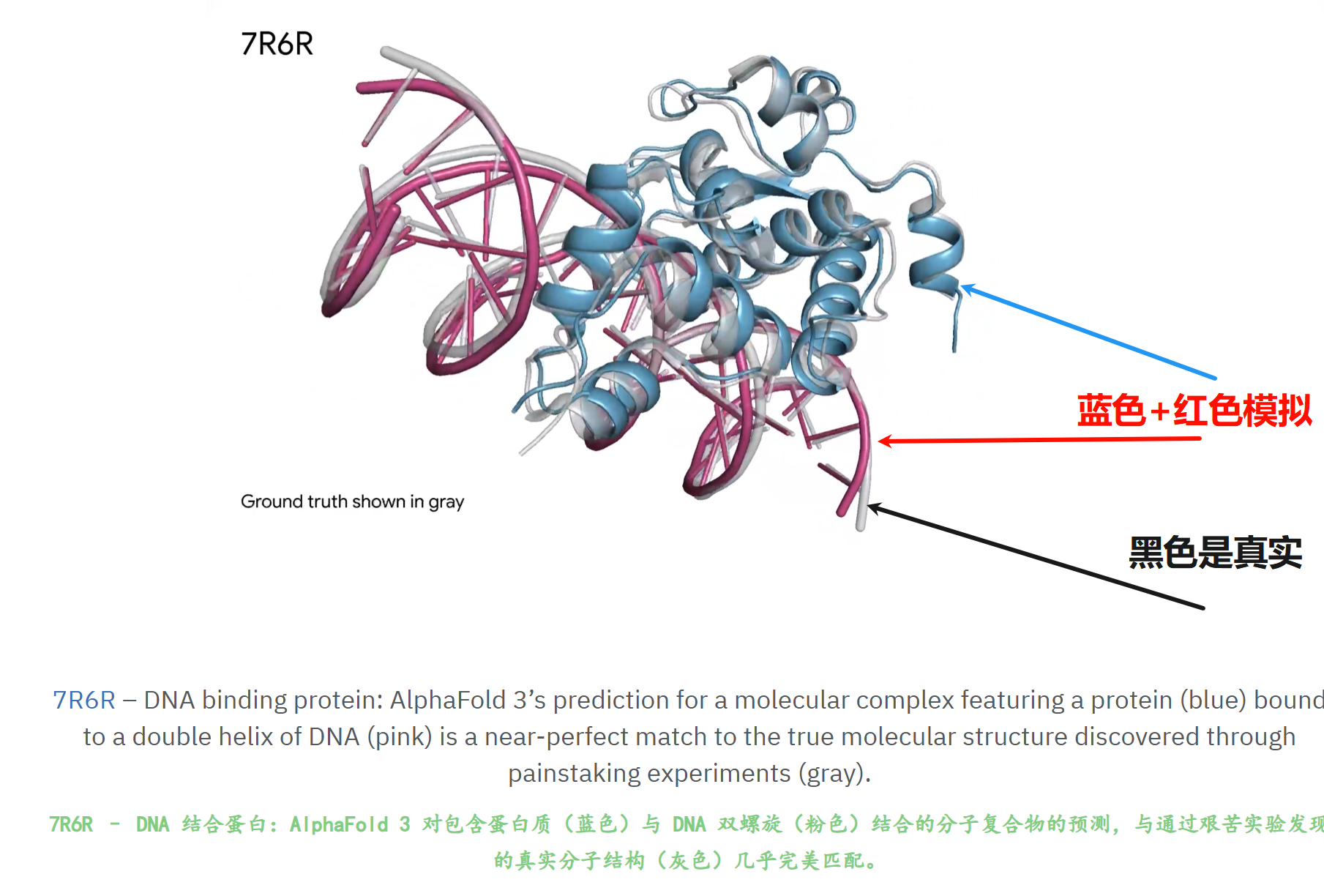
抗原-抗体-糖的超大复合物:

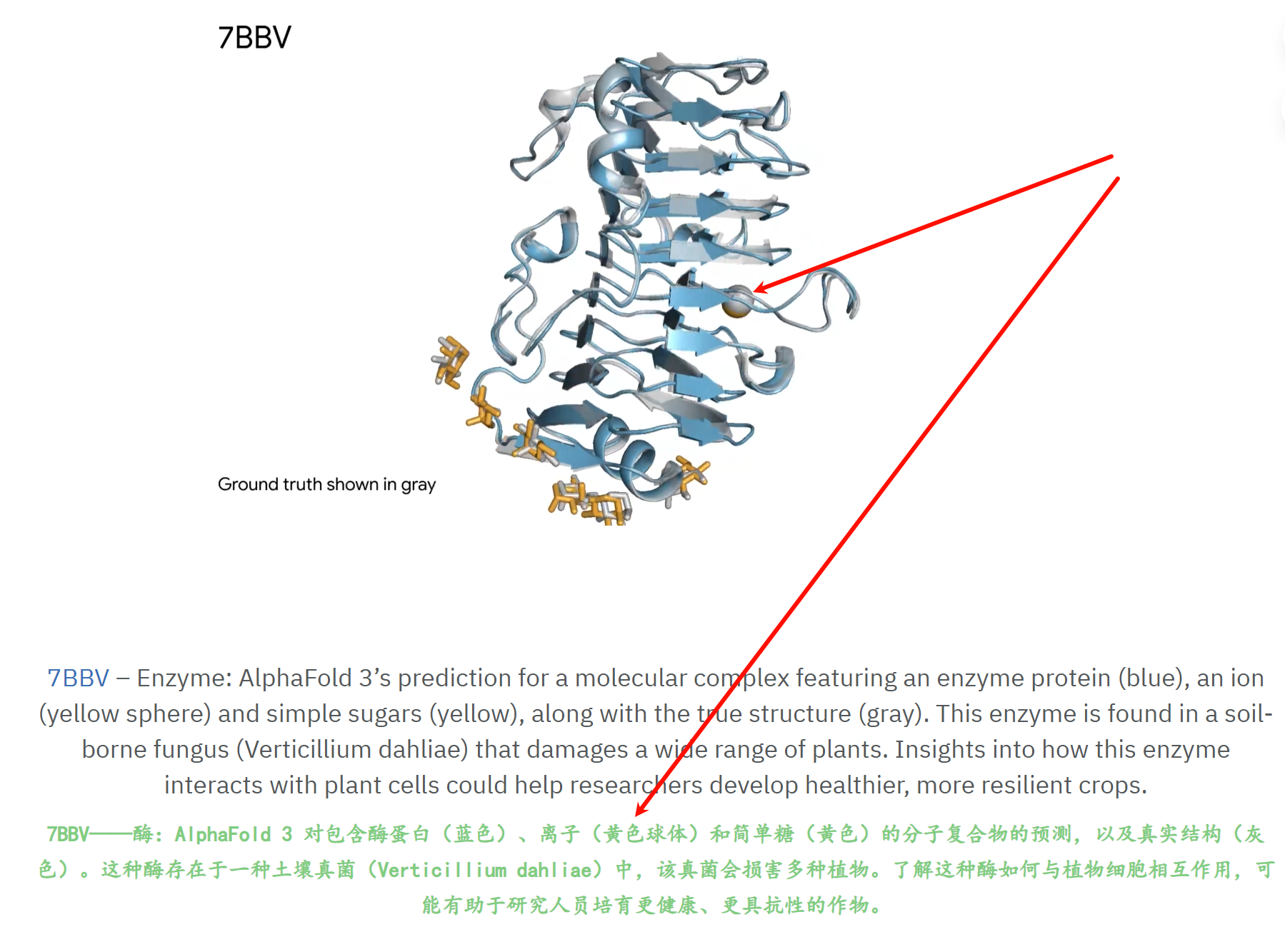
还有配体的模拟:

AlphaFold 3 输入概述

关于化学成分词典,参考PDB官网:https://www.wwpdb.org/data/ccd
我在之前的博客<如何批量提交AlphaFold3 web server json服务>中提到过这个标准,
https://blog.csdn.net/weixin_62528784/article/details/154954399?spm=1001.2014.3001.5502

 -
-
如何访问AlphaFold3?

其中github仓库网址参考:https://github.com/google-deepmind/alphafold3
使用需求参考:https://github.com/google-deepmind/alphafold3/blob/main/WEIGHTS_TERMS_OF_USE.md
2,AlphaFold 3 是如何工作的?


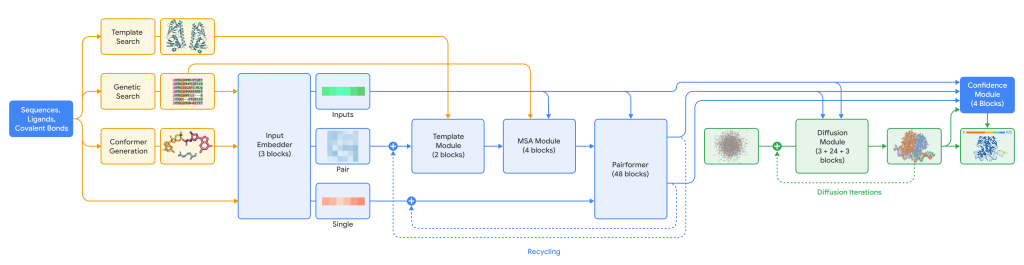

更正:AlphaFold3还是保留了MSA多序列比对部分的信息,以及模块处理


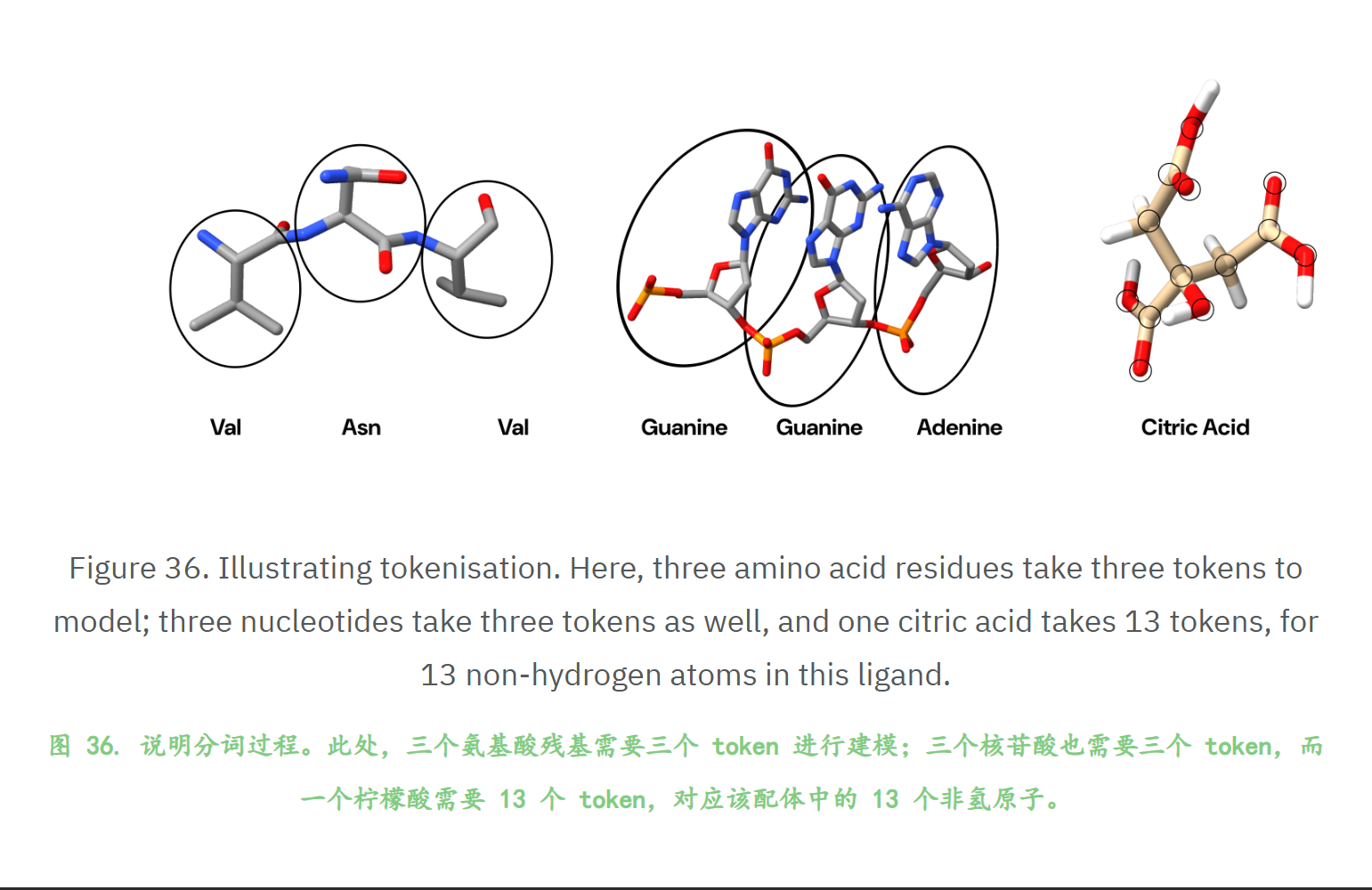
Tokenisation 分词




这里有一个重点需要强调,AlphaFold3中PAE图是针对token进行计算,而不是2中针对氨基酸参加进行。
Diffusion 扩散


AlphaFold 3 输出概述
网页服务器给出的5个model,其实就是5次采样扩散过程的结果;

另外指标这一方面:
pLDDT依旧是对于局部(原子)位置预测的置信指标,PAE是相对位置预测置信,
pTM依旧是对整体,ipTM是复合物角度的PAE,
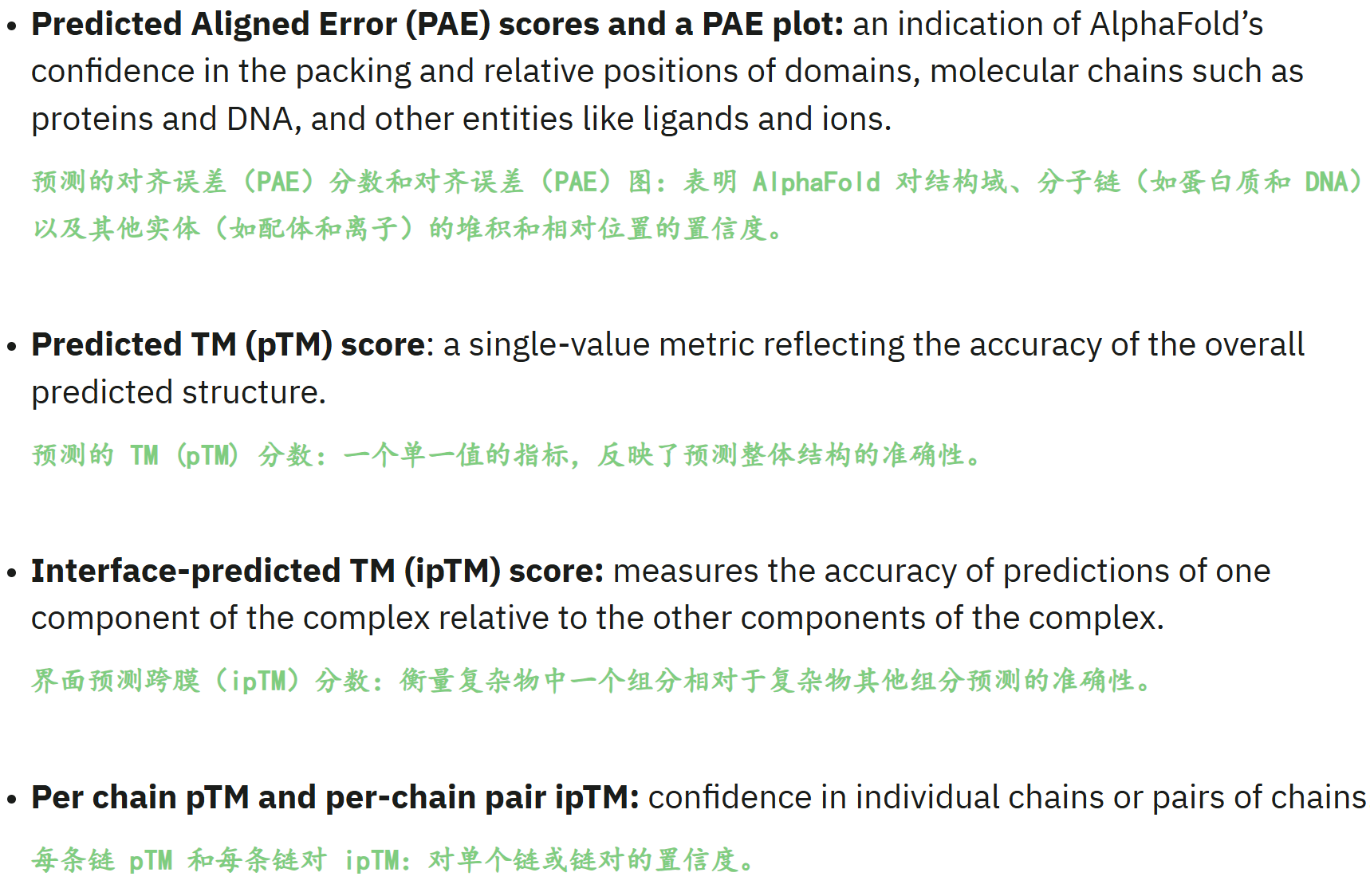
**再次强项AlphaFold3和2的区别:
**AlphaFold 3 预测复杂结构中单个原子的坐标,这与 AlphaFold 2 不同,AlphaFold 2 预测的是氨基酸残基及其侧链的位置。
3,AlphaFold 3 面临的问题
PDB中静态结构,也就是缺少结构变异性,也就是无法捕捉多构象集合性质;
这一点其实我在前一篇博客中提到了,问题本质其实就是机器学习的通病,学到的是训练集的分布规律,多多少少对训练集有bias。


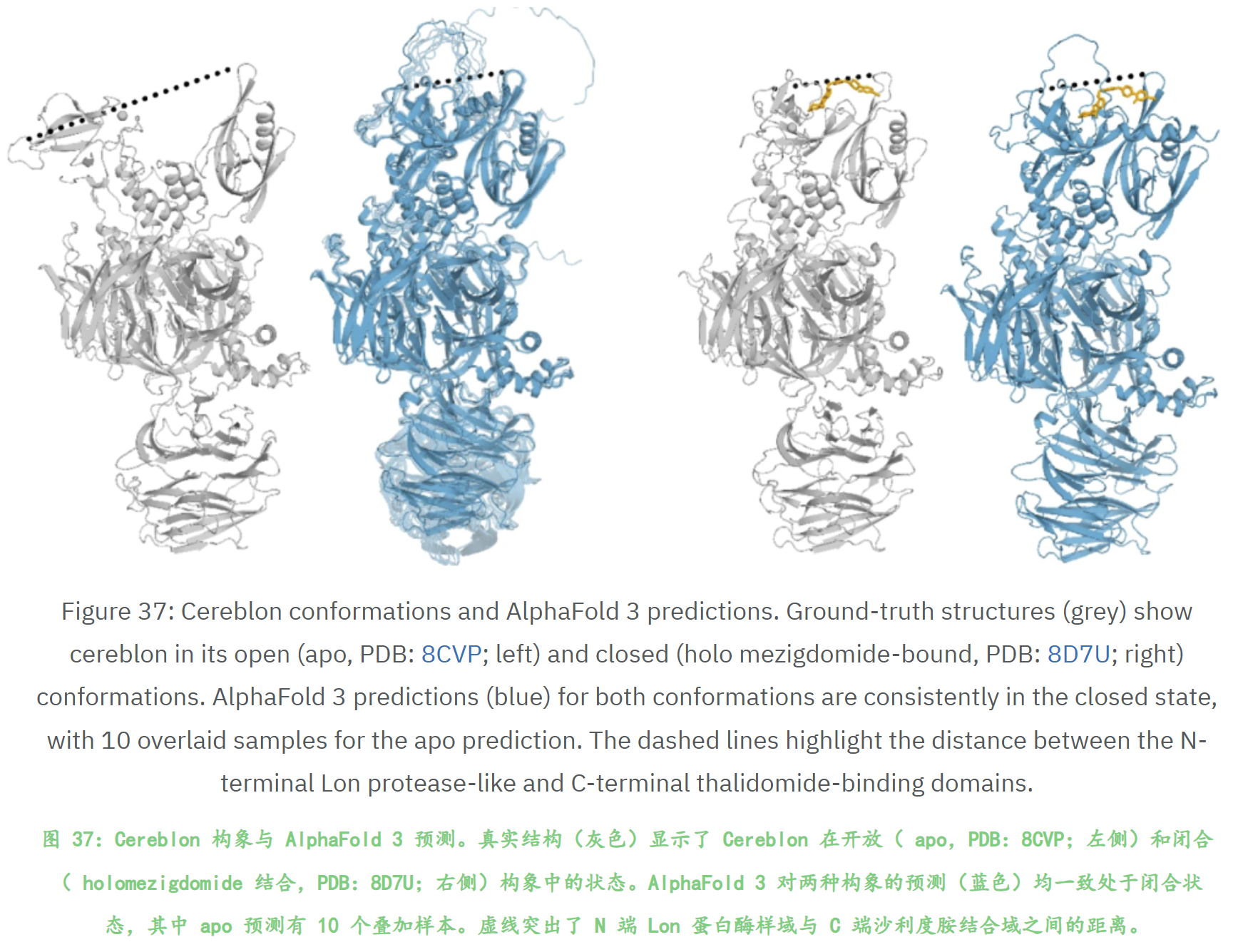

官网给出的一个非常典型的例子:
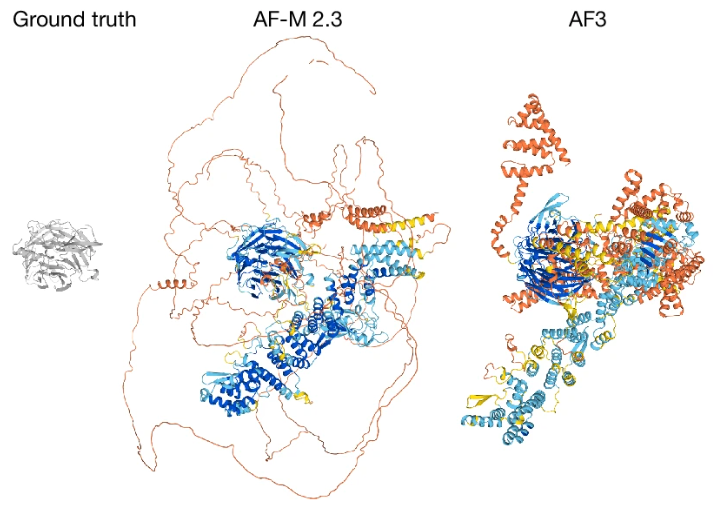
model的幻觉,我们依然是能够从pLDDT置信度的指标上看出来的,
但是预测的结构,其实是难以预估的。


4,我应该使用 AlphaFold 2 还是 AlphaFold 3?


这其实是一个非常重要的问题:
对于复合物的建模,需要考虑非聚合物环境也就是上下文context的影响,
尤其是我们在正常建模特定配以有无时的聚合物结构时,很容易一不小心就将必要的稳定化配体当做case-control都去掉。
这里举一个例子------在使用AlphaFold 3预测锌指蛋白与DNA转录因子结合序列的互作结构时,锌离子作为非聚合物配体,其加入或移除会对聚合物的置信度评分产生显著影响:
- AlphaFold 3的工作原理与置信度评分机制:AlphaFold 3的置信度评分是针对标记计算的,这使其能够预测包含多种分子类型的异质复合物。其置信度指标包括pLDDT、PTM、iPTM、成对iPTM和亚基间接触的PAE等,这些指标反映了预测结构的可靠性和相互作用的置信度。
- 锌离子对蛋白质-DNA互作结构的影响:锌指蛋白的结构和功能依赖于锌离子的存在,锌离子与锌指蛋白中的特定氨基酸残基结合,稳定其三维结构,进而影响锌指蛋白与DNA的结合模式和亲和力。如果在预测时不加入锌离子,AlphaFold 3可能无法准确捕捉到锌指蛋白的正确构象,以及其与DNA相互作用的精确方式,从而导致置信度评分降低。
- 非聚合物环境对置信度评分的影响:根据AlphaFold 3的特性,非聚合物环境如锌离子等配体的加入或移除,会改变复合物的整体能量状态和分子间相互作用,进而影响模型对结构预测的置信度。在锌指蛋白与DNA互作的例子中,锌离子作为稳定配体,其存在与否会影响蛋白质-DNA界面的结构稳定性和相互作用强度,AlphaFold 3会根据这些变化调整置信度评分。
- 与AlphaFold 2的比较:AlphaFold 2主要针对蛋白质结构预测,不考虑非聚合物环境的影响,因此在预测蛋白质-DNA互作时,无需考虑锌离子等配体的加入或移除问题。但这也可能导致其在预测包含非聚合物成分的复合物结构时,结构准确性略低于AlphaFold 3,不过避免了因非聚合物环境因素带来的置信度评分复杂性。
总的来说:
AlphaFold 3对聚合物(如蛋白质)的置信度评分,会受"非聚合物环境"(如离子、稳定配体等)的显著影响------加入或移除这类物质,可能大幅改变评分结果。
若要在"仅含聚合物"的场景(如研究蛋白质-蛋白质相互作用)中使用它,需根据情况适当添加非聚合物环境,才能让置信度评分更可靠。AlphaFold 2无需考虑"非聚合物环境"的影响,能避开上述操作复杂性,但代价可能是其预测的结构准确度会略低于AlphaFold 3。
5,AlphaFold 3 的预测结果是如何得到验证的?

替代方案的出现
HelixFold-3,参考:https://github.com/PaddlePaddle/PaddleHelix

Chai-1:参考https://github.com/chaidiscovery/chai-lab

Boltz-1 and Boltz-2:参考https://github.com/jwohlwend/boltz
 Protenix:参考https://github.com/bytedance/Protenix
Protenix:参考https://github.com/bytedance/Protenix

AlphaFold 3 的独立基准测试


当然,也有一些阴性结果报道:



一个核心优势,就是静态相互作用预测表现突出。
AF3 在预测 "静态蛋白质 - 配体相互作用" 时表现优异 ------ 这类相互作用的特点是,蛋白质与配体结合后,自身构象变化极小(蛋白质与未结合配体时的 "去蛋白状态" 相比,RMSD 值<0.5Å,RMSD 是衡量分子结构相似性的关键指标,数值越小表示构象差异越小)。
在该场景下,AF3 显著优于传统对接方法,尤其在 "侧链取向准确性" 上优势明显,还能作为 "真阳性二元相互作用建模工具",为经实验验证的结合对生成可靠的结构模型,这对药物研发中确认有效结合关系有实际价值。
不过这种类似于刚性对接的静态互作场景毕竟是比较少见的,谁也不能够保证,尤其直觉或者常理来说如果一个蛋白质要和一个配体进行互作,其构象应该会发生比较大的变化,比如说酶的活性位点附近的构象,应该会发生比较大的构象变化。


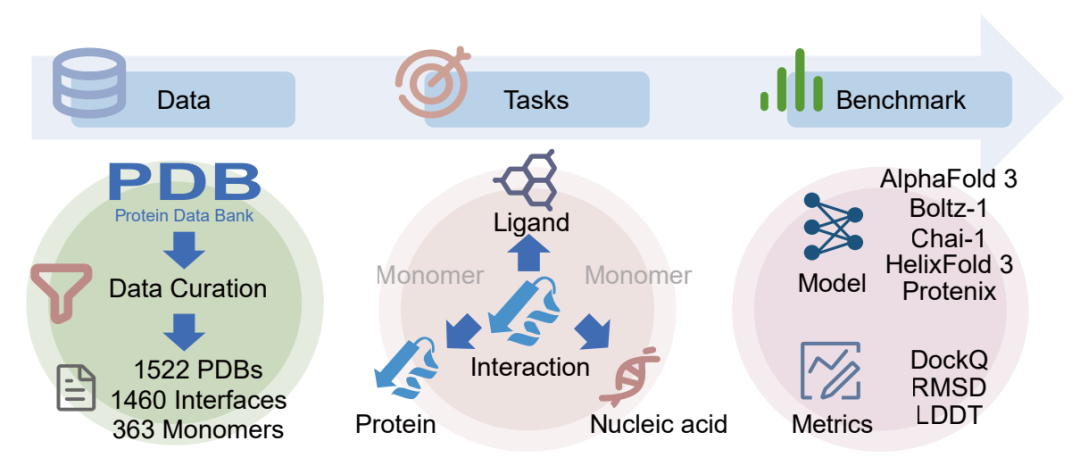
一个基准测试,有点意思,参考:https://github.com/BEAM-Labs/FoldBench
名为 FoldBench 的系统性评测上线,全面评估了当前全原子预测模型在生物分子结构预测中的表现。该研究由复旦大学、上海交通大学、香港中文大学等机构的科研人员联合完成,评估对象包括DeepMind最新发布的 AlphaFold 3 以及其他四款全原子预测模型。

新一代预测工具


主要是ABCFold和AlphaBridge。
6,AlphaFold 服务器:通往 AlphaFold 3 的入口

AlphaFold Server 能做什么?

我们可以使用 AlphaFold Server 对由以下一种或多种生物分子组成的结构进行建模:




AlphaFold Server 不能做什么?

关于配额,以及任务提交的问题,

7,使用 AlphaFold Server 生成预测结果的逐步指南
这一小节其实就很直白了,基本上就是网页端的操作简单指南:
官网也给出了一个教程,具体视频地址在油管上:
https://youtu.be/9ufplEgtq8w
具体的输入操作非常简单:


添加复合物
其实就是多聚物中单体的拷贝数,也就是副本数目

添加修饰

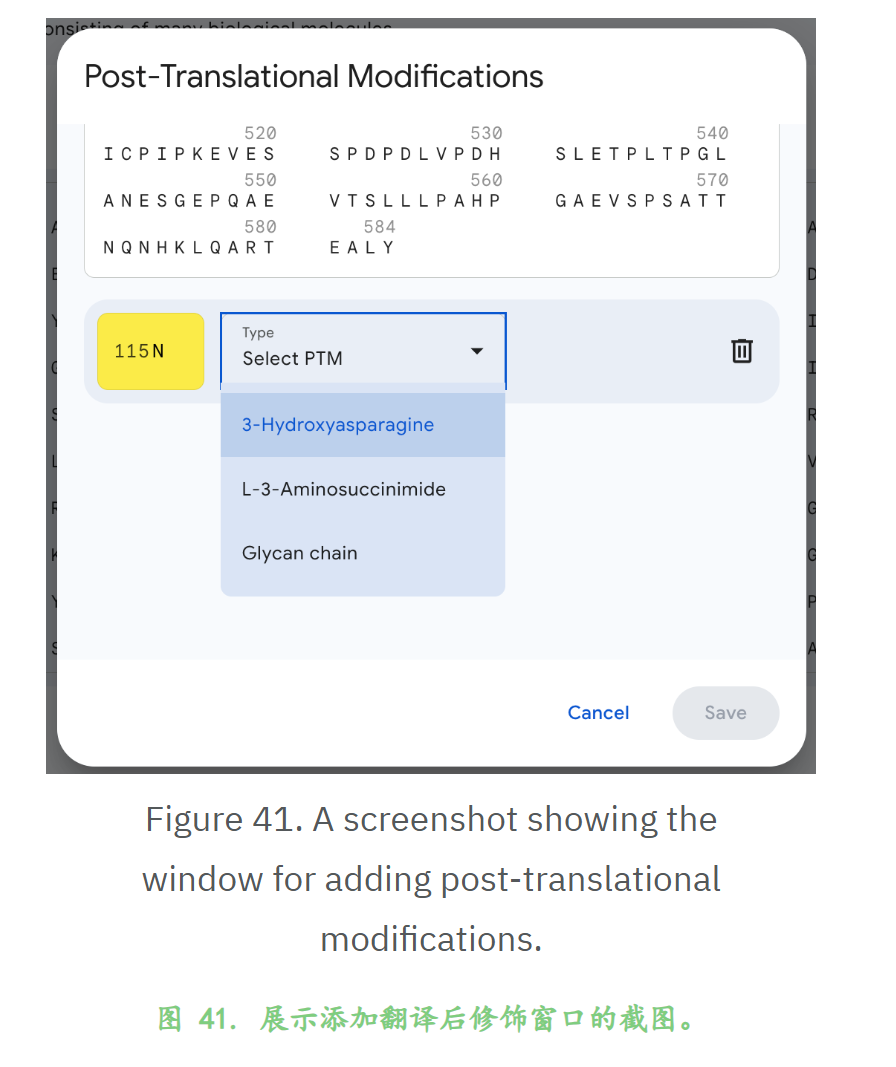
翻译后最是非常简单,网页端只需要在添加实体entity也就是蛋白质序列链中点击所需要修饰的氨基酸残基即可,
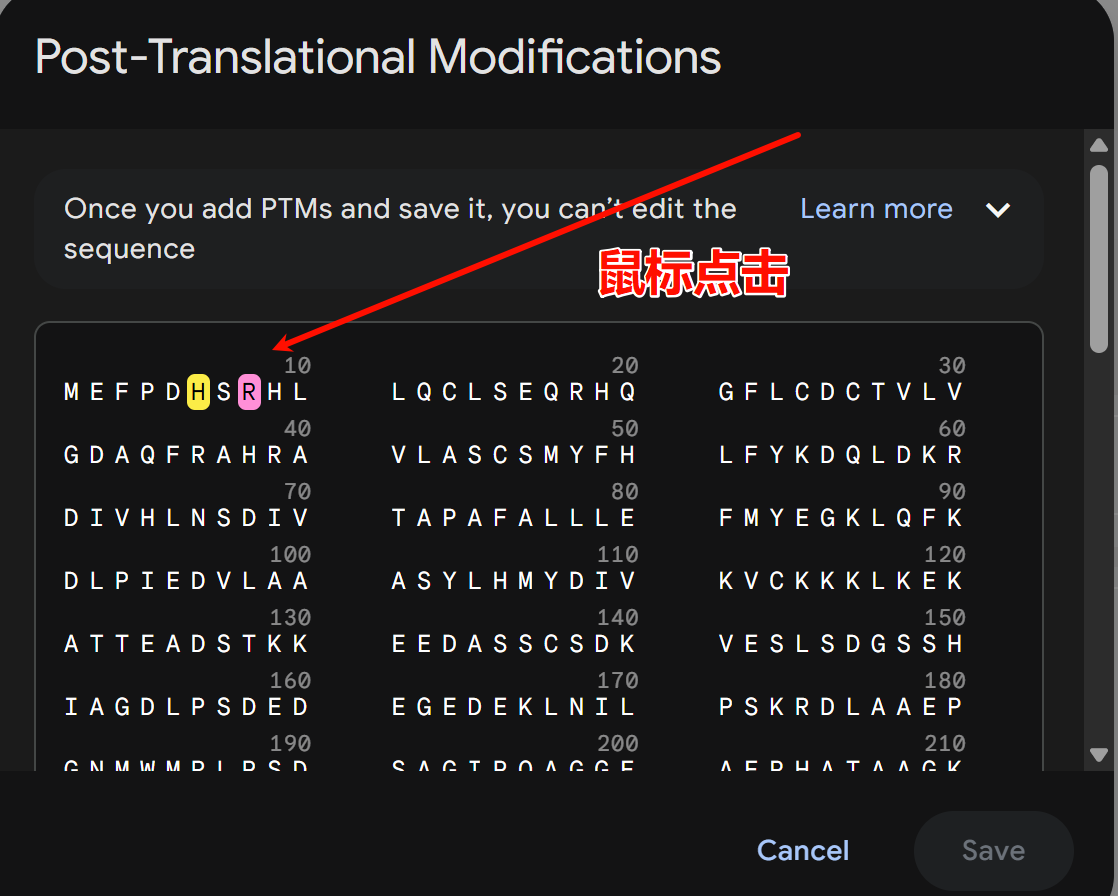
然后再选对应类型,
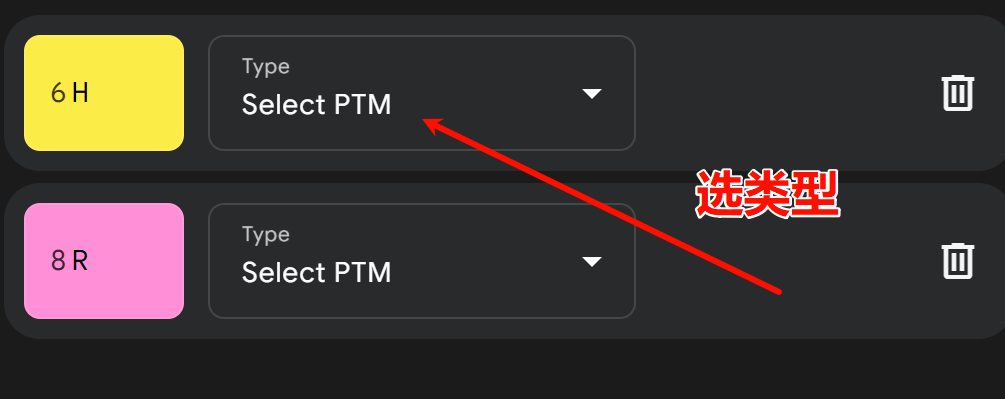
基本上就是选残基(position)+选类型(type)这两步,
如果是json设置的话,就是一个entity中的两个键值对,具体可以参考我之前批量提交网页服务的博客:
https://blog.csdn.net/weixin_62528784/article/details/154954399?spm=1001.2014.3001.5502
对于核酸其实同理,但是我们知道像DNA这样的双链结构,其实修饰是有区别的,
所以一般的修饰还是要看你是在哪一条链上的。

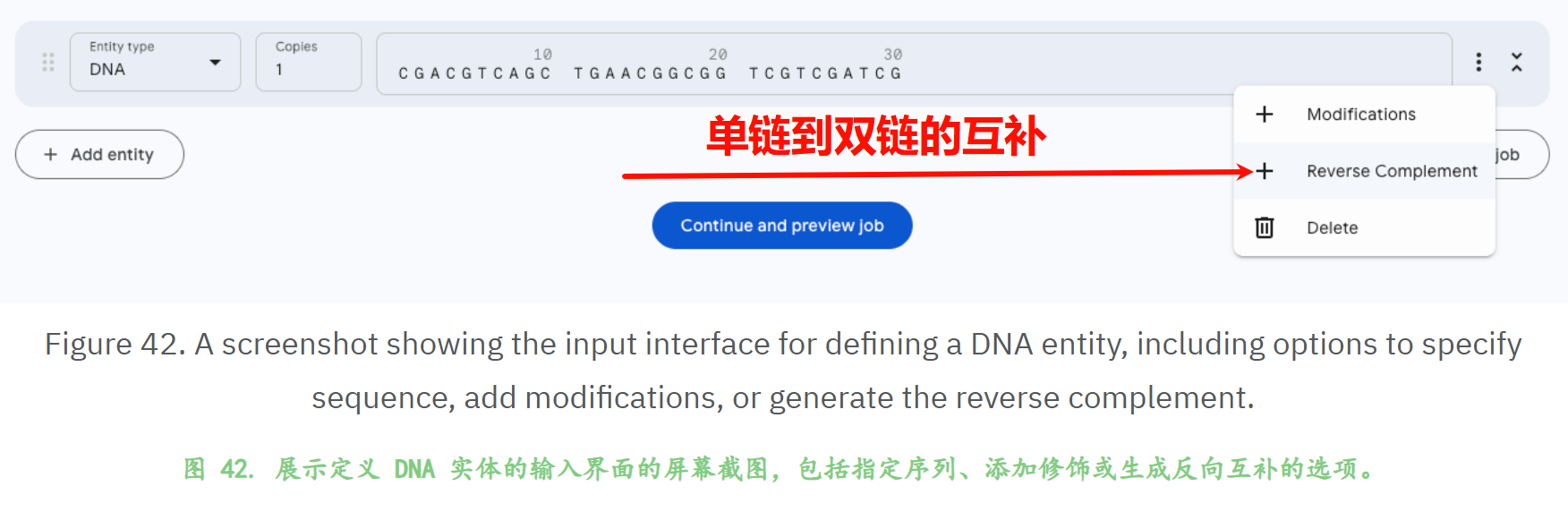
建模顺序和排列顺序之间,实际的排列顺序影响不大:


任务提交

结果




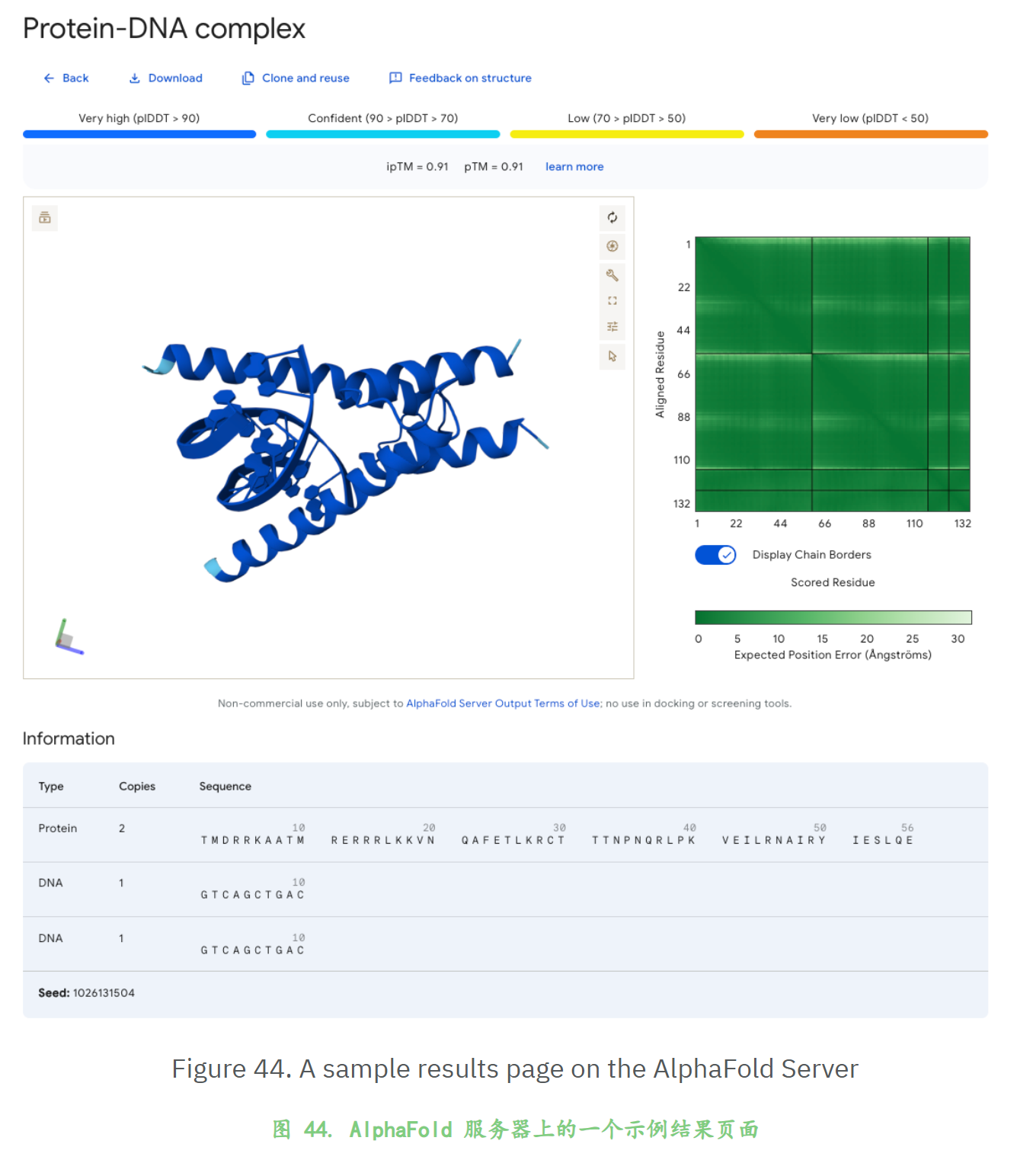

8,AlphaFold Server 的高级功能

JSON 任务提交
这个其实我之前的博客就提到过了,具体参考:https://blog.csdn.net/weixin_62528784/article/details/154954399?spm=1001.2014.3001.5502


采样多个种子
这一点也很重要,采样嘛,基本上就是奔着结构预测效果提升,或者是看更多的构象去的,
官方是建议:采用起码20个左右的种子(也就是一个任务每次采用不同的种子,起码运行20次),这样的采样潜在空间基本上够用,然后再依据前面提到过的各种评估置信指标值,对预测结果进行评估,主要是排序。
当然,我们知道每一个seed种子的任务,其实是有5个model输出的,也就是有5次采样扩散过程的结果。

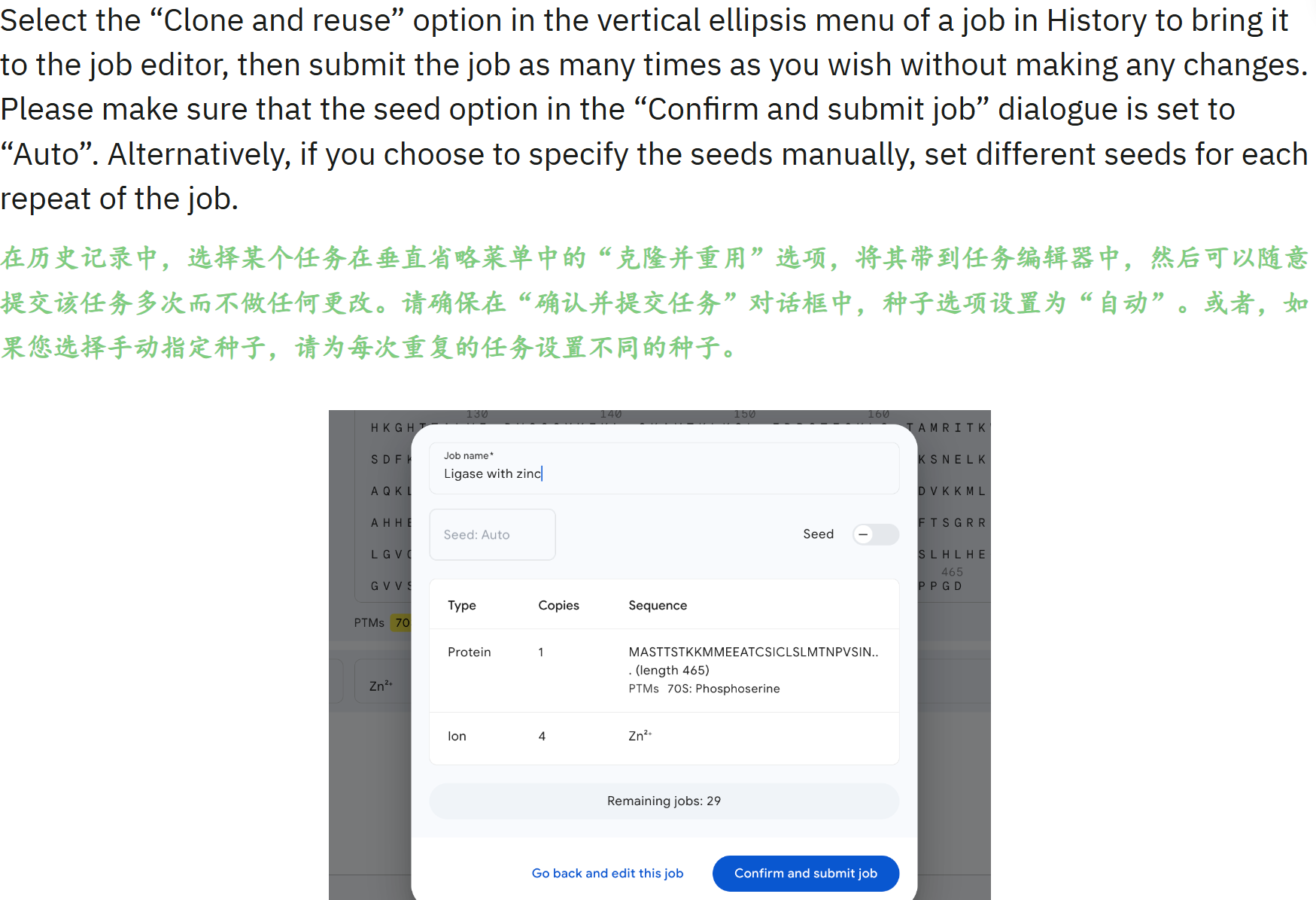

复现任务(seed)
其实就是预测结果的可重复性,以及你最后提交,比如说是文献的时候一个依据凭证,
简单来说:就是保留你每次运行任务的json文件,这个文件很重要,甚至是可以作为文献发表中的附近资料。

9,解读AlphaFold Server输出的结果
这一点其实很重要,因为我看到太多的人,主要是做湿实验、也就是纯生物那一批人,基本山只看最后结构的交互图,要么截一下图示意一下,PAE都很少看;有点意识的会把mmCIF文件下载回去之后在pymol等可视化软件中进查看;至于指标,其实很多人并不是很关注。
但是,这些也仅仅只是表面信息,
真正有用的信息,

AlphaFold Server 提供的输出
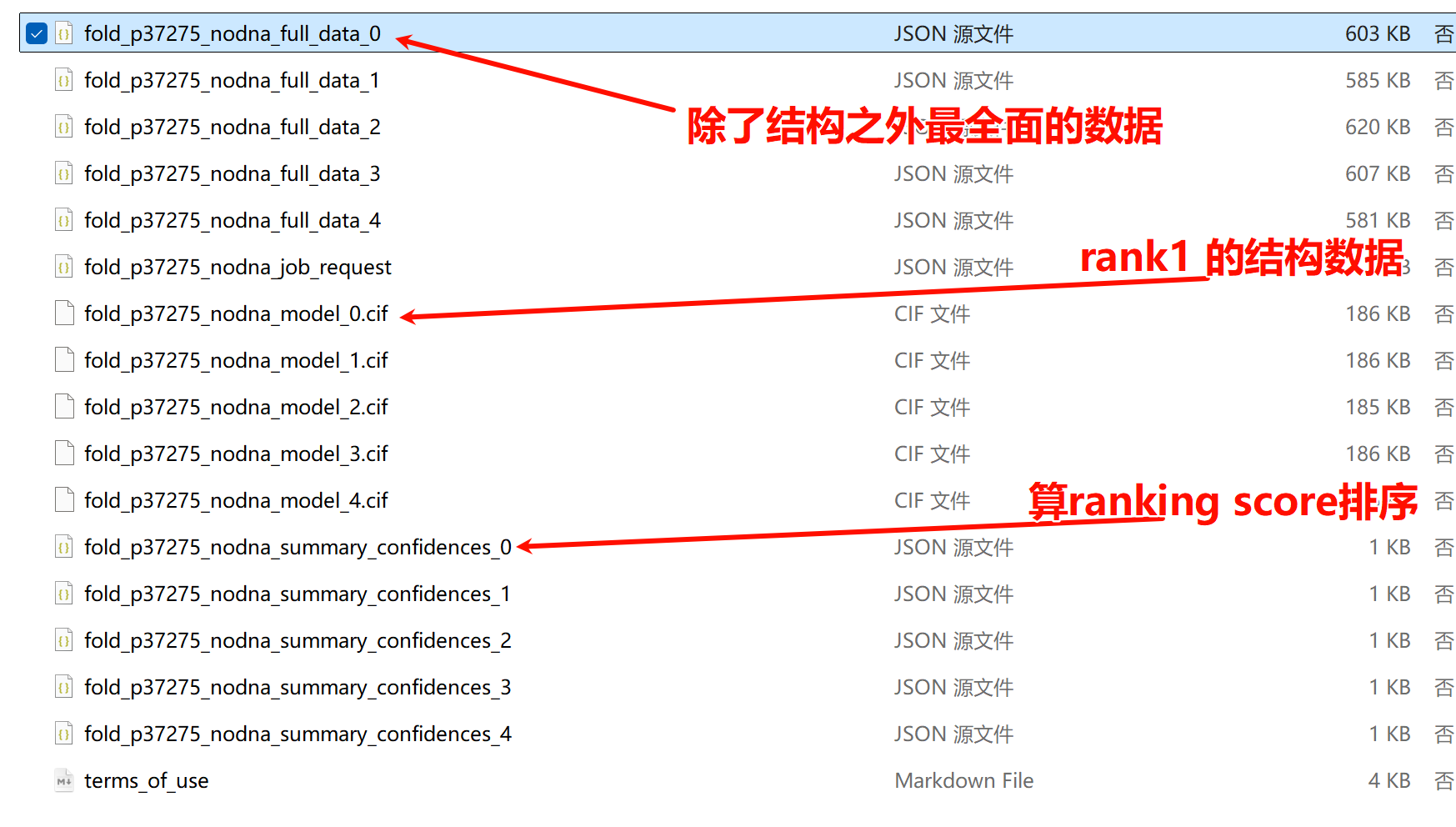
比如说我这里的一个结构预测的job的输出,
大致上就有下面这些文件:
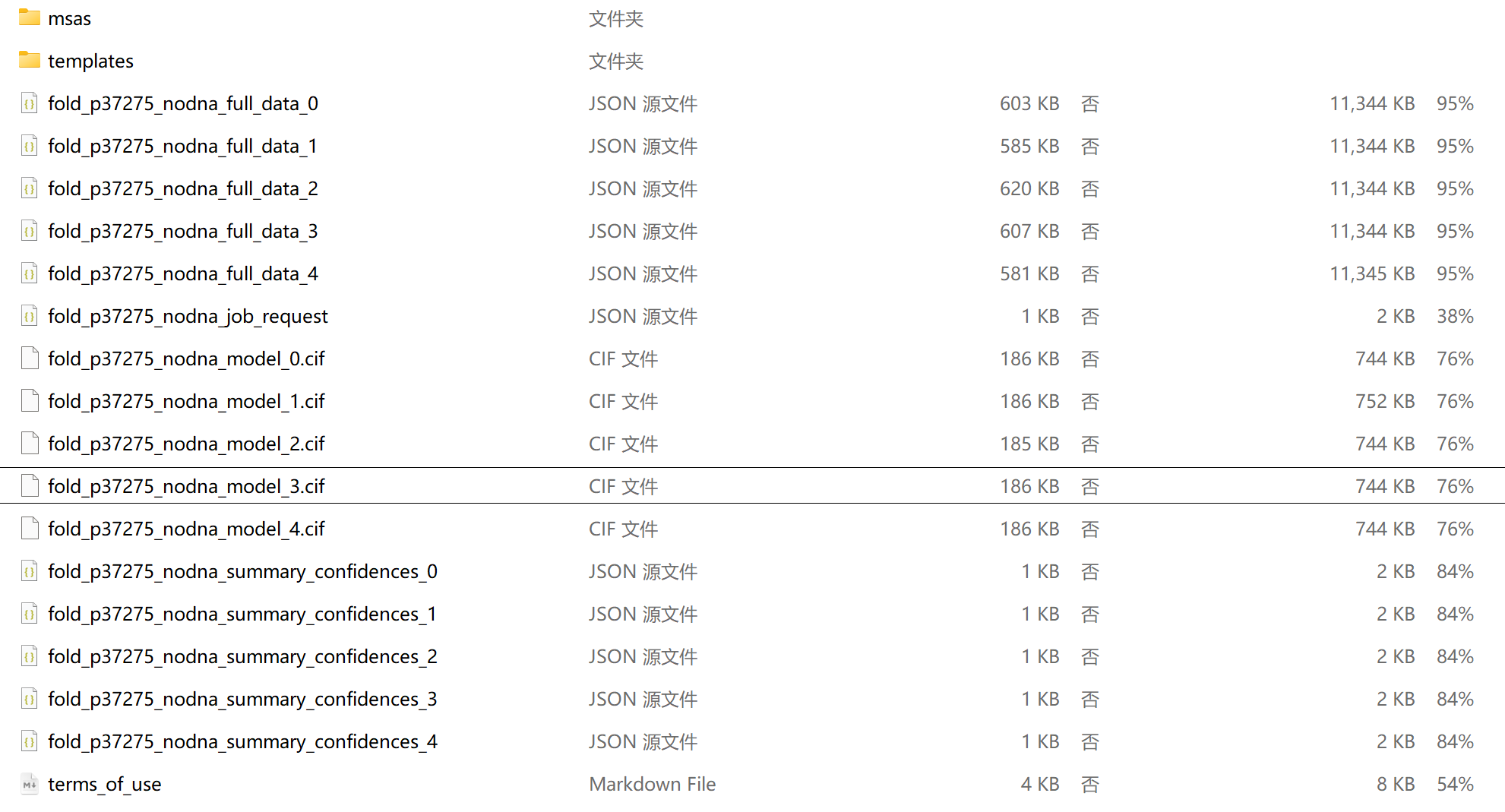
full data数据比较多,文件比较大,其中键值对内容主要参考下图中文件右上角:
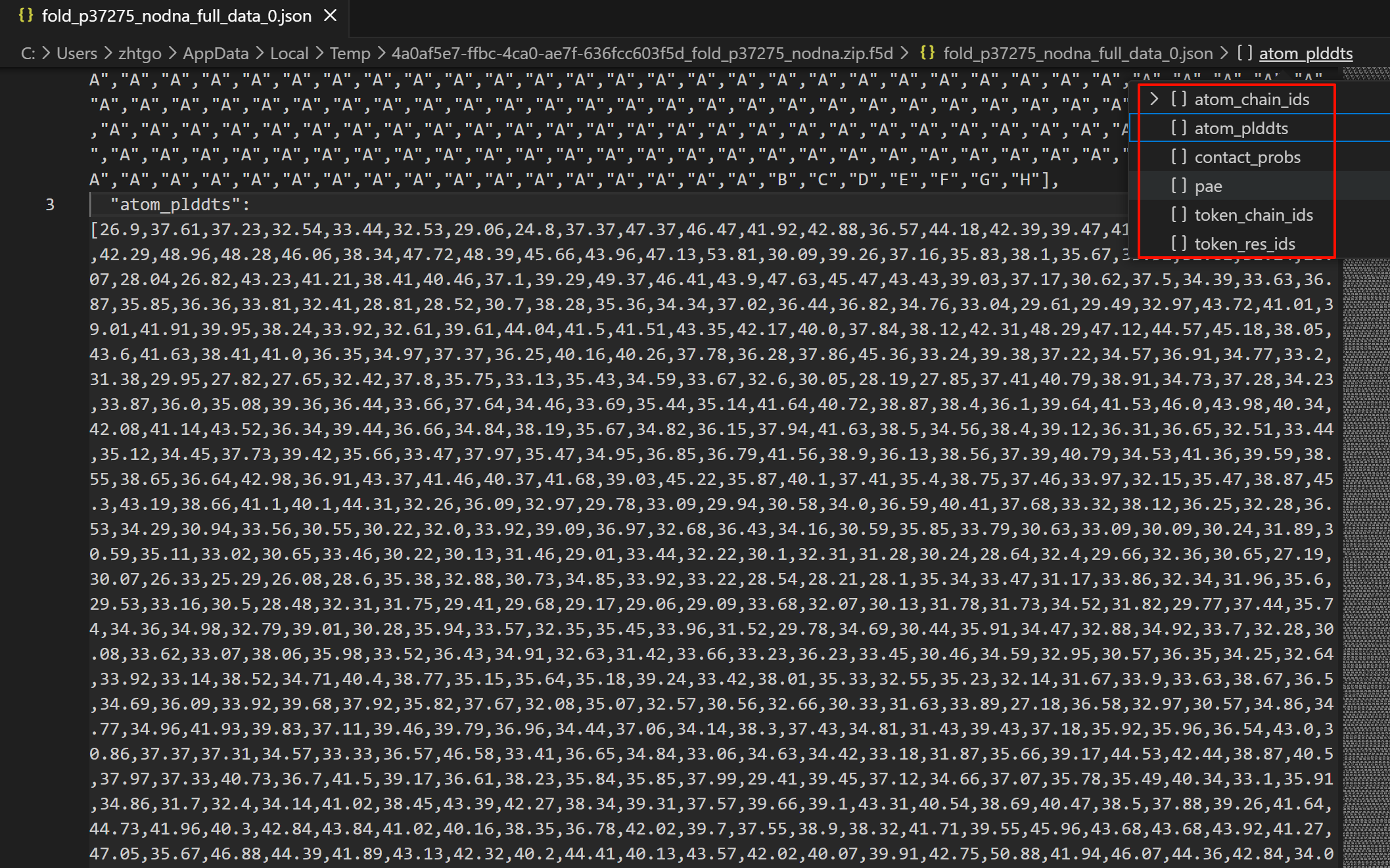
这一部分信息比较杂,


request就没什么好说了,其实就是对任务job的描述:
至于model的cif文件,其实就是预测结果的结构坐标文件
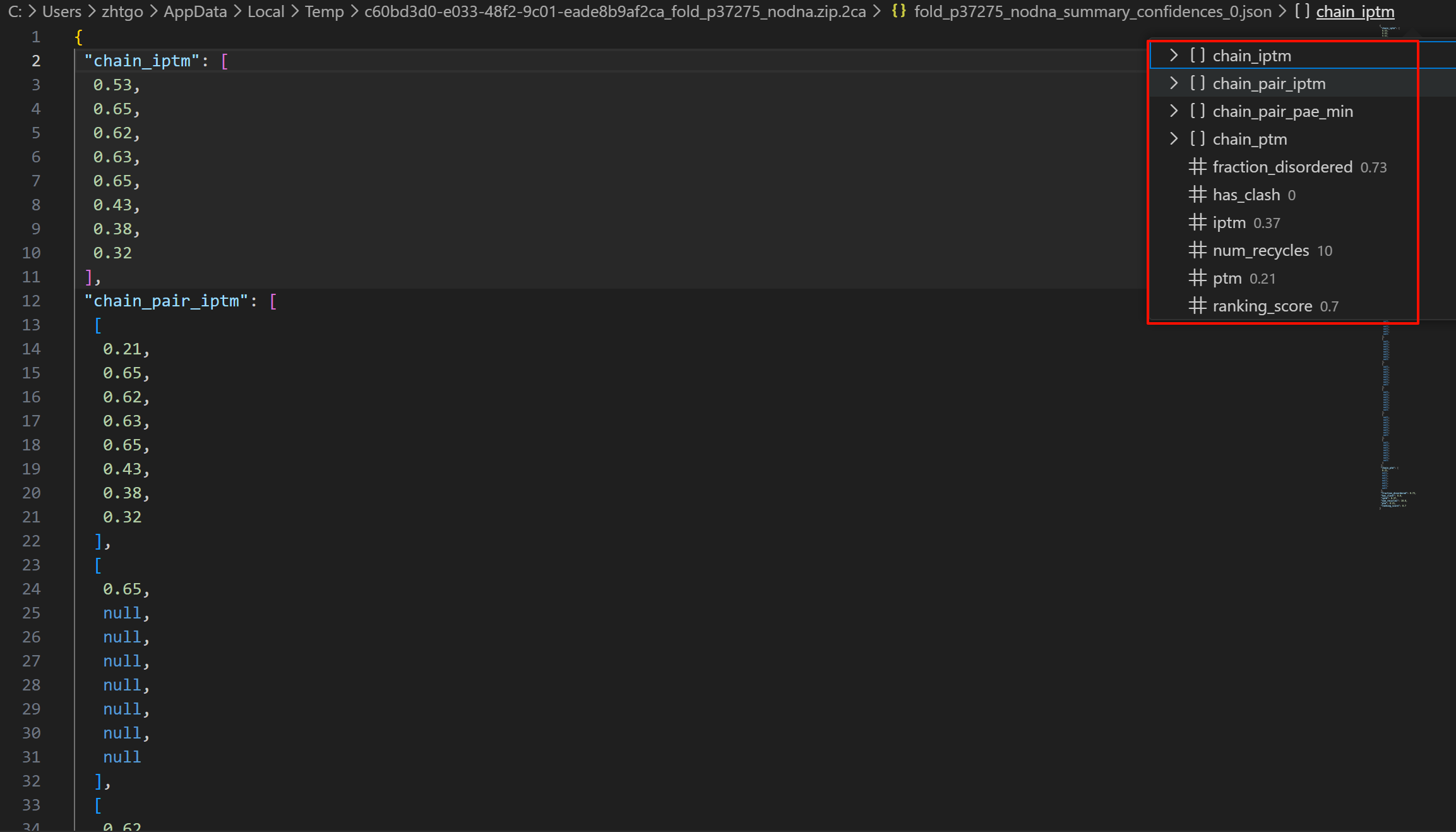
然后就是置信指标的summary文件:
具体有哪些指标,可以看下图右上角的键值对信息
term of use的话就是一堆废话
再次强调,每个种子有5次扩散采样,但是前面提到的种子扩展需要至少20个左右的种子seed;

5个model,有个排名rank,主要是对结构进行的置信排名,从前面我们可以知道,主要是对pTM以及ipTM等指标的综合考虑



下面就是一个fold__summary_confidences_.json文件内容的示例,展示了多个置信度指标。
plain
{
"chain_iptm": [
0.85,
0.86,
0.59,
0.59
],
"chain_pair_iptm": [
[
0.82,
0.9,
0.83,
0.83
],
[
0.9,
0.82,
0.83,
0.84
],
[
0.83,
0.83,
0.03,
0.1
],
[
0.83,
0.84,
0.1,
0.03
]
],
"chain_pair_pae_min": [
[
0.76,
0.79,
1.0,
1.12
],
[
0.79,
0.76,
1.11,
1.0
],
[
0.98,
1.06,
0.78,
0.92
],
[
1.05,
0.97,
0.92,
0.78
]
],
"chain_ptm": [
0.82,
0.82,
0.03,
0.03
],
"fraction_disordered": 0.18,
"has_clash": 0.0,
"iptm": 0.91,
"num_recycles": 10.0,
"ptm": 0.91,
"ranking_score": 1.0
}置信度指标

**下面的指标中,有一个是我个人认为比较重要的,就是chain pair pae min,这个指标能够用来分析chain之间是否互作。 **

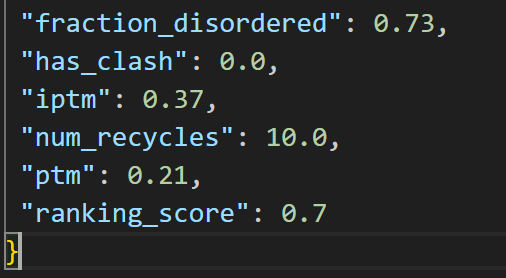

下面这个就是对于5个model的一个rank的依据:就是算这个ranking score

总得来说,对于生成的这5个model,用summary中的指标计算一个ranking score,
我们主要关注下面这3个文件:
其中cif文件是用来结构可视化,full data的json是用来注释+最方便用来在python中进行结构数据分析
这节培训内容其实比较重要,课程链接参考:https://www.ebi.ac.uk/training/online/courses/alphafold/alphafold-3-and-alphafold-server/alphafold-server-your-gateway-to-alphafold-3/interpreting-results-from-alphafold-server/
10,如何评估 AlphaFold 3 预测的质量
指标的对象变了,之前是残基,现在是token;
以及应该关注的点也变了,之前因为预测复合物不是很多,所以更加关注于结构的局部置信度,也就是pLDDT之类的指标;
现在应该更多关注于结构相互作用之间的指标

下面的这个例子我之前讲过了:

AlphaFold 3 中的 pLDDT 分数

再次强调,AlphaFold3中对于除蛋白质序列之外的建模,也有pLDDT分数,
比如说下面的DNA序列
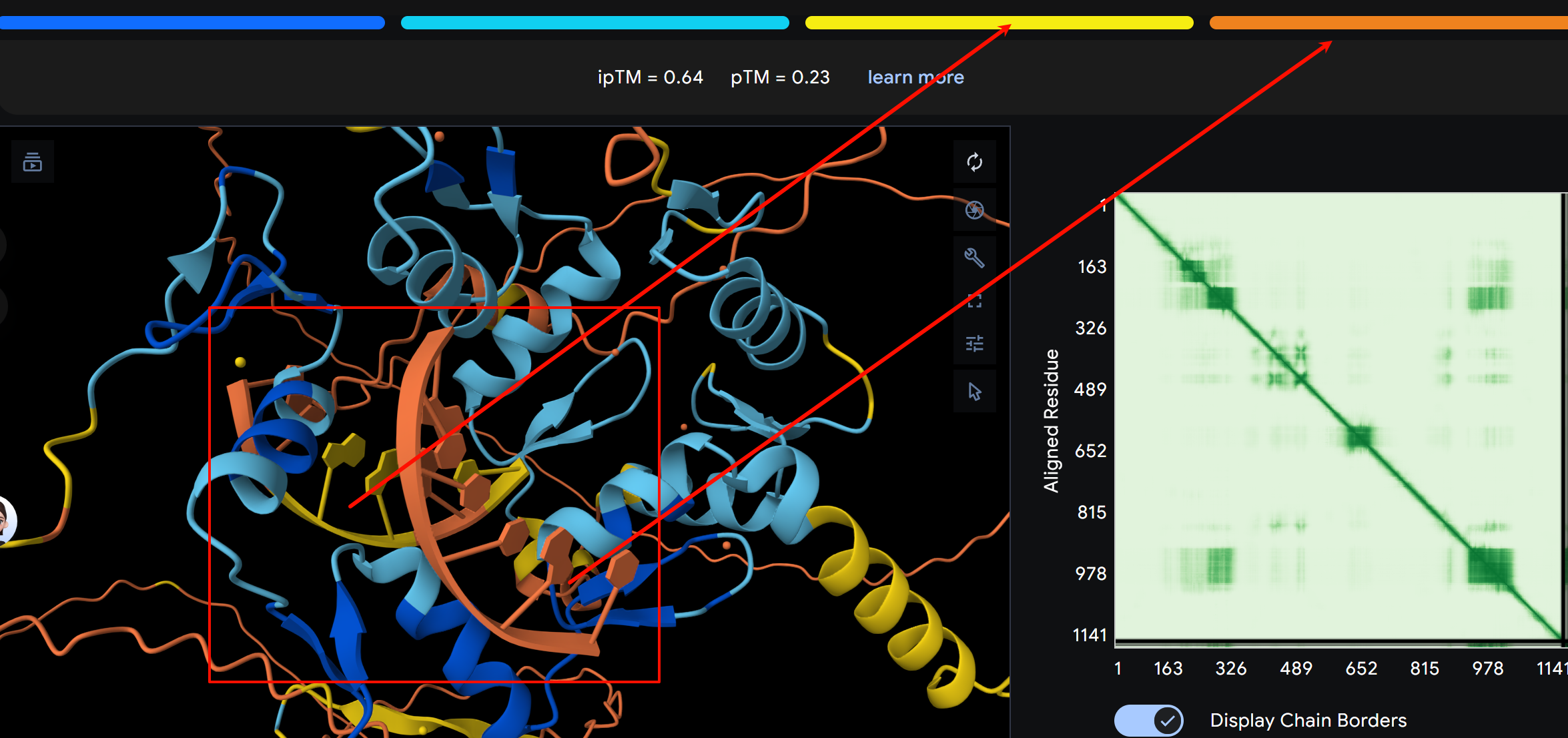

AlphaFold 3 中的 PAE 分数

PAE类似,除了蛋白质序列之外还有。
另外PAE也可以用于互作的某种指标。


pTM 和 ipTM,每链评分和成对评分

iPTM同样可以用来评估相互作用的某种指标。

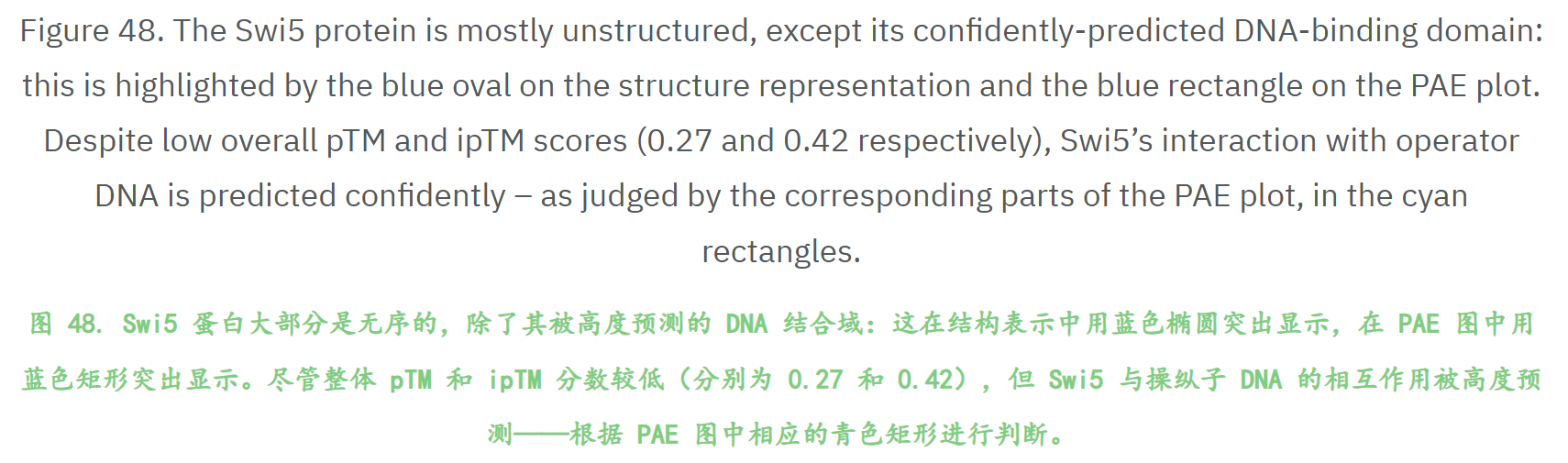
这一节培训内容也同样非常重要,参考:https://www.ebi.ac.uk/training/online/courses/alphafold/alphafold-3-and-alphafold-server/how-to-assess-the-quality-of-alphafold-3-predictions/
11,使用 AlphaFold 3 源代码
省流:A100,或H100


初始结构预测的注意事项


开源社区中已经有人做到将CPU part以及GPU part简单的拆分开来了。
比如说下面的

https://github.com/Zuricho/ParallelFold

AlphaFold 3 输入格式


与 AlphaFold Server JSON 兼容

特殊考虑

12,总结
错义变异效应的预测


13,课程ppt下载
参考:https://www.ebi.ac.uk/training/online/courses/alphafold/course-slides/
二,术语表
参考链接:https://www.ebi.ac.uk/training/online/courses/alphafold/glossary-of-terms/