欢迎关注『youcans论文精读』系列
【youcans论文精读】VM-UNet:面向医学图像分割的视觉Mamba UNet架构
-
- [0. 论文简介](#0. 论文简介)
-
- [0.1 基本信息](#0.1 基本信息)
- [0.2 论文速览](#0.2 论文速览)
- [0.3 摘要](#0.3 摘要)
- [1. 引言](#1. 引言)
- [2. 相关工作](#2. 相关工作)
-
- [2.1 基于CNN的模型](#2.1 基于CNN的模型)
- [2.2 基于Transformer的模型](#2.2 基于Transformer的模型)
- [2.3 基于SSM的模型](#2.3 基于SSM的模型)
- [3. 方法](#3. 方法)
-
- [3.1 预备知识](#3.1 预备知识)
- [3.2 视觉 Mamba UNet(VM-UNet)](#3.2 视觉 Mamba UNet(VM-UNet))
- [3.3 VSS模块](#3.3 VSS模块)
- [3.4 损失函数](#3.4 损失函数)
- [4. 实验](#4. 实验)
-
- [5.1 数据集](#5.1 数据集)
- [5.2 实现细节](#5.2 实现细节)
- [5.3 与先进模型对比](#5.3 与先进模型对比)
- [5.4 消融实验](#5.4 消融实验)
- [5.5 可视化分析](#5.5 可视化分析)
- [5.6 讨论](#5.6 讨论)
- [6. 结论](#6. 结论)
- [7. GitHub 项目介绍](#7. GitHub 项目介绍)
-
- [7.1 环境配置](#7.1 环境配置)
- [7.2 使用步骤](#7.2 使用步骤)
- [8. 参考文献](#8. 参考文献)
0. 论文简介
0.1 基本信息
2024年 Jiacheng Ruan 等 在 arXiv 发布论文 【VM-UNet:面向医学图像分割的视觉Mamba UNet架构】(VM-UNet: Vision Mamba UNet for Medical Image Segmentation)。
本文提出首个纯基于状态空间模型(SSMs)的医学图像分割模型 VM-UNet,该模型以 VMamba 中的视觉状态空间(VSS)块为基础构建非对称编解码器结构,在保证线性计算复杂度以解决 Transformer 二次复杂度问题的同时,增强长距离依赖建模能力以弥补 CNN 局限。
论文标题: VM-UNet: Vision Mamba UNet for Medical Image Segmentation
作者: Jiacheng Ruan, Jincheng Li, and Suncheng Xiang
论文地址: acm, arxiv
代码仓库: github
引用格式: Ruan, J., Xiang, S.: VM-UNET: vision mamba UNet for medical image segmentation. arXiv preprint arXiv:2402.02491 (2024).

0.2 论文速览
研究背景
自动医学图像分割技术可辅助医生快速病理诊断,提升患者护理效率,现有主流模型分为 CNN-based 和 Transformer-based 两类,但均存在固有局限。
- CNN-based(如 UNet、UNet++):局部感受野限制,长距离建模能力弱。特征提取不充分,分割结果欠佳。
- Transformer-based(如 TransUnet、Swin-UNet):自注意力机制导致二次计算复杂度(与图像尺寸相关)。计算负担重,尤其不适用于医学图像分割这类密集预测任务。
- 以 Mamba 为代表的现代状态空间模型(SSMs),既擅长长距离交互建模,又保持线性计算复杂度,已在语言理解、通用视觉领域验证有效性。
现有 SSM 相关医学分割模型:U-Mamba、SegMamba 均为 SSM-CNN 混合模型,纯 SSM-based 医学图像分割模型尚未被探索,存在研究空白。
VM-UNet 模型设计:
VM-UNet 采用 U 型结构,核心组件包括 Patch Embedding 层、编码器、解码器、Final Projection 层及跳跃连接,整体为非对称设计(减少卷积层以降低计算成本)
- Patch Embedding 层:将输入图像(H×W×3)分割为 4×4 非重叠补丁,映射通道数为 C(默认 96)
- 编码器 4 个阶段,每阶段含 VSS 块 + 前 3 阶段末尾的 Patch Merging(下采样,减半尺寸、翻倍通道)
- 解码器 4 个阶段,每阶段含 VSS 块 + 后 3 阶段开头的 Patch Expanding(上采样,翻倍尺寸、减半通道)
- Final Projection 层 4 倍上采样(Patch Expanding)恢复尺寸,投影层恢复通道数,匹配分割目标
- 跳跃连接 采用简单加法操作,无额外参数,突出纯 SSM 模型的分割性能
主要贡献
- 提出VM-UNet,首次探索纯 SSM-based 模型在医学图像分割中的应用;
- 在 ISIC17、ISIC18、Synapse 三大数据集上验证 VM-UNet 的竞争力;
- 为纯 SSM-based 医学图像分割模型建立基准,为后续高效 SSM-based 方法提供思路。
0.3 摘要
在医学图像分割领域,基于CNN与基于Transformer的模型已得到广泛研究。然而,CNN在长程建模能力上存在局限,而Transformer则受限于二次计算复杂度。近期,以Mamba为代表的状态空间模型展现出巨大潜力,其不仅能有效建模长程依赖关系,还保持了线性计算复杂度。
本文基于状态空间模型,提出了一种用于医学图像分割的U型架构模型------视觉Mamba UNet(VM-UNet)。具体而言,我们引入视觉状态空间模块作为基础模块以捕获广泛上下文信息,并构建了包含少量卷积层的不对称编码器-解码器结构以降低计算成本。
我们在ISIC17、ISIC18和Synapse数据集上进行了全面实验,结果表明VM-UNet在医学图像分割任务中具有显著竞争力。据我们所知,这是首个基于纯状态空间模型构建的医学图像分割模型。我们期望通过此项研究奠定基线,并为未来开发更高效、更强大的基于状态空间模型的分割系统提供有价值的研究见解。
代码已开源:https://github.com/JCruan519/VM-UNet。
1. 引言
自动化医学图像分割技术能够辅助医生加速病理诊断,从而提升诊疗效率。近年来,基于CNN与基于Transformer的模型在多种视觉任务中展现出卓越性能,尤其在医学图像分割领域。以CNN模型为代表的UNet[1]以其结构简洁、扩展性强著称,后续诸多改进均基于该U型架构[2-8]。而基于Transformer的模型先驱TransUnet[9]首次在编码阶段采用视觉Transformer进行特征提取,并在解码阶段结合CNN,展现出强大的全局信息获取能力。随后TransFuse[11]通过并行架构融合ViT与CNN,同步捕获局部与全局特征。Swin-UNet[12]则将Swin Transformer与U型架构结合,首次提出纯Transformer的U型模型,其详细发展脉络如图1所示。
然而,基于CNN与Transformer的模型均存在固有局限[14]。CNN模型受局部感受野限制,难以有效捕获长程信息,常导致特征提取不充分进而影响分割效果。Transformer模型虽在全局建模方面表现优异,但其自注意力机制需消耗与图像尺寸平方成正比的计算量[15,10,16],对于医学图像分割这类密集预测任务尤为显著。现有模型的这些缺陷促使我们探索新型架构,以兼顾长程依赖建模与线性计算复杂度的优势。
近期,状态空间模型(State Space Models,SSM)引起研究界广泛关注。在经典SSM[17]研究基础上,现代SSM(如Mamba[18])不仅能建立长程依赖,还保持输入尺寸的线性计算复杂度。基于SSM的模型已在语言理解[19,18]、通用视觉[20,21]等领域取得丰硕成果。特别地,U-Mamba[22]首次提出SSM-CNN混合模型并应用于医学图像分割;SegMamba[23]则在编码器嵌入SSM,解码器仍采用CNN,构建了针对3D脑肿瘤分割的混合架构。尽管这些研究已尝试将SSM用于医学图像分割,但纯SSM架构的潜力尚未得到充分探索。
受VMamba[21]在图像分类任务中成功的启发,本文首次提出视觉Mamba UNet------一种纯SSM架构的医学图像分割模型。
具体而言,VM-UNet包含编码器、解码器和跳跃连接三部分:编码器由VMamba的VSS模块(视觉状态空间)构成,通过补丁合并实现下采样;解码器则通过VSS模块与补丁扩展操作恢复分割图尺寸;跳跃连接采用最基础的加法融合,以凸显纯SSM模型的原生性能。
通过在器官分割与皮肤病变分割任务上的系统实验,我们验证了纯SSM模型在医学图像分割中的潜力。在Synapse[24]、ISIC17[25]和ISIC18[26]数据集上的实验表明,VM-UNet可获得具有竞争力的性能。需特别说明的是,该模型未引入任何特殊设计模块,体现了最基础的纯SSM分割架构。
本文主要贡献如下:
- 提出首个纯SSM医学图像分割模型VM-UNet,探索了该架构在分割任务中的应用潜力;
- 在三个数据集上完成系统实验,证明模型具备显著竞争力;
- 为医学图像分割任务建立了纯SSM基准模型,为开发更高效、更强大的SSM方法提供重要参考。
本文后续结构如下:第二章回顾基于CNN、Transformer及SSM的医学图像分割方法;第三章详细阐述VM-UNet的架构设计与学习流程;第四章通过对比实验与综合分析验证模型性能;第五章总结研究并展望未来方向。
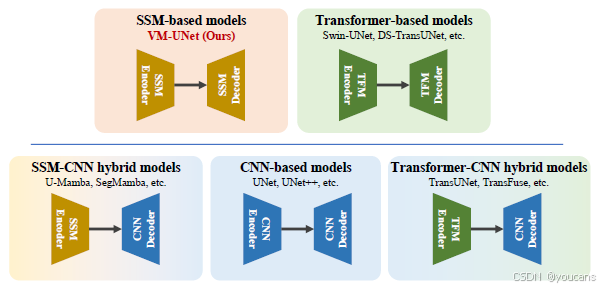
图 1:医学图像分割领域U型架构发展的简要介绍。
2. 相关工作
本节将简要回顾常见医学图像分割方法的相关研究工作。这些现有方法的核心思想在于高效建模上下文信息,可大致划分为基于CNN的方法、基于Transformer的方法以及基于SSM的方法。
2.1 基于CNN的模型
传统医学图像分割方法通常基于机器学习技术[27,28]。随着卷积神经网络的发展,UNet[1]作为端到端分割模型在医学图像分割中取得了显著成效。得益于UNet结构简洁、扩展性强且分割能力优异,后续许多研究均基于该U型架构进行改进。例如,UNet++[3]通过稠密连接替换原始跳跃连接,以增强特征表征并弥补采样造成的信息损失;Attention-UNet[29]引入注意力门控机制,通过权重分配使模型更聚焦目标区域;MALUNet[4]设计了四种高效注意力模块,在显著降低参数量与计算负载的同时实现了更优的分割性能。需要指出的是,理解全局图像信息对医学图像分割至关重要,但CNN模型固有的局部感知特性使其难以建立长程依赖关系。
2.2 基于Transformer的模型
受ViT[10]在通用视觉领域成功的启发,众多研究将Transformer模型引入医学图像分割任务以增强长程依赖建模能力。例如TransFuse[11]采用CNN与ViT并行的编码器结构,同步处理局部与全局特征;Swin-UNet[12]将Swin Transformer[13]与UNet架构融合,首次构建了纯Transformer的医学图像分割模型;DS-TransUNet[30]通过输入多尺度图像块并采用双路Swin Transformer编码,以捕获多尺度特征信息;MEW-UNet[6]则在Transformer模块中引入频域操作,提出多轴外部权重模块以获取更丰富的频域特征。然而,基于Transformer的模型需要承受与输入尺寸平方成正比的计算复杂度,导致较高的计算负担。
2.3 基于SSM的模型
源自控制理论线性状态空间方程的 状态空间模型(State Space Models,SSM),如 HiPPO[31]、LSSL[32] 等,主要应用于序列数据建模任务。文献[19]提出的结构化状态空间序列模型(S4)仅需序列长度的线性计算量即可实现长程建模。近期出现的Mamba[18]作为CNN与Transformer的新替代方案,通过集成数据依赖型SSM层扩展了S4框架,在自然语言处理任务中展现出竞争力,进一步推动了SSM模型的发展。受此启发,Vision Mamba[20]与VMamba[21]在通用视觉领域实现了基于数据依赖型SSM层的模型,取得显著性能。此外,U-Mamba[22]和SegMamba[23]作为SSM-CNN混合模型,初步展现了SSM在医学图像分割领域的潜力。然而,纯SSM模型在分割任务中的能力尚未得到充分探索。为此,本文首次提出纯SSM基础架构VM-UNet,该模型通过构建能够捕获广泛上下文信息的基础模块,为医学图像分割研究提供新范式。
3. 方法
3.1 预备知识
在现代基于状态空间模型(SSM)的架构中,结构化状态空间序列模型(S4)与Mamba均建立在经典的连续系统之上。该系统通过中间隐状态 h ( t ) ∈ R N h(t) \in \mathbb{R}^N h(t)∈RN 将一维输入函数或序列 x ( t ) ∈ R x(t) \in \mathbb{R} x(t)∈R 映射至输出 y ( t ) ∈ R y(t) \in \mathbb{R} y(t)∈R。该过程可表述为线性常微分方程:
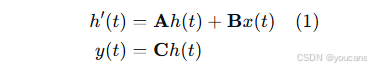
其中 A ∈ R N × N \mathbf{A} \in \mathbb{R}^{N \times N} A∈RN×N 为状态矩阵, B ∈ R N × 1 \mathbf{B} \in \mathbb{R}^{N \times 1} B∈RN×1 和 C ∈ R N × 1 \mathbf{C} \in \mathbb{R}^{N \times 1} C∈RN×1 为投影参数。
为适配深度学习场景,S4与Mamba需对该连续系统进行离散化。具体而言,通过引入时间尺度参数 Δ \mathbf{\Delta} Δ,并采用固定离散化规则(通常为零阶保持法)将 A \mathbf{A} A、 B \mathbf{B} B 转换为离散参数 A ‾ \mathbf{\overline{A}} A、 B ‾ \mathbf{\overline{B}} B:
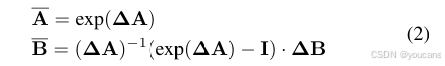
离散化后,基于SSM的模型可通过线性递归或全局卷积两种方式计算,分别对应公式(3)与公式(4):
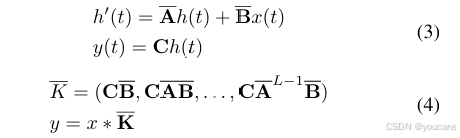
其中 K ‾ ∈ R L \mathbf{\overline{K}} \in \mathbb{R}^L K∈RL 为结构化卷积核, L L L 表示输入序列 x x x 的长度。
在本节中,我们首先介绍VM-UNet的整体结构。随后,我们详细阐述核心组件VSS块。最后,我们描述在训练过程中使用的损失函数。
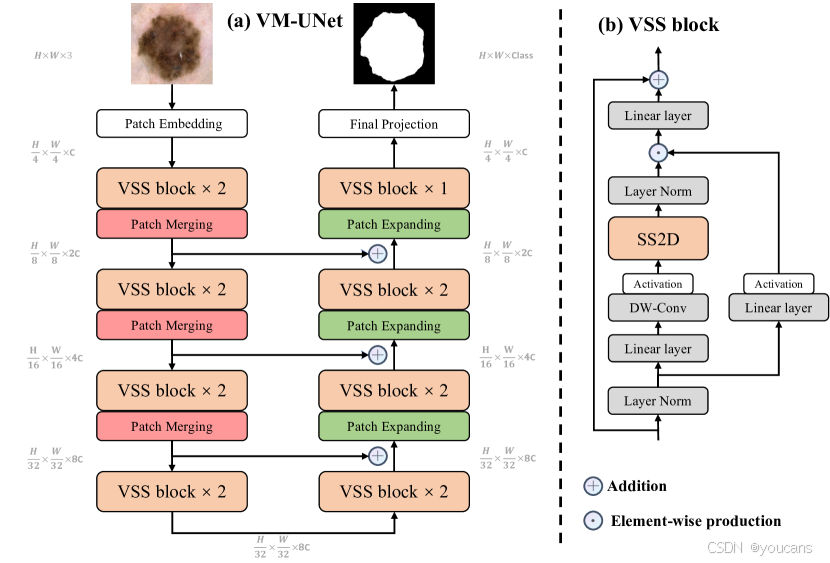
图2:(a) VM-UNet的整体架构。 (b) VSS块(视觉状态空间)是VM-UNet的主要构建块,而SS2D是VSS块中的核心操作。
3.2 视觉 Mamba UNet(VM-UNet)
如图2(a)所示,本文展示了VM-UNet的整体架构。该架构包含补丁嵌入层、编码器、解码器、最终投影层及跳跃连接。与先前方法[12]不同,我们未采用对称结构,而是使用了非对称设计。
补丁嵌入层将输入图像 x ∈ R H × W × 3 x \in \mathcal{R}^{H \times W \times 3} x∈RH×W×3 划分为 4 × 4 4 \times 4 4×4 的非重叠图块,随后将图像维度映射至 C C C(默认设为96)。该过程生成嵌入图像 x ′ ∈ R H 4 × W 4 × C x' \in \mathcal{R}^{\frac{H}{4} \times \frac{W}{4} \times C} x′∈R4H×4W×C。最后,我们通过层归一化[33]对 x ′ x' x′ 进行标准化,再送入编码器进行特征提取。编码器包含四个阶段,在前三阶段末尾应用补丁合并操作以降低特征图尺寸并增加通道数。四个阶段分别使用 [2, 2, 2, 2] 个VSS模块,各阶段通道数依次为 [C, 2C, 4C, 8C]。
解码器同样分为四个阶段。在后三阶段起始处通过补丁扩展操作减少特征通道数并增大空间尺寸。四个阶段分别使用 [2, 2, 2, 1]个VSS模块,通道数依次为 [8C, 4C, 2C, C]。解码器后接的最终投影层通过补丁扩展进行4倍上采样恢复特征图尺寸,再经投影层调整通道数以匹配分割目标。
跳跃连接采用极简的加法操作,未引入任何额外参数。
3.3 VSS模块
源自VMamba[21]的VSS模块是VM-UNet的核心组件,其结构如图2(b)所示。输入经层归一化后分流至双支路:第一支路通过线性层与激活函数;第二支路依次经线性层、深度可分离卷积与激活函数处理后,送入二维选择性扫描模块进行特征提取。随后对特征进行层归一化,并与第一支路输出进行逐元素相乘以融合双路信息。最后通过线性层进行特征混合,并与残差连接叠加构成模块输出。本文默认采用SiLU[34]作为激活函数。
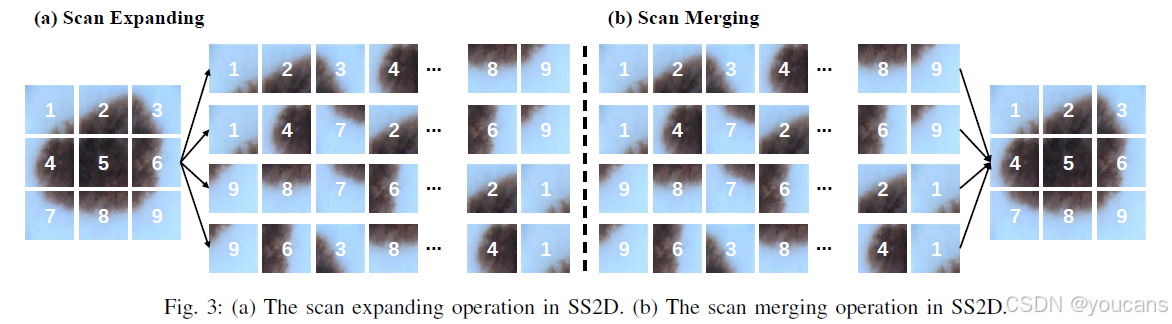
图3:(a) SS2D中的扫描扩展操作。 (b) SS2D中的扫描合并操作。
SS2D由扫描扩展、S6模块和扫描合并三部分组成。如图3(a)所示,扫描扩展操作沿四个方向(左上至右下、右下至左上、右上至左下、左下至右上)将输入图像展开为序列。这些序列经S6模块进行特征提取,确保全方位信息被充分扫描以捕获多样化特征。继而如图3(b)所示,扫描合并操作对四向序列进行加和归并,将输出图像恢复至输入尺寸。源自Mamba[18]的S6模块在S4[19]基础上引入选择性机制,通过根据输入调整SSM参数使模型能区分并保留相关信息、过滤无关信息。S6模块的伪代码详见算法1。

3.4 损失函数
VM-UNet的提出旨在验证纯SSM模型在医学图像分割任务中的应用潜力。因此,我们仅采用最基础的二元交叉熵与Dice损失(BceDice损失)及交叉熵与Dice损失(CeDice损失)分别作为二元分割与多类分割任务的损失函数,具体如公式(5)与公式(6)所示。

其中 N N N 表示样本总数, C C C 代表类别总数。 y i y_i yi 与 y ^ i \hat{y}i y^i 分别表示真实标签与预测标签。 y i , c y{i,c} yi,c 为指示函数,当样本 i i i 属于类别 c c c 时取值为1,否则为0。 y ^ i , c \hat{y}_{i,c} y^i,c 表示模型预测样本 i i i 属于类别 c c c 的概率。 ∣ X ∣ |X| ∣X∣ 与 ∣ Y ∣ |Y| ∣Y∣ 分别代表真实标注与预测结果。 λ 1 \lambda_1 λ1、 λ 2 \lambda_2 λ2、 λ 3 \lambda_3 λ3、 λ 4 \lambda_4 λ4 用于控制公式(5)与公式(6)中不同损失项的权重,默认经验性设置为1。
综上所述,我们提出了一个简洁而有效的训练框架VM-UNet,旨在探索纯SSM模型在医学图像分割中的潜在应用。此外,我们为医学图像分割任务建立了纯SSM模型的基准,为开发更高效、更强大的SSM方法提供了重要参考。据我们所知,这是首个基于纯SSM架构构建的医学图像分割模型。我们期待这项工作能为医学领域的相关研究提供启示,推动该方向的进一步发展。
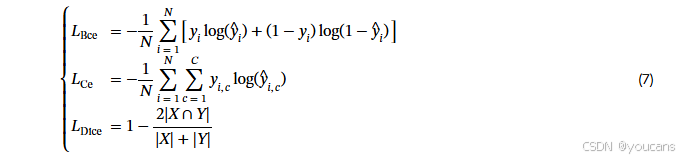
4. 实验
本节针对皮肤病变分割与器官分割任务,对VM-UNet进行了全面实验验证。具体而言,我们在ISIC17、ISIC18和Synapse数据集上评估了VM-UNet在医学图像分割任务中的性能表现。
5.1 数据集
ISIC17与ISIC18数据集:国际皮肤影像合作组织2017与2018挑战赛数据集(ISIC17与ISIC18)[25,35,26,36]是公开的皮肤病变分割数据集,分别包含2,150张和2,694张带分割掩码标签的皮肤镜图像。遵循先前研究[4]的设定,我们将数据集按7:3比例划分为训练集与测试集。具体而言,ISIC17数据集的训练集包含1,500张图像,测试集包含650张图像;ISIC18数据集的训练集包含1,886张图像,测试集包含808张图像。针对这两个数据集,我们从交并比均值(mIoU)、戴斯相似系数(DSC)、准确率(Acc)、灵敏度(Sen)和特异度(Spe)等多个指标进行了详细评估。
Synapse多器官分割数据集(Synapse):该公开数据集[24,45]包含30例腹部CT病例的3,779张轴位临床CT图像,涵盖8类腹部器官(主动脉、胆囊、左肾、右肾、肝脏、胰腺、脾脏、胃)。遵循前人研究设定[9,12],我们采用18例作为训练集,12例作为测试集。针对该数据集,我们汇报戴斯相似系数(DSC)与95%豪斯多夫距离(HD95)两项指标。
5.2 实现细节
参照先前研究[4,12],我们将ISIC17和ISIC18数据集的图像尺寸调整为256×256,Synapse数据集的图像尺寸调整为224×224。为防止过拟合,采用随机翻转与随机旋转等数据增强技术。ISIC17与ISIC18数据集使用BceDice损失函数,Synapse数据集采用CeDice损失函数。批大小设置为32,使用AdamW优化器[46]并设置初始学习率为1e-3。采用余弦退火学习率调度器[47],最大迭代50次,最小学习率为1e-5。训练周期设为300轮。对于VM-UNet,编码器与解码器权重均采用在ImageNet-1k上预训练的VMamba-S模型[21]进行初始化。所有实验均在单张NVIDIA RTX A6000 GPU上完成。
5.3 与先进模型对比
-
ISIC数据集
在ISIC17与ISIC18数据集上,我们将VM-UNet与多种先进模型进行对比。如表I所示,VM-UNet在皮肤病变分割任务中展现出强劲性能。具体而言,在ISIC17数据集上,VM-UNet在mIoU、DSC、Acc和Sen四项指标中均取得最佳结果;在ISIC18数据集上,VM-UNet亦在mIoU、DSC和Acc三项指标上超越其他模型。值得注意的是,相较于强大的Transformer-CNN混合模型TransFuse,我们的VM-UNet在mIoU和DSC指标上分别提升0.72%和0.44%。
-
Synapse数据集
针对Synapse数据集,我们在CNN模型、Transformer模型及混合模型间进行了更全面的对比。结果如表II所示:相较于首个纯Transformer图像分割模型Swin-UNet,我们的VM-UNet在DSC和HD95指标上分别提升1.95%与2.34毫米;特别是在"胃部"器官分割任务中,VM-UNet的DSC达到81.40%,较前人模型取得显著增益。这充分证明了基于SSM的模型在医学图像分割任务中相较于其他模型的优越性。
5.4 消融实验
5.4.1 初始权重
本节我们分别采用VMamba-T与VMamba-S的预训练权重对VM-UNet进行初始化,在ISIC17和ISIC18数据集上进行了消融实验(注:VMamba-T与VMamba-S在ImageNet-1k上取得的Top-1准确率分别为82.2%与83.5%)。实验结果如表III所示:相较于未使用预训练权重的VM-UNet,采用最强预训练模型VMamba-S初始化的VM-UNet在两个ISIC数据集的mIoU和DSC指标上平均提升2.67%与1.65%。这表明更强的预训练权重能显著提升VM-UNet在下游任务中的性能,印证了预训练权重对模型的重要影响。
5.4.2 丢弃率设置
为缓解下游图像分割任务中的过拟合现象,我们引入了丢弃技术[48]。如表IV所示,我们尝试了0.0至0.3间的不同丢弃率。在ISIC17数据集上,未使用丢弃技术(取值为0.0)时模型达到最佳性能,mIoU与DSC分别达80.45%与89.17%;而在ISIC18数据集上,最佳丢弃率为0.2,此时mIoU与DSC分别为81.35%与89.71%。这些发现表明丢弃率对VM-UNet性能的影响存在数据集差异性:虽然提升丢弃率会降低ISIC17数据集的性能,但适中的丢弃率可增强ISIC18数据集的泛化能力,暗示ISIC18数据集可能面临更显著的过拟合风险。
5.4.3 编码器-解码器架构
本文采用强编码器-弱解码器配置的非对称结构设计VM-UNet,以降低参数量与计算负载。为阐明该设计的优势,我们对模型架构进行了消融实验,结果如表V所示。具体而言,当采用对称结构{2,2,2,2-2,2,2,2}时,VM-UNet不仅参数量增加0.1M,计算负载也提升0.24 GFLOPs,同时导致性能下降。进一步将VM-UNet的模型规模扩大至{2,2,9,2-2,9,2,2}后,我们观察到模型性能持续衰退。因此,本文最终将VM-UNet设计为{2,2,2,2-2,2,2,1}的非对称结构。
5.4.4 输入分辨率
随着输入序列长度的增加,Mamba在DNA序列数据上展现出卓越性能[18]。然而在视觉Mamba模型(如Vim[20]与VMamba[21])中,尚未对输入序列长度进行系统性消融研究。因此,本节通过提升输入图像分辨率(从而增加输入序列长度),探究Mamba在视觉领域------特别是医学图像分割任务中的表现。如表VI所示,尽管输入序列长度增加,VM-UNet的性能却未达预期。该现象的具体成因值得进一步深入研究。
5.5 可视化分析
本节通过ISIC18数据集上的分割结果可视化,对比展示了VM-UNet与其他先进模型的性能,如图4所示。
这些结果证明了VM-UNet在多种挑战性场景下具有卓越的鲁棒性。
具体而言:首行可视化结果表明,TransFuse[11]和UTNetV2[37]等模型常对非目标区域产生误分类,导致分割性能下降,而VM-UNet在此方面表现出更强稳定性;第三行可视化案例显示,在小目标分割任务中,VM-UNet能有效减少冗余预测;第四行及末行可视化进一步表明,该模型能精准处理复杂边界的分割任务,即使面对高度不规则的轮廓仍能准确勾勒目标边缘。这些可视化证据共同验证了基于纯SSM架构的VM-UNet在医学图像分割领域的重要潜力。
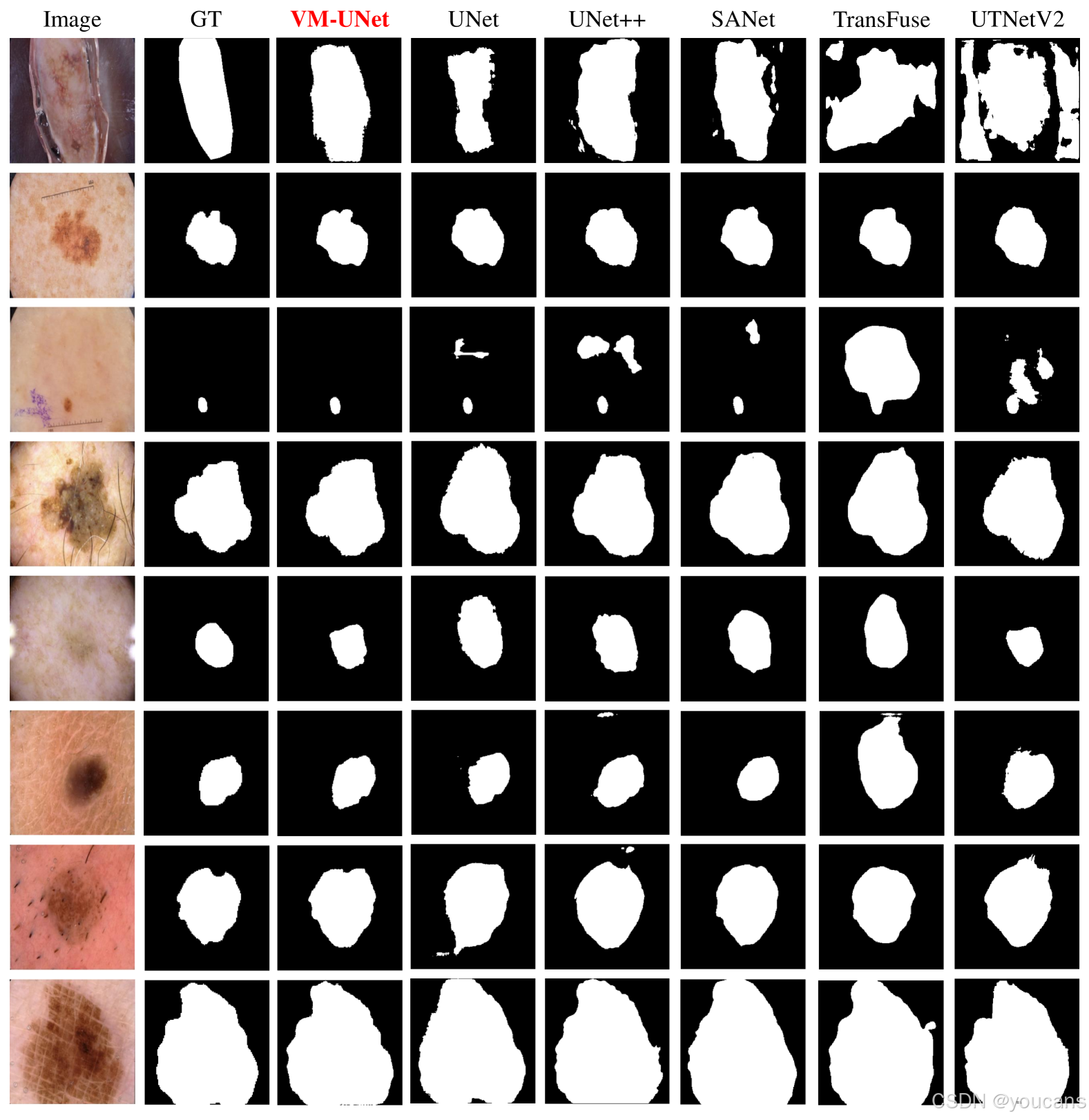
图4:ISIC18数据集上的视觉对比。GT表示 ground truth。
5.6 讨论
本研究提出VM-UNet框架以探索纯SSM模型在医学图像分割中的应用潜力。尽管该方法在部分数据集上取得了良好性能,但当前SSM版本仍存在明显局限:
-
首先,结构化SSM最初被定义为连续系统的离散化形式,对感知信号(如音频、视频)等连续时间数据模态具有强归纳偏置,这可能导致模型在长度泛化能力上的不足------尤其当处理超出训练序列长度的数据时。此外,VM-UNet参数量约30M,需通过人工设计或压缩策略进一步精简,以增强其在真实医疗场景中的适用性。
-
其次,SSM的优势在于长序列信息捕获能力,这为在更大分辨率下系统探索纯SSM模型的分割性能创造了条件。然而当前实证研究受限于较小模型规模(未达到多数强开源LLM的参数量阈值),在实际部署时需对这些问题进行深入考量。
-
最后,本文方法虽能动态捕获医学图像分割所需的广泛上下文信息,但在皮肤病变分割中仍存在局限:如图5所示,模型仅能有效分割深色区域(1-2行),对浅色区域预测效果欠佳;同时由于视觉表征中的上下文判别特性,模型对毛发干扰(第3行)高度敏感,可能对下游任务产生负面影响。这些挑战在真实场景部署VM-UNet时需重点研究解决。
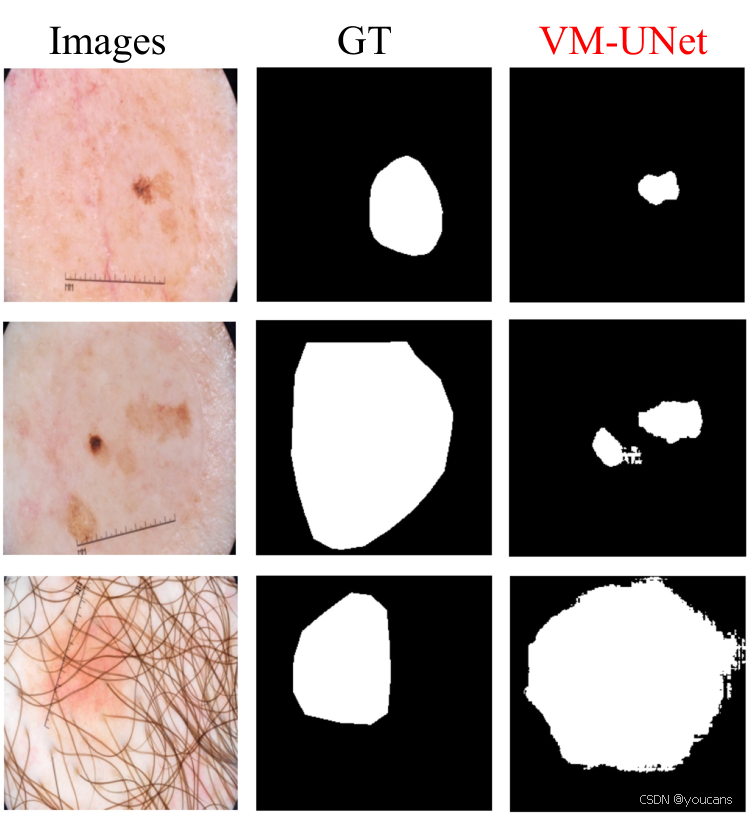
图5:VM-UNet在ISIC18数据集上的部分失败案例。
6. 结论
本文首次在医学图像分割领域引入纯基于状态空间模型的架构,提出了作为基准模型的VM-UNet。为充分发挥SSM模型的潜能,我们采用VSS模块构建VM-UNet,并使用预训练的VMamba-S权重进行初始化。在皮肤病变和多器官分割数据集上的综合实验表明,纯SSM模型在医学图像分割任务中具有显著竞争力,值得医学界深入探索。未来我们将继续探索SSM在其他医学影像任务(如检测、配准与重建等)中的应用。
7. GitHub 项目介绍
7.1 环境配置
bash
conda create -n vmunet python=3.8
conda activate vmunet
pip install torch==1.13.0 torchvision==0.14.0 torchaudio==0.13.0 --extra-index-url https://download.pytorch.org/whl/cu117
pip install packaging
pip install timm==0.4.12
pip install pytest chardet yacs termcolor
pip install submitit tensorboardX
pip install triton==2.0.0
pip install causal_conv1d==1.0.0 # 对应文件:causal_conv1d-1.0.0+cu118torch1.13cxx11abiFALSE-cp38-cp38-linux_x86_64.whl
pip install mamba_ssm==1.0.1 # 对应文件:mamba_ssm-1.0.1+cu118torch1.13cxx11abiFALSE-cp38-cp38-linux_x86_64.whl
pip install scikit-learn matplotlib thop h5py SimpleITK scikit-image medpy yacscausal_conv1d 和 mamba_ssm 的.whl 安装文件可通过百度网盘或Google云盘获取
7.2 使用步骤
- 数据集准备
(1)ISIC数据集
按7:3比例划分的ISIC17和ISIC18数据集可从百度网盘获取。下载后请将数据集分别放置于:
bash
./data/isic17/
├── train
│ ├── images
│ │ └── *.png
│ └── masks
│ └── *.png
└── val
├── images
│ └── *.png
└── masks
└── *.png(2)ISIC18数据集目录结构同上
(3)Synapse数据集
可参照Swin-UNet的指南获取原始数据,或从百度网盘下载预处理版本。下载后组织为以下结构:
bash
./data/Synapse/
├── lists
│ └── list_Synapse
│ ├── all.lst
│ ├── test_vol.txt
│ └── train.txt
├── test_vol_h5
│ └── casexxxx.npy.h5
└── train_npz
└── casexxxx_slicexxx.npz
bash
cd VM-UNet
python train.py # 在ISIC17/ISIC18数据集上训练并测试
python train_synapse.py # 在Synapse数据集上训练并测试注:若仅需使用训练好的模型进行推理测试并保存结果图像:
(1)在配置文件config_setting中:
-
设置参数only_test_and_save_figs = True
-
在best_ckpt_path中填入训练好的模型路径
-
在img_save_path中指定结果保存路径
(2)执行脚本:完成上述设置后运行train.py
8. 参考文献
bash
[1] O. Ronneberger, P. Fischer, and T. Brox, "U-net: Convolutional networks for biomedical image segmentation," in International Conference on Medical image computing and computer-assisted intervention. Springer, 2015, pp. 234--241.
[2] ¨ O. C¸ic¸ek, A. Abdulkadir, S. S. Lienkamp, T. Brox, and O. Ronneberger, "3d u-net: learning dense volumetric segmentation from sparse annotation," in International conference on medical image computing and computer-assisted intervention. Springer, 2016, pp. 424--432.
[3] Z. Zhou, M. M. Rahman Siddiquee, N. Tajbakhsh, and J. Liang, "Unet++: A nested u-net architecture for medical image segmentation," in Deep learning in medical image analysis and multimodal learning for clinical decision support. Springer, 2018, pp. 3--11.
[4] J. Ruan, S. Xiang, M. Xie, T. Liu, and Y. Fu, "Malunet: A multi-attention and light-weight unet for skin lesion segmentation," in 2022 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). IEEE, 2022, pp. 1150--1156.
[5] J. Ruan, M. Xie, J. Gao, T. Liu, and Y. Fu, "Ege-unet: an efficient group enhanced unet for skin lesion segmentation," in International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, 2023, pp. 481--490.
[6] J. Ruan, M. Xie, S. Xiang, T. Liu, and Y. Fu, "Mew-unet: Multiaxis representation learning in frequency domain for medical image segmentation," arXiv preprint arXiv:2210.14007, 2022.
[7] H. Li, D.-H. Zhai, and Y. Xia, "Erdunet: An efficient residual doublecoding unet for medical image segmentation," IEEE Transactions on Circuits and Systems for Video Technology, 2023.
[8] T. Zhou, Y. Zhou, G. Li, G. Chen, and J. Shen, "Uncertainty-aware hierarchical aggregation network for medical image segmentation," IEEE Transactions on Circuits and Systems for Video Technology, 2024.
[9] J. Chen, Y. Lu, Q. Yu, X. Luo, E. Adeli, Y. Wang, L. Lu, A. L. Yuille, and Y. Zhou, "Transunet: Transformers make strong encoders for medical image segmentation," arXiv preprint arXiv:2102.04306, 2021.
[10] A. Dosovitskiy, L. Beyer, A. Kolesnikov, D. Weissenborn, X. Zhai, T. Unterthiner, M. Dehghani, M. Minderer, G. Heigold, S. Gelly et al., "An image is worth 16x16 words: Transformers for image recognition at scale," arXiv preprint arXiv:2010.11929, 2020.
[11] Y. Zhang, H. Liu, and Q. Hu, "Transfuse: Fusing transformers and cnns for medical image segmentation," in International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, 2021, pp. 14--24.
[12] H. Cao, Y. Wang, J. Chen, D. Jiang, X. Zhang, Q. Tian, and M. Wang, "Swin-unet: Unet-like pure transformer for medical image segmentation," in European conference on computer vision. Springer, 2022, pp. 205--218.
[13] Z. Liu, Y. Lin, Y. Cao, H. Hu, Y. Wei, Z. Zhang, S. Lin, and B. Guo, "Swin transformer: Hierarchical vision transformer using shifted windows," in Proceedings of the IEEE/CVF International Conference on Computer Vision, 2021, pp. 10012--10022.
[14] J. Liu, Y. Chen, B. Ni, and Z. Yu, "Joint global and dynamic pseudo labeling for semi-supervised point cloud sequence segmentation," IEEE Transactions on Circuits and Systems for Video Technology, 2023.
[15] A. Vaswani, N. Shazeer, N. Parmar, J. Uszkoreit, L. Jones, A. N. Gomez, Ł. Kaiser, and I. Polosukhin, "Attention is all you need," Advances in neural information processing systems, vol. 30, 2017.
[16] P. Mu, G. Wu, J. Liu, Y. Zhang, X. Fan, and R. Liu, "Learning to search a lightweight generalized network for medical image fusion," IEEE Transactions on Circuits and Systems for Video Technology, 2023.
[17] R. E. Kalman, "A new approach to linear filtering and prediction problems," 1960.
[18] A. Gu and T. Dao, "Mamba: Linear-time sequence modeling with selective state spaces," arXiv preprint arXiv:2312.00752, 2023.
[19] A. Gu, K. Goel, and C. R´e, "Efficiently modeling long sequences with structured state spaces," arXiv preprint arXiv:2111.00396, 2021.
[20] L. Zhu, B. Liao, Q. Zhang, X. Wang, W. Liu, and X. Wang, "Vision mamba: Efficient visual representation learning with bidirectional state space model," arXiv preprint arXiv:2401.09417, 2024.
[21] Y. Liu, Y. Tian, Y. Zhao, H. Yu, L. Xie, Y. Wang, Q. Ye, and Y. Liu, "Vmamba: Visual state space model," arXiv preprint arXiv:2401.10166, 2024.
[22] J. Ma, F. Li, and B. Wang, "U-mamba: Enhancing long-range dependency for biomedical image segmentation," arXiv preprint arXiv:2401.04722, 2024.
[23] Z. Xing, T. Ye, Y. Yang, G. Liu, and L. Zhu, "Segmamba: Long-range sequential modeling mamba for 3d medical image segmentation," arXiv preprint arXiv:2401.13560, 2024.
[24] B. Landman, Z. Xu, J. Igelsias, M. Styner, T. Langerak, and A. Klein, "Miccai multi-atlas labeling beyond the cranial vault--workshop and challenge," in Proc. MICCAI Multi-Atlas Labeling Beyond Cranial Vault---Workshop Challenge, vol. 5, 2015, p. 12.
[25] M. Berseth, "Isic 2017-skin lesion analysis towards melanoma detection," arXiv preprint arXiv:1703.00523, 2017.
[26] N. Codella, V. Rotemberg, P. Tschandl, M. E. Celebi, S. Dusza, D. Gutman, B. Helba, A. Kalloo, K. Liopyris, M. Marchetti et al., "Skin lesion analysis toward melanoma detection 2018: A challenge hosted by the international skin imaging collaboration (isic)," arXiv preprint arXiv:1902.03368, 2019.
[27] H. Seo, M. Badiei Khuzani, V. Vasudevan, C. Huang, H. Ren, R. Xiao, X. Jia, and L. Xing, "Machine learning techniques for biomedical image segmentation: an overview of technical aspects and introduction to stateof-art applications," Medical physics, vol. 47, no. 5, pp. e148--e167, 2020.
[28] S. Sun, D. R. Haynor, and Y. Kim, "Semiautomatic video object segmentation using vsnakes," IEEE Transactions on Circuits and Systems for Video Technology, vol. 13, no. 1, pp. 75--82, 2003.
[29] O. Oktay, J. Schlemper, L. L. Folgoc, M. Lee, M. Heinrich, K. Misawa, K. Mori, S. McDonagh, N. Y. Hammerla, B. Kainz et al., "Attention u-net: Learning where to look for the pancreas," arXiv preprint arXiv:1804.03999, 2018.
[30] A. Lin, B. Chen, J. Xu, Z. Zhang, G. Lu, and D. Zhang, "Ds-transunet: Dual swin transformer u-net for medical image segmentation," IEEE Transactions on Instrumentation and Measurement, 2022.
[31] A. Gu, T. Dao, S. Ermon, A. Rudra, and C. R´e, "Hippo: Recurrent memory with optimal polynomial projections," Advances in neural information processing systems, vol. 33, pp. 1474--1487, 2020.
[32] A. Gu, I. Johnson, K. Goel, K. Saab, T. Dao, A. Rudra, and C. R´e, "Combining recurrent, convolutional, and continuous-time models with linear state space layers," Advances in neural information processing systems, vol. 34, pp. 572--585, 2021.
[33] J. L. Ba, J. R. Kiros, and G. E. Hinton, "Layer normalization," arXiv preprint arXiv:1607.06450, 2016.
[34] S. Elfwing, E. Uchibe, and K. Doya, "Sigmoid-weighted linear units for neural network function approximation in reinforcement learning," Neural networks, vol. 107, pp. 3--11, 2018.
[35] https://challenge.isic-archive.com/data/#2017.
[36] https://challenge.isic-archive.com/data/#2018.
[37] Y. Gao, M. Zhou, D. Liu, and D. Metaxas, "A multi-scale transformer for medical image segmentation: Architectures, model efficiency, and benchmarks," arXiv preprint arXiv:2203.00131, 2022.
[38] J. Wei, Y. Hu, R. Zhang, Z. Li, S. K. Zhou, and S. Cui, "Shallow attention network for polyp segmentation," in International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, 2021, pp. 699--708.
[39] F. Milletari, N. Navab, and S.-A. Ahmadi, "V-net: Fully convolutional neural networks for volumetric medical image segmentation," in 2016 fourth international conference on 3D vision (3DV). IEEE, 2016, pp. 565--571.
[40] M. Z. Alom, C. Yakopcic, M. Hasan, T. M. Taha, and V. K. Asari, "Recurrent residual u-net for medical image segmentation," Journal of Medical Imaging, vol. 6, no. 1, pp. 014006--014006, 2019.
[41] R. Azad, M. T. Al-Antary, M. Heidari, and D. Merhof, "Transnorm: Transformer provides a strong spatial normalization mechanism for a deep segmentation model," IEEE Access, vol. 10, pp. 108205--108215, 2022.
[42] R. Azad, M. Heidari, M. Shariatnia, E. K. Aghdam, S. Karimijafarbigloo, E. Adeli, and D. Merhof, "Transdeeplab: Convolution-free transformerbased deeplab v3+ for medical image segmentation," in International Workshop on PRedictive Intelligence In MEdicine. Springer, 2022, pp. 91--102.
[43] H. Wang, P. Cao, J. Wang, and O. R. Zaiane, "Uctransnet: rethinking the skip connections in u-net from a channel-wise perspective with transformer," in Proceedings of the AAAI Conference on Artificial Intelligence, vol. 36, no. 3, 2022, pp. 2441--2449.
[44] H. Wang, S. Xie, L. Lin, Y. Iwamoto, X.-H. Han, Y.-W. Chen, and R. Tong, "Mixed transformer u-net for medical image segmentation," in ICASSP 2022-2022 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). IEEE, 2022, pp. 2390--2394.
[45] https://www.synapse.org/#!Synapse:syn3193805/wiki/217789.
[46] I. Loshchilov and F. Hutter, "Decoupled weight decay regularization," arXiv preprint arXiv:1711.05101, 2017.
[47] Loshchilov, Ilya and Hutter, Frank, "Sgdr: Stochastic gradient descent with warm restarts," arXiv preprint arXiv:1608.03983, 2016.
[48] N. Srivastava, G. Hinton, A. Krizhevsky, I. Sutskever, and R. Salakhutdinov, "Dropout: a simple way to prevent neural networks from overf itting," The journal of machine learning research, vol. 15, no. 1, pp. 1929--1958, 2014引用格式: Ruan, J., Xiang, S.: VM-UNET: vision mamba UNet for medical image segmentation. arXiv preprint arXiv:2402.02491 (2024).
版权说明:
youcans@xidian 作品,转载必须标注原文链接:
【youcans论文精读】VM-UNet:面向医学图像分割的视觉Mamba UNet架构
Crated:2025-11