引言
空间转录组数据分析依赖大量 Python/R 包和系统级依赖,版本冲突是常见问题。Conda 作为环境管理工具,能够创建完全隔离、可移植的软件环境,确保分析流程的稳定性和可重复性。VSCode 作为轻量级但功能强大的编辑器,其 Remote 开发扩展允许开发者在本地编辑代码,而实际运行环境位于远程服务器或 WSL 子系统中,实现"本地手感,远程算力"的无缝体验。二者的结合,兼顾了环境配置的灵活性与开发效率,是生信分析的理想方案。
第一部分:基础设施
场景 A:如果你有远程 Linux 服务器
你需要准备的是:
-
服务器 IP 地址
-
用户名和密码
-
SSH 端口(默认22)
场景 B:如果你使用本地 Windows 电脑 (WSL2)
对于没有服务器,但又想体验 Linux 强大命令行工具的 Windows 用户,可以使用WSL2 (Windows Subsystem for Linux 2) ,它不是虚拟机,而是深度集成在 Windows 内的完整 Linux 内核。
1. 开启 WSL2
(注:要求 Windows 10 版本 2004+ 或 Windows 11)
-
以管理员身份打开 PowerShell。
-
输入命令:
wsl --install这条指令会自动开启必要的虚拟化功能,并默认下载安装 Ubuntu 发行版。
-
重启电脑。
-
重启后,Ubuntu 终端会自动弹出,提示你设置 UNIX 用户名和密码(注意:输入密码时屏幕不会显示,这是 Linux 的特性,放心输)。
为什么选择 WSL2 而不是虚拟机?
-
启动秒开:WSL 几乎瞬间启动,而 VM 需要几分钟。
-
文件互通 :在 Linux 里可以直接访问 C 盘 (
/mnt/c),在 Windows 资源管理器里可以直接访问 Linux 文件 (\\wsl$)。 -
资源动态分配:不会像虚拟机那样预先切走你的内存,用多少占多少。
第二部分:VS Code连接一切
1. 下载与安装
去 code.visualstudio.com 下载安装。
2. 必装插件清单
打开 VS Code 左侧的扩展商店(四个方块的图标),搜索并安装:
-
Remote - SSH: 连接远程服务器的核心。
-
WSL: 连接本地 WSL 子系统的核心。
-
Python: 提供代码补全、调试。
-
R: 强大的 R 语言支持(LSP)。
-
Jupyter: 在 VS Code 里直接跑 Notebook。
-
Remote - Development: 这是一个插件包,包含了上面提到的 Remote 系列。
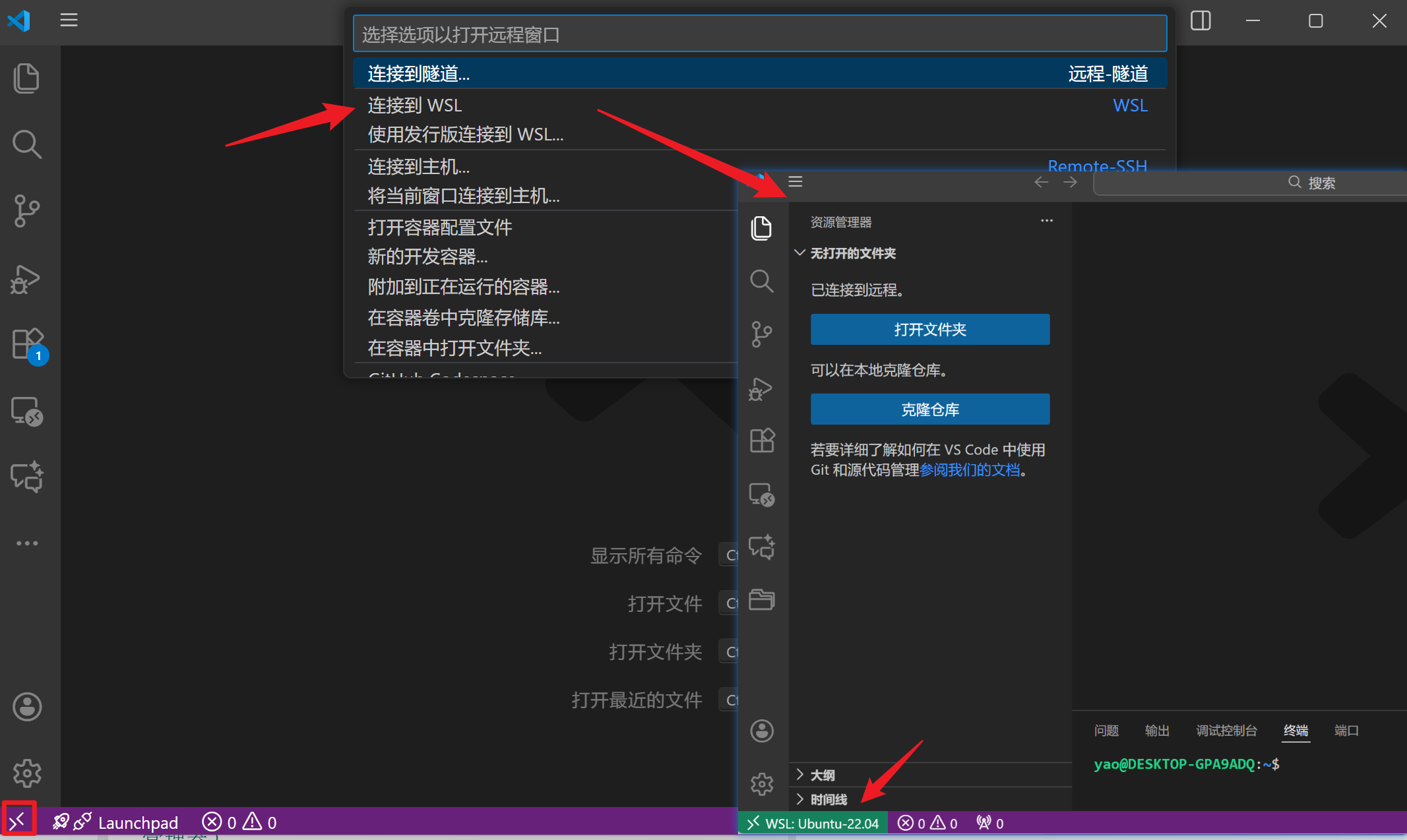
3. 连接 WSL (本地用户)
-
打开 VS Code。
-
点击左下角的绿色按钮
><。 -
选择 **"Connect to WSL"**。
-
瞬间,你的 VS Code 窗口就"穿越"到了 Ubuntu 内部。打开终端(Ctrl+`),你会发现是 Linux 的 shell。
4. 连接远程服务器 (SSH)
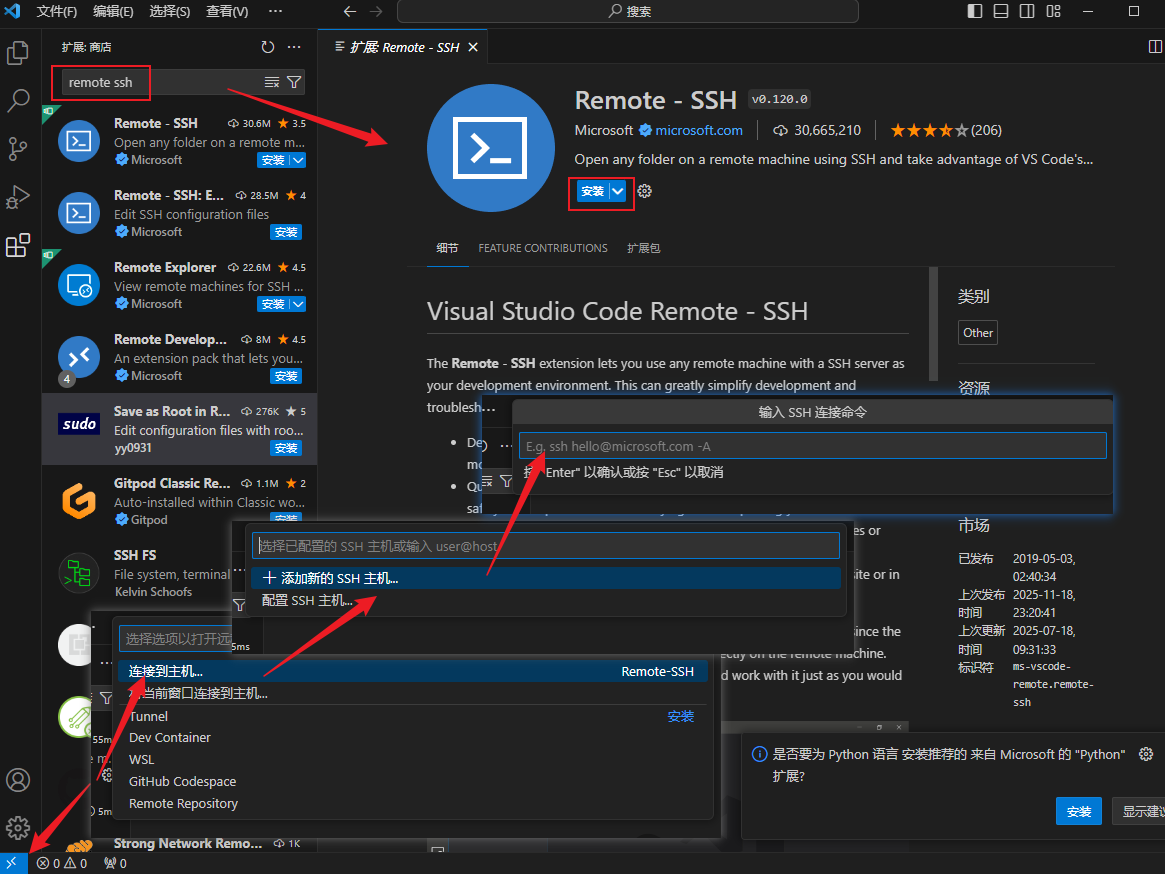
基础连接
-
点击左下角的绿色按钮
><。 -
选择 连接到主机。
-
点击加号
+ 添加新的ssh主机,输入:ssh username@your_server_ip -p 22 -A。 -
按回车,选择配置文件保存位置(默认
C:\Users\YourName\.ssh\config)。 -
在列表中右键你的服务器,选择 **"Connect to Host in Current Window"**。
-
输入密码,连接成功。
进阶:配置免密登录 (Public Key Authentication)
每次输密码太烦?尤其是服务器网络波动掉线重连时。
-
在本地电脑生成密钥: 打开本地终端(PowerShell 或 CMD):
ssh-keygen -t rsa -b 4096一路回车。这会在
~/.ssh/下生成id_rsa(私钥) 和id_rsa.pub(公钥)。 -
上传公钥到服务器 : 你需要把
id_rsa.pub的内容追加到服务器的~/.ssh/authorized_keys文件中。方法一(手动): 用记事本打开本地
id_rsa.pub,复制内容。 登录服务器,运行:mkdir -p ~/.ssh nano ~/.ssh/authorized_keys # 粘贴内容,保存退出 (Ctrl+O, Enter, Ctrl+X) chmod 600 ~/.ssh/authorized_keys方法二(PowerShell 自动化):
type $env:USERPROFILE\.ssh\id_rsa.pub | ssh user@host "cat >> .ssh/authorized_keys" -
配置 VS Code 的 Config 文件 : 打开 VS Code 的 SSH 配置文件,添加
IdentityFile字段:Host MySuperServer HostName 192.168.1.100 User xiaoming Port 22 IdentityFile "C:\Users\XiaoMing\.ssh\id_rsa"现在,你可以秒连服务器,无需密码。
第三部分:Conda环境管理
有了 Linux 环境(无论是远程还是 WSL),第一步绝不是安装软件,而是安装包管理器。
这里我们强烈推荐 **Miniforge (Mambaforge)**,而不是 Anaconda。
-
Anaconda:太臃肿,预装几百个你不需要的包。
-
Miniconda:干净,但默认解算器(Solver)速度慢。
-
Mamba :C++ 重写的 Conda,速度快 10-100 倍,解决依赖冲突的能力极强。
1. 安装 Miniforge (自带 Mamba)
在你的 Linux 终端中执行:
# 下载安装脚本
wget "https://github.com/conda-forge/miniforge/releases/latest/download/Miniforge3-Linux-x86_64.sh"
# 执行安装
bash Miniforge3-Linux-x86_64.sh
# 一路 Enter,遇到 Do you wish the installer to initialize Miniforge3? 选 yes安装完成后,重启终端 或输入 source ~/.bashrc。你会看到命令行前面多了一个 (base)。
2. 配置镜像源
空间转录组的包通常很大,如果不换源,下载速度会让你怀疑人生。编辑 ~/.condarc 文件:
channels:
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
show_channel_urls: true
default_channels:
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/r/
custom_channels:
conda-forge: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud
bioconda: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud🌟 环境一:python分析环境 (spatial-py)
# 1. 创建环境,指定 python 版本(推荐 3.10 或 3.11)
mamba create -n spatial-py python=3.11
# 2. 激活环境
mamba activate spatial-py
# 3. 安装各种库
mamba install leidenalg spatialdata[extra]==0.4.0 scanpy==1.11.2 scikit-misc==0.5.1 squidpy==1.6.5 harmonypy==0.0.10 geosketch==1.3 igraph==0.11.8
# 5. 安装其他常用分析库
mamba install matplotlib seaborn pandas numpy scipy statsmodels scikit-learn
# 6. 安装 Jupyter 内核支持
mamba install ipykernel🔍 避坑指南: 如果你在安装 squidpy 时遇到 GDAL 相关的报错,这通常是系统库缺失。使用 Conda 安装的好处是它会把这些二进制依赖都打包好。如果必须使用 pip 安装,请确保你已经安装了系统级的 libgdal-dev。
🌟 环境二:R 语言分析环境 (Spatial-R)
Seurat V5 对空间转录组的支持大幅增强。虽然在 Conda 里装 R 经常被诟病,但只要配置得当,它比在系统里编译 R 要方便得多。
# 1. 创建环境,直接指定 R-base 版本(推荐 4.3+)
mamba create -n spatial-r r-base=4.3.1
# 2. 激活环境
mamba activate spatial-r
# 3. 安装关键的系统依赖(这一步至关重要!)
# 很多 R 包安装失败是因为缺了 Linux 的动态库,Conda 可以直接装这些库
mamba install -c conda-forge r-devtools r-hdf5r r-rgeos r-rgdal r-sf r-cairo r-png r-jpeg pkg-config libxml2 libgit2
# 4. 安装 R 内核以便在 VS Code 中使用
mamba install r-irkernel
# 5. 进入 R 终端安装生物信息包
R在 R 终端内部:
# 这里的安装我们使用 R 自己的 install.packages 或 devtools
# 因为 CRAN/Bioconductor 的包更新比 Conda 快
# 安装 Seurat V5
install.packages('Seurat')
# 安装 Bioconductor 基础
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
# 安装空间数据常用的读取库
BiocManager::install(c('rhdf5', 'limma', 'ComplexHeatmap'))
# 注册内核给 Jupyter
IRkernel::installspec(name = 'spatial-r', displayname = 'R (Spatial)')第四部分:实战演练与工作流整合
现在,万事俱备。我们将演示如何在 VS Code 中调用刚才创建的 Conda 环境来跑一段空间转录组代码。
1. 项目文件管理
在连接到远程/WSL 的 VS Code 中:
-
File->Open Folder。 -
创建一个项目文件夹,例如
/home/username/project_spatial。 -
新建一个 Jupyter Notebook 文件:
analysis.ipynb。
2. 选择内核 (The Magic Step)
这是新手最容易卡住的地方:怎么让 VS Code 知道我要用 spatial-py 这个环境?
-
打开
analysis.ipynb。 -
点击右上角的 **"Select Kernel"**。
-
选择 **"Python Environments..."**。
-
列表里会自动显示出 Conda 检测到的环境。选择
spatial-py (Python 3.9)。 *(如果是 R 语言,选择 R 核心,然后选 spatial-r)*。
3. 测试代码 (Python 版)
import scanpy as sc
import squidpy as sq
# 打印版本,确认环境无误
print(f"Scanpy version: {sc.__version__}")
print(f"Squidpy version: {sq.__version__}")
# 下载一个示例空间数据 (Visium)
# 注意:这需要网络连接,如果服务器不能联网,需手动下载数据
adata = sq.datasets.visium_fluo_image_crop()
# 简单的可视化
sq.pl.spatial_scatter(adata, color="cluster")当你点击运行,图片直接渲染在 VS Code 内部。你可以缩放、保存,体验堪比本地 APP。
4. 解决"服务器无图形界面"报错
在做空间分析时,如果你用 matplotlib 直接 plt.show(),有时会报错 UserWarning: Matplotlib is currently using agg, which is a non-GUI backend。
解决方案 : 在 VS Code + Jupyter 模式下,这通常不是问题,因为 Jupyter 会内联显示图片。但如果你跑 .py 脚本,请确保开头加上:
import matplotlib
matplotlib.use('Agg') # 设置为非交互式后端
import matplotlib.pyplot as plt
# 画图后必须保存,不能 show
plt.plot(...)
plt.savefig('result.png')