在进行apa分析的时候发现如果对生成的loop列表进行评估,就必须输入对应细胞类型的bulk hic数据,但是在snaphic中只是给出了对应bulk hic数据的参考文件,而且在GEO上只能下载fastq,没有找到直接是hic数据类型的数据。
问过人工智能,通过分析有以下回答:(包括了小鼠和人类的数据集)
✅ SnapHiC 使用的 bulk Hi-C 数据来源(从论文和代码确定)
在 SnapHiC 论文(Nature Methods 2021)中,他们使用 bulk Hi-C 数据 只用于 benchmark(构建 reference loop list) 。这些 bulk Hi-C 数据全部来自 Bonev et al., Cell 2017 的小鼠胚胎干细胞(mES)Hi-C。
在 Methods 里写得很清楚:
"HiCCUPS loops at 10-kb resolution from bulk in situ Hi-C using pooled datasets of all four biological replicates from the Bonev et al. study."
所以你需要下载的是:
📌 SnapHiC 使用的 bulk Hi-C 数据(mES)
来自 Bonev et al., Cell 2017, GSE96107
你需要下载:
1. Bulk in situ Hi-C (4 个 replicates)
GEO accession:
🔗 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE96107
你可以在 GEO 的 "SRA Run Selector" 下载:
-
mES in situ Hi-C replicate 1
-
mES in situ Hi-C replicate 2
-
mES in situ Hi-C replicate 3
-
mES in situ Hi-C replicate 4
格式是 FASTQ,可以直接用于 Juicer 或 HiC-Pro。
📌 SnapHiC-G 使用的 bulk Hi-C、HiChIP、PLAC-seq(用于评估)
SnapHiC-G 用三类 bulk 数据作为 ground truth:
2. H3K4me3 PLAC-seq (for mES)
GEO:
🔗 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE99152
3. Cohesin HiChIP (mES)
可能来自 Mumbach et al. 2017:
🔗 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE80820
4. H3K27ac HiChIP (mES)
同样来自 Mumbach et al.:
📌 如何下载这些 bulk Hi-C/HiChIP 数据(最简单步骤)
方法 A(推荐)------ 使用 GEO → SRA Run Selector 一键下载
例如下载:GSE96107
-
打开:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE96107 -
找到:SRA Run Selector
-
点击:Download → Accession List
-
然后用命令行下载:
prefetch SRRxxxxxx fastq-dump --split-files SRRxxxxxx
方法 B ------ 直接用 wget 下载 fastq.gz(某些 GEO 会给 FTP)
进入 GEO → "FTP" → 选择 fastq 文件:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRRxxxx/SRRxxxx.fastq.gz
📌 如果你只想要 loop-level ground truth(BED 文件) 而不是原始 Hi-C
SnapHiC 提供了:
-
bulk Hi-C loop list(HiCCUPS)
-
PLAC-seq interaction list
-
H3K27ac HiChIP interaction list
-
cohesin HiChIP interaction list
地址:
🔗 https://github.com/HuMingLab/SnapHiC/tree/master/data
里面你可以直接下载他们用于 benchmark 的所有 reference loops,无需处理 fastq。
具体实施:
SnapHiC 使用的 bulk Hi-C 来自:
Bonev et al., Cell 2017 --- mouse embryonic stem cells (mES)
GEO:GSE96107
这篇文章的 bulk Hi-C 已经上传到 4DNucleome 数据库,可以直接下载现成的 .hic 文件:
来源:4DNucleome data portal (4DN)
这些是 HiC-Pro + Juicer 标准化处理后的 .hic 文件,非常适合做 APA。
🔗 mES bulk Hi-C(Bonev 2017)
打开:
Bonev B et al. (2017) PMID:29053968 -- 4DN Data Portal
你会看到多个文件,其中 .hic 格式通常对应:
- 4DNESxxxxxx.hic(KR normalized / multi-resolution)
例如:
| File name | Species | Resolution | Notes |
|---|---|---|---|
| 4DNESJZQTS2V.hic | Mouse (mm10) | multi-res | Bonev et al. bulk Hi-C |
| 4DNES6D7YQ9F.hic | Mouse (mm10) | multi-res | Bonev replicate |
| 4DNESR6P6W5L.hic | Mouse (mm10) | multi-res | Bonev replicate |
4DN 会提供:
-
hic
-
cool
-
mcool
你选择 hic 最适合直接用于 APA。
📌 如何确认这是 SnapHiC 用的同一批 mES bulk Hi-C?
SnapHiC 方法部分明确写:
"HiCCUPS loops ... from Bonev et al. bulk in situ Hi-C (GSE96107)".
4DN 收录的 Bonev 数据就是从 GSE96107 导入的。
所以 你直接用 4DN 的 hic 文件 = SnapHiC 所用 bulk HiC 的 hic 文件。
打开以后可以看到有下面的这些数据集,那么我们应该选哪一个?
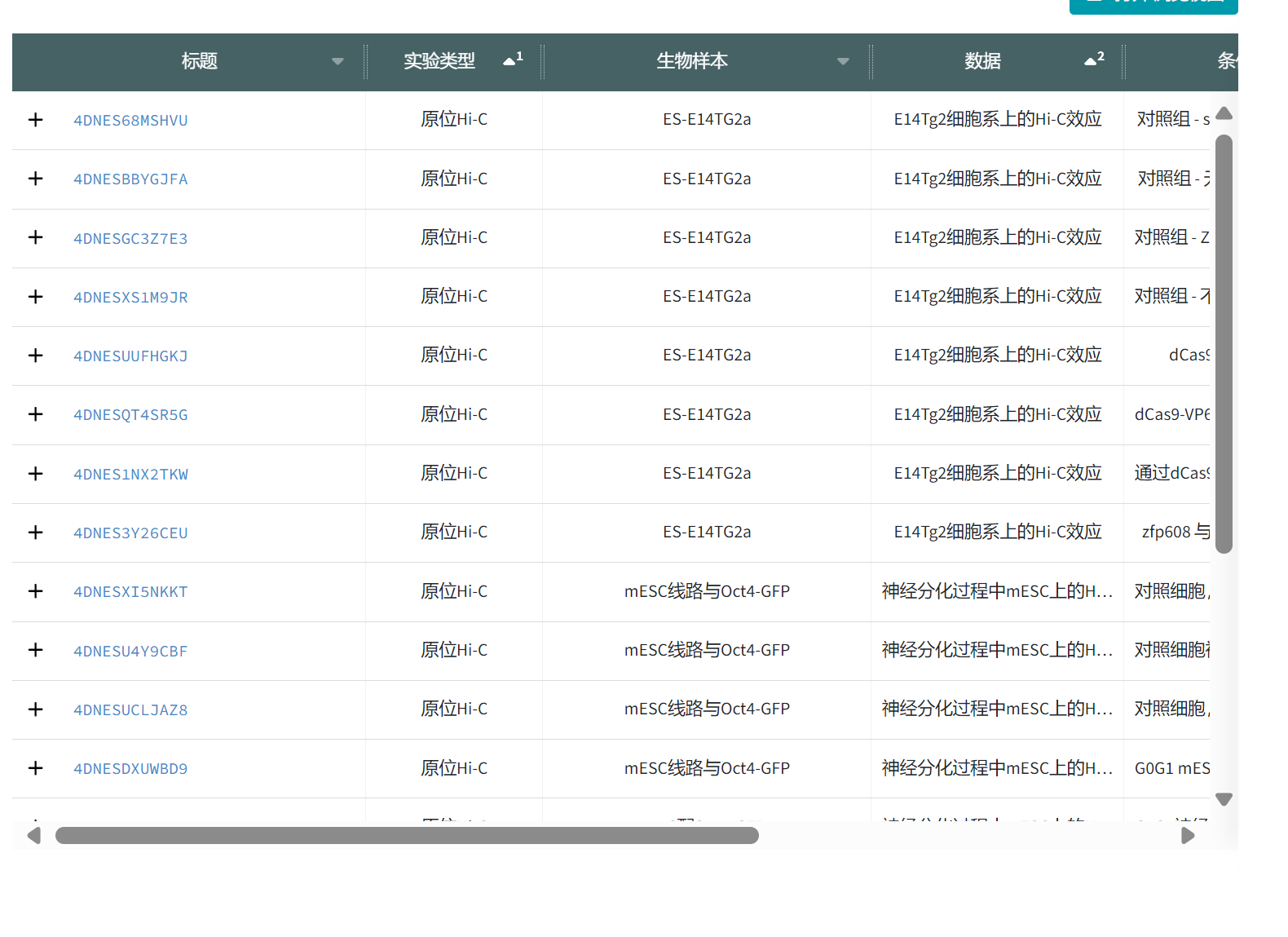
回答:
你现在看到的是 4DN 数据库中所有与 E14TG2a(mES)相关的 in situ Hi-C 数据 ,但 并不是所有这些都对应 SnapHiC 使用的 "Bonev et al. 2017" bulk Hi-C。
其中有很多是:
-
CRISPR 干扰实验(dCas9、VP64 等)
-
转录因子扰动(zfp608、GOG1 等)
-
神经分化相关的 mESC → NPC 时间序列
-
其他实验扰动组
这些都不是你需要的。
你的目标是:
🎯 SnapHiC 使用的 bulk Hi-C = Bonev et al. (Cell 2017) 的原始 wild-type mES in situ Hi-C(无干扰)
你应该下载的 .hic 文件有非常明显的特征:
-
细胞类型:ES-E14TG2a(没问题)
-
实验类型:原位Hi-C(没问题)
-
数据描述:正常的 Hi-C(无 CRISPR / 无干预 / 无时间点)
-
条目名称中通常没有 dCas9 / VP64 / knockout / differentiation / neural / treatment 等字样
-
来源对应 GSE96107 或 Bonev 2017
✅ 我帮你从图中列表筛选 ------ 你需要下载"没有任何干扰"的 E14TG2a Hi-C
列表中你贴出的前几行:
| 文件名 | 实验类型 | 生物样本 | 数据说明 | 是否符合 bulk Hi-C |
|---|---|---|---|---|
| 4DNES68MSHVU | 原位 Hi-C | ES-E14TG2a | E14TG2a 细胞系上的 Hi-C 反应 | ✔ 可能是骨架数据(需继续点开确认) |
| 4DNESBBYGJFA | 原位 Hi-C | ES-E14TG2a | E14TG2a 细胞系上的 Hi-C 反应 | ✔ 可能是骨架数据 |
| 4DNESGC3Z7E3 | 原位 Hi-C | ES-E14TG2a | E14TG2a 细胞系上的 Hi-C 反应 | ✔ 可能是骨架数据 |
| 4DNESXS1M9JR | 原位 Hi-C | ES-E14TG2a | E14TG2a 细胞系上的 Hi-C 反应 | ✔ 可能是骨架数据 |
| 4DNE5UUFHKGJ | 原位 Hi-C | ES-E14TG2a | E14TG2a 细胞系上的 Hi-C 反应(但数据描述含 dCas9) | ❌ 排除------干扰实验 |
再往下你会看到:
| 文件名 | 说明 |
|---|---|
| 4DNESQT4SRS6 | 含 dCas9-VPE → ❌ 排除 |
| 4DNES1NX2TKW | 含 Cas9 → ❌ 排除 |
| 4DNES3Y26CEU | 含 zfp608 → ❌ 排除 |
| 4DNESX1SNKKT | 神经分化 → ❌ 排除 |
| 4DNESU4Y9CBF | 神经分化 → ❌ 排除 |
| 4DNESUCLJAZ8 | 神经分化 → ❌ 排除 |
🎉 结论:你在列表中需要的就是前 4 个
✔ 你应该下载这些四个(全部是 ES-E14TG2a、无干预):
1. 4DNES68MSHVU
2. 4DNESBBYGJFA
3. 4DNESGC3Z7E3
4. 4DNESXS1M9JR
它们最可能对应:
SnapHiC 论文使用的 4 个 replicates bulk in situ Hi-C(Bonev et al. 2017)
你只需要点开每一个,检查:
-
文件格式:是否提供
.hic(有就完美) -
README / Source:如果写 "Bonev et al. 2017" = 100% 对应 SnapHiC 数据
这 4 个合并后就能做你的 APA。
点开第一个,页面信息如下:
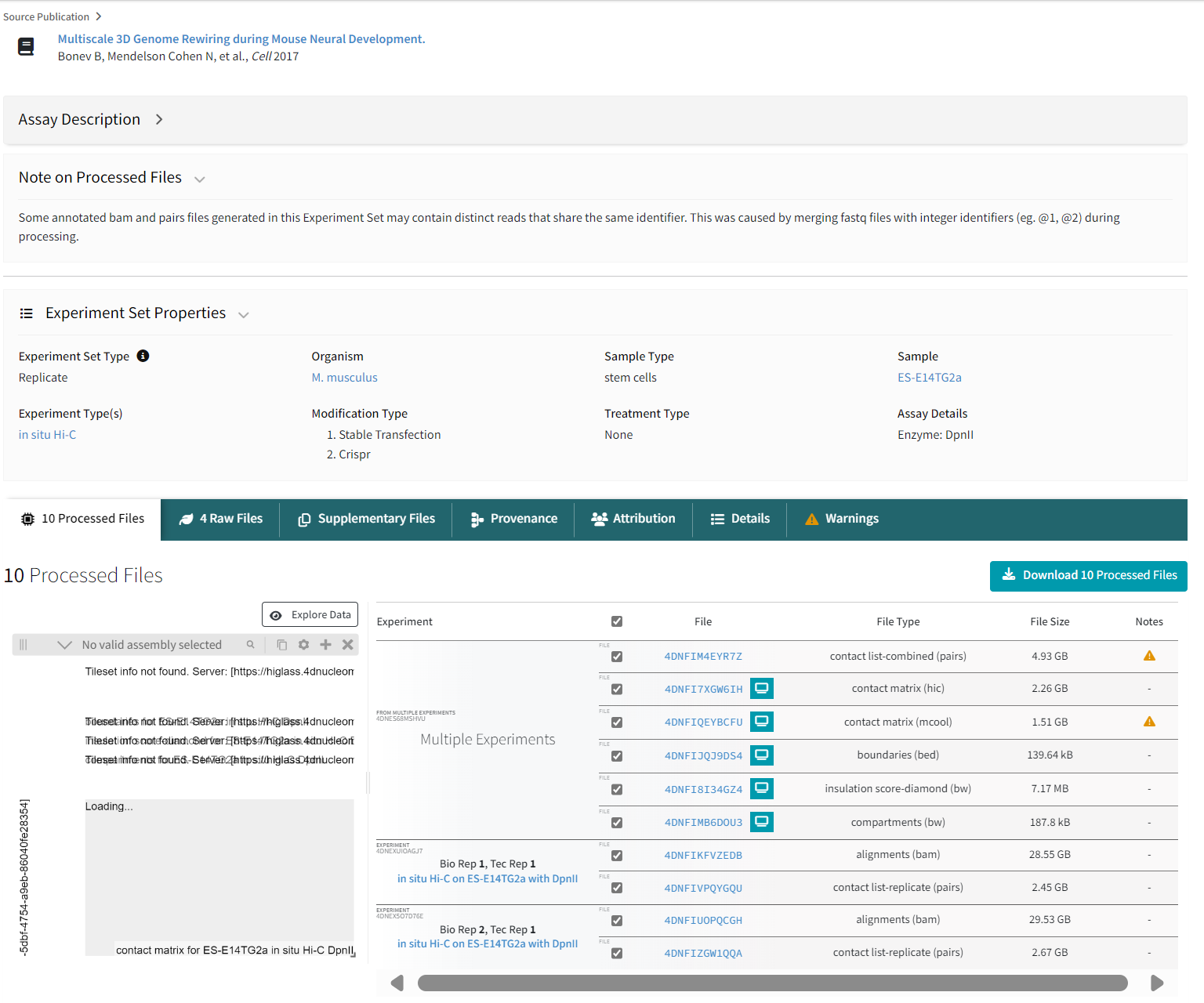
这个 4DN 页面完全确认了它就是 SnapHiC 使用的 Bonev et al. 2017 的 bulk Hi-C 数据 。
你现在需要的是用来做 APA 的 bulk .hic 文件 ,我帮你从这个页面里直接告诉你 ------ 你应该下载哪一个文件。
🎯 你的目标:找到 bulk Hi-C 的 multi-resolution .hic 文件
(即 Juicer 用的 hic 格式)
✅ 从你截图中提取到的 10 个 processed files(我逐条解释哪一个是你需要的)
你截图的文件列表包含:
| 文件名 | 类型 | 是否用于 APA |
|---|---|---|
| 4DNF1ME4YR7Z | pairs(contact list combined) | ❌ 不是 APA 输入 |
| 4DNF7TXW6GIH | contact matrix (.hic) | ✔✔✔ 你需要的 bulk hic 文件(APA 直接用) |
| 4DNF1QEYBCFU | contact matrix (.mcool) | ❌ 不是 Juicer 格式,APA 不支持 |
| 4DNF13Q905D4 | boundaries (bed) | ❌ |
| 4DNF1TB346Z4 | insulation score | ❌ |
| 4DNF1MB6OU3 | compartments | ❌ |
| 4DNF1FXVE20B | bam | ❌ |
| 4DNF1VPQV9CQ | contact list replicate (pairs) | ❌ |
| 4DNF1IQOPCGH | bam | ❌ |
| 4DNF1QM1Q0A | contact list replicate | ❌ |
🎉 结论(非常明确):你需要下载的是这个文件:
✅ 4DNF7TXW6GIH (contact matrix .hic)
➤ File Type: contact matrix (hic)
➤ Size: ~2.26 GB
✔ 完全适合用于 Juicer tools 的 APA 分析
✔ 来自 Bonev et al. 2017
✔ SnapHiC 使用的 bulk Hi-C 就是这套数据
上面页面信息是想下哪一个类型就点进去下载就行,如果想批量下载就选择多个点右上方的蓝色下载键。
这里贴出四个数据集的链接:
4DNES68MSHVU -- 4DN Data Portal
4DNESBBYGJFA -- 4DN Data Portal
4DNESGC3Z7E3 -- 4DN Data Portal
4DNESXS1M9JR -- 4DN Data Portal
然后贴出四个数据集对应的hic类型(也可以从上面的链接点进去直接找就行了)
4DNFI7XGW6IH.hic -- 4DN Data Portal
4DNFIDQ3THN9.hic -- 4DN Data Portal
4DNFI47P5978.hic -- 4DN Data Portal
4DNFIK1EJG8I.hic -- 4DN Data Portal
下面进行apa分析直接将其中一个,或者将四个合并像snaphic那样,集中一起在aoa上做分析。