本期教程
写在前面
今天自己学习一下使用ggNetView包绘制网络图,这个包开发者在去年时候添加过好友。她的绘图功底很厉害,包开发的也全面。这个包是今年10月份上线Github,ggNetView包,网络图绘制,大家应该都比较需要,期待很久!,今年年底事情比较多,从知道上线到目前,自己还未真正的使用此包。因此,也借此机会,一起学习一下。
Github仓库:
R
https://github.com/Jiawang1209/ggNetView包的安装
自己已经快有半年没有认真的是使用本地佮R软件,因此自己的R版本停留在version 4.4版本。自己在安装ggNetView包时一直报错,无法下载安装,也是折腾了很久。
若是你的R版本是在5.0以前的,我们还是建议你进行升级。
因为很多R包,可能在老版本中无法运行。但是,若是你未遇到无法安装R包的情况,我们是不建议你进行升级(具体升不升,视情况而定)。

1. 升级R版本
进入R软件官网,https://www.r-project.org/
最新R版本为4.5.2,,我们选择下载最新版本
2. 下载安装Rtools软件
网址:
R
https://cran.r-project.org/bin/windows/Rtools/
选择自己的适合的版本,正常安装即可。
3. 在Rstudio中选择最新版本的R版本

4. 在新版本R中导入已安装的R包路径
在R语言中如何更改R包安装路径教程中有详细说明。
R
## 我们操作在Rstuido中
## 新建一个初始启动文件
file.edit('~/.Rprofile')
##-------------
## 更改R包默认安装路径
.libPaths("D:/软件安装/R_libaray") ## 输入你的路径
myPaths <- c("D:/软件安装/R_libaray")
.libPaths(myPaths)安装ggNetView包
我自己在安装包中遇到以下问题:
BiocManager包版本太低,需要升级- 提前安装好
SpiecEasi和mascarade包
1. 升级BiocManager包
遇到的报错如下:

安装或升级对应的版本:
R
# install.packages("BiocManager")
BiocManager::install(version = "3.22")
2. 按SpiecEasi和mascarade包
安装SpiecEasi
R
https://github.com/zdk123/SpiecEasi?tab=readme-ov-file#installation安装方式:
R
if (!require("BiocManager", quietly = TRUE)) install.packages("BiocManager")
BiocManager::install("SpiecEasi")我使用此方式无法安装,你可以试一试。
R
if (!require("remotes", quietly = TRUE)) install.packages("remotes")
remotes::install_github("zdk123/SpiecEasi")自己安装成功的命令如下:
R
# install.packages("pak")
pak::pak("zdk123/SpiecEasi")
安装mascarade包
网址:
R
https://github.com/alserglab/mascarade
R
remotes::install_github("alserglab/mascarade")安装ggNetView包
R
# install.packages("pak")
pak::pak("Jiawang1209/ggNetView")
R
library(ggNetView)
- 导入数据
R
data("otu_tab")
data("otu_rare")
# 相关性数据
data("otu_rare_relative")
otu_rare_relative[1:5, 1:5]
## 导入load node annotation
data("tax_tab")- 创建graph
R
obj <- build_graph_from_mat(
mat = otu_rare_relative,
transfrom.method = "none",
method = "WGCNA",
cor.method = "pearson",
proc = "BH",
r.threshold = 0.7,
p.threshold = 0.05,
node_annotation = tax_tab
)
R
> obj[1:5,1:5]
5 x 5 sparse Matrix of class "dgCMatrix"
ASV_916 ASV_777 ASV_606 ASV_740 ASV_1449
ASV_916 . 0.9119817 0.8756662 0.9310804 0.9031801
ASV_777 0.9119817 . 0.8392998 0.8862393 0.8400058
ASV_606 0.8756662 0.8392998 . 0.8876777 0.9306098
ASV_740 0.9310804 0.8862393 0.8876777 . 0.8583368
ASV_1449 0.9031801 0.8400058 0.9306098 0.8583368 .
R
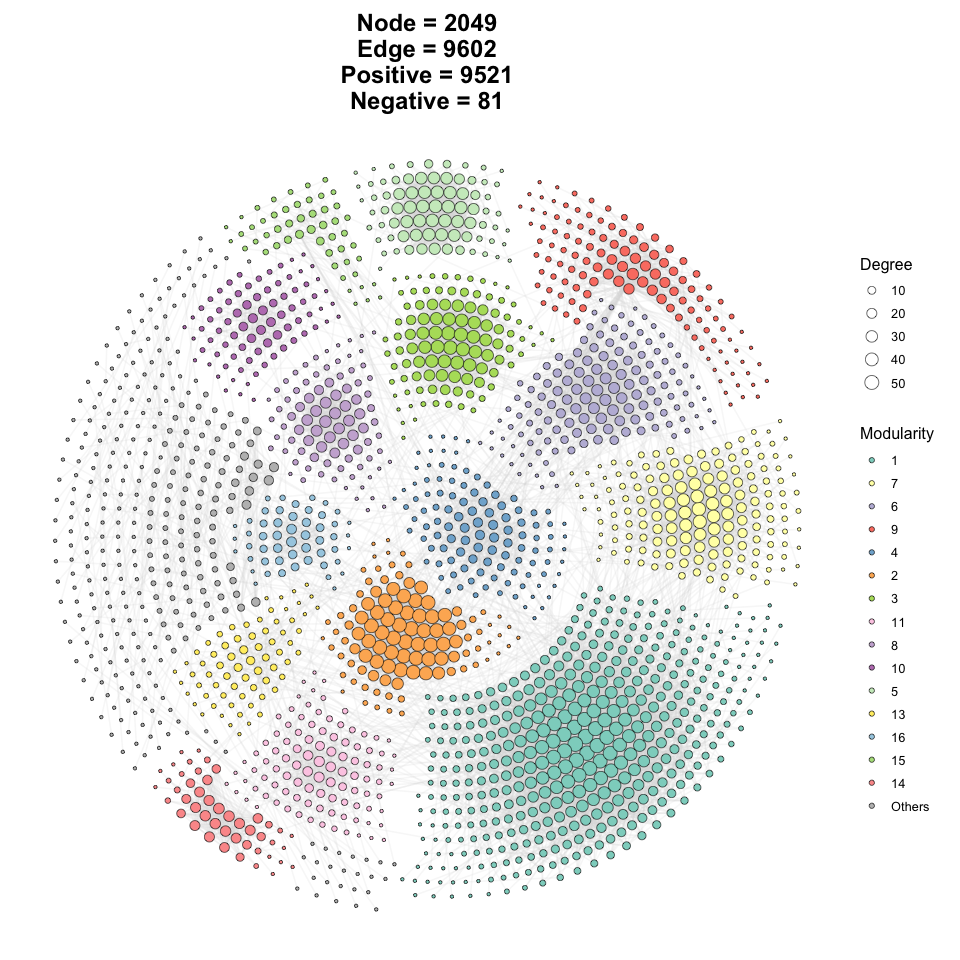
p1 <- ggNetView(
graph_obj = obj,
layout = "gephi",
layout.module = "adjacent",
group.by = "Modularity",
fill.by = "Modularity",
pointsize = c(1, 5),
center = F,
jitter = F,
mapping_line = F,
shrink = 0.9,
linealpha = 0.2,
linecolor = "#d9d9d9"
) 
绘制带有模块信息
R
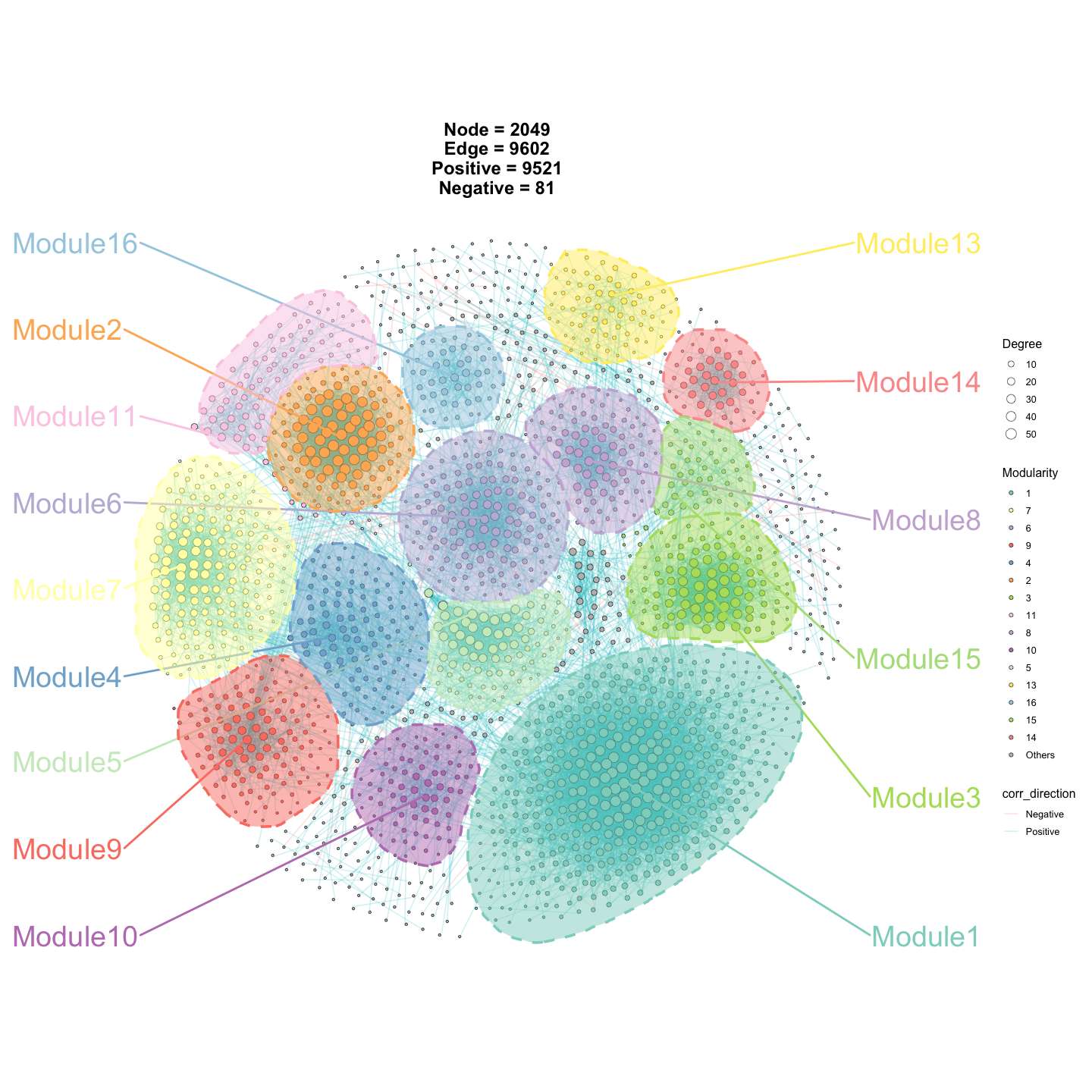
p2 <- ggNetView(
graph_obj = obj,
layout = "gephi",
layout.module = "random",
group.by = "Modularity",
fill.by = "Modularity",
pointsize = c(1, 5),
center = F,
jitter = TRUE,
jitter_sd = 0.15,
mapping_line = TRUE,
shrink = 0.9,
linealpha = 0.2,
linecolor = "#d9d9d9",
add_outer = T,
label = T
) 
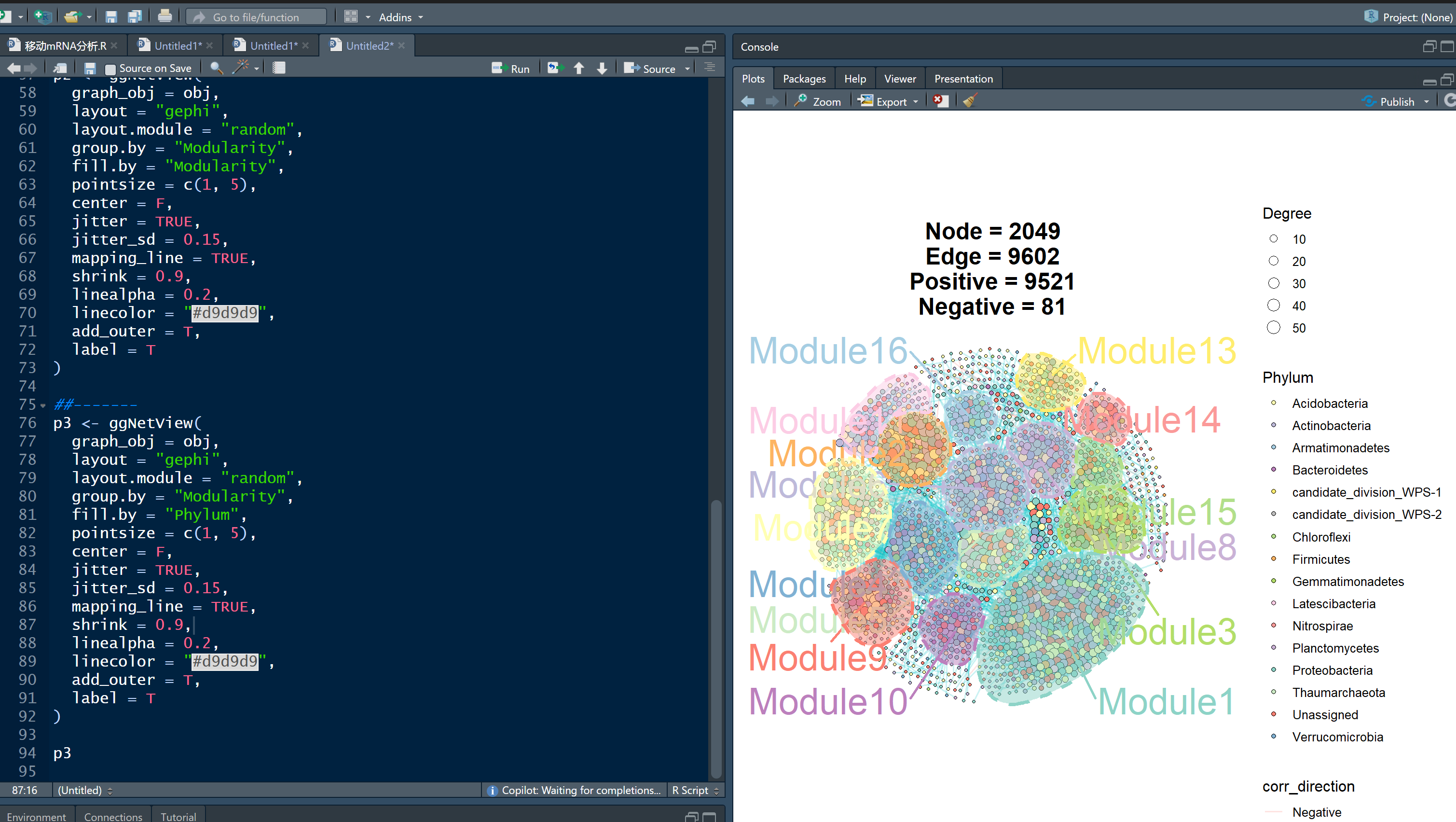
R
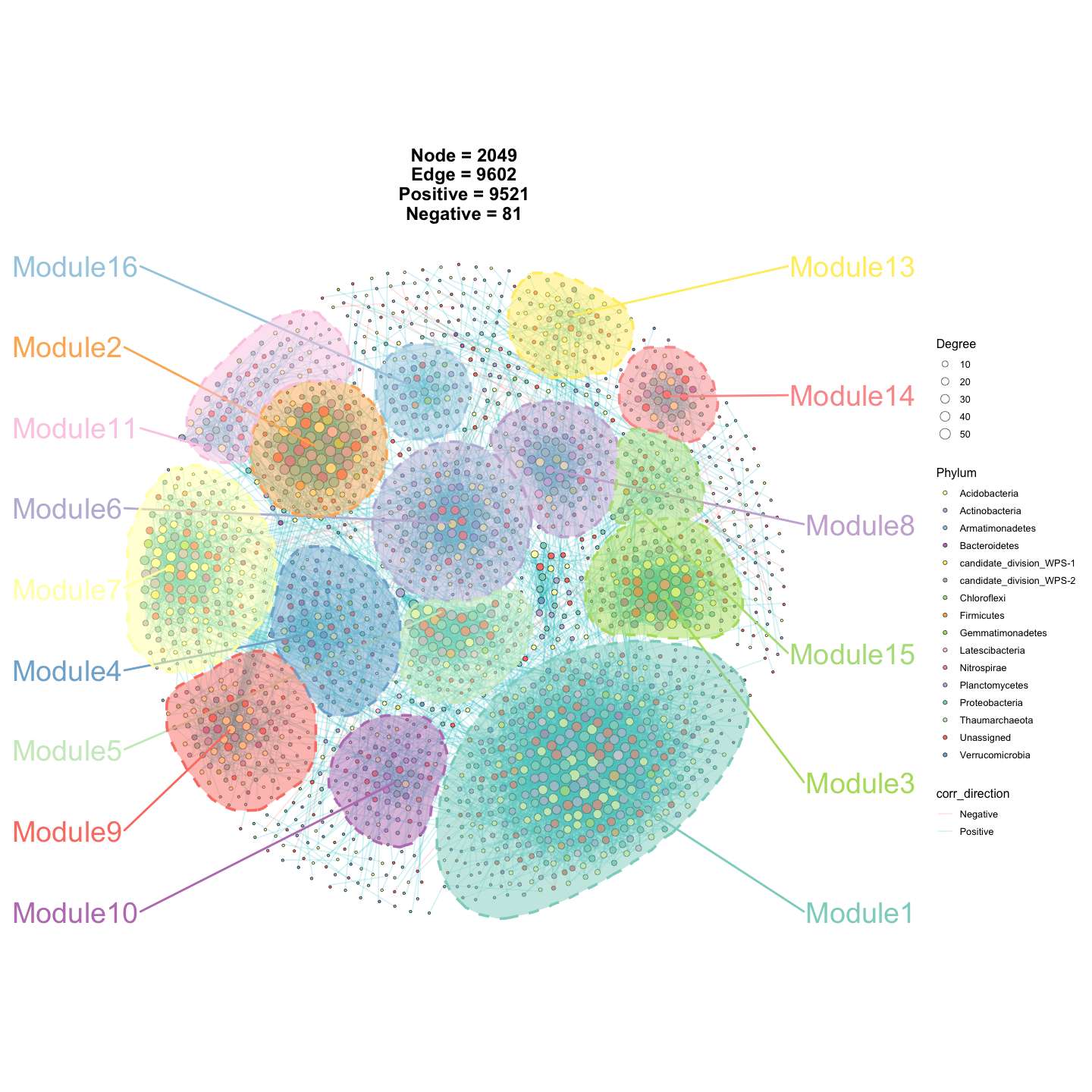
p3 <- ggNetView(
graph_obj = obj,
layout = "gephi",
layout.module = "random",
group.by = "Modularity",
fill.by = "Phylum",
pointsize = c(1, 5),
center = F,
jitter = TRUE,
jitter_sd = 0.15,
mapping_line = TRUE,
shrink = 0.9,
linealpha = 0.2,
linecolor = "#d9d9d9",
add_outer = T,
label = T
) 
2024已离你我而去,2025加油!!
往期部分文章
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
推荐大家购买最新的教程,若是已经购买以前WGNCA教程的同学,可以在对应教程留言,即可获得最新的教程。(注:此教程也仅基于自己理解,不仅局限于此,难免有不恰当地方,请结合自己需求,进行改动。)
2. 精美图形绘制教程
3. 转录组分析教程
4. 转录组下游分析
BioinfoR生信筆記 ,注于分享生物信息学相关知识和R语言绘图教程。