精彩专栏推荐订阅:在下方主页👇🏻👇🏻👇🏻👇🏻
💖🔥作者主页 :计算机毕设木哥🔥 💖
文章目录
- 一、项目介绍
- 二、视频展示
- 三、开发环境
- 四、系统展示
- 五、代码展示
- 六、项目文档展示
- 七、项目总结
- [<font color=#fe2c24 >大家可以帮忙点赞、收藏、关注、评论啦 👇🏻](#fe2c24 >大家可以帮忙点赞、收藏、关注、评论啦 👇🏻)
一、项目介绍
这套系统主要是围绕癌症医疗数据展开的全流程分析平台,核心在于利用大数据技术处理规模较大的临床病例信息,涵盖了从患者人口统计学特征、临床治疗方案到生存预后等多个维度的深度挖掘。系统底层采用Spark作为分布式计算引擎,能够高效处理结构化的癌症数据集,在上层则通过数据可视化技术将复杂的医疗统计结果转化为直观的图表形态,比如不同癌种的地域分布热力图、生存率随时间变化的趋势线等。具体功能模块包括患者年龄性别等基础画像分析、肿瘤分期与治疗方案的关联性挖掘、基于Cox回归的多因素生存风险建模,以及诊断数据的时空模式识别等,整套流程从ETL数据清洗到最终的可视化展示形成了闭环,既满足了医疗数据分析的严谨性要求,又通过大数据技术解决了传统单机处理海量医疗数据时的性能瓶颈。
癌症作为全球性的重大公共卫生挑战,其临床数据呈现出规模大、维度多、关联复杂的典型特征,传统的单机数据分析工具在面对数万甚至数十万的病例记录时往往显得力不从心,处理速度慢且难以支撑实时的交互式分析需求。与此同时,现代肿瘤学研究越来越强调多因素综合评估,需要同时考虑患者的基因突变情况、生活习惯、地域环境以及临床治疗路径等多重变量之间的交互影响,这对数据处理能力提出了更高要求。在这种背景下,运用分布式计算框架来构建癌症数据分析系统就显得尤为必要,它不仅能够突破硬件资源的限制实现海量数据的快速运算,还能通过机器学习算法挖掘出隐藏在庞大医疗数据背后的疾病规律和治疗模式,为临床决策提供更为精准的数据支撑。
做这个系统的实际价值主要体现在几个方面吧。从学习角度来说,算是把大学几年学的大数据技术真正串起来用了一遍,从Spark的RDD操作到DataFrame的SQL查询,再到MLlib的机器学习应用,整个过程下来对分布式计算的理解深了不少,这比单纯看书做题要实在得多。从应用层面看,虽然只是个毕业设计,规模比不上医院里的真实系统,但这种多维度分析的思路对医疗数据管理还是有参考意义的,比如通过可视化展示不同治疗方案的效果差异,能让医学生或者临床医生更直观地理解数据背后的规律。另外这个系统在处理患者隐私数据时也考虑了脱敏和权限控制,算是对医疗数据安全保护的一次实践探索。说到底就是个练手项目,不可能直接拿去给三甲医院用,但在技术整合和问题解决能力上确实让自己进步不少,也算是给大学生活交了个有技术含量的作业。
二、视频展示
三、开发环境
- 大数据技术:Hadoop、Spark、Hive
- 开发技术:Python、Django框架、Vue、Echarts
- 软件工具:Pycharm、DataGrip、Anaconda
- 可视化 工具 Echarts
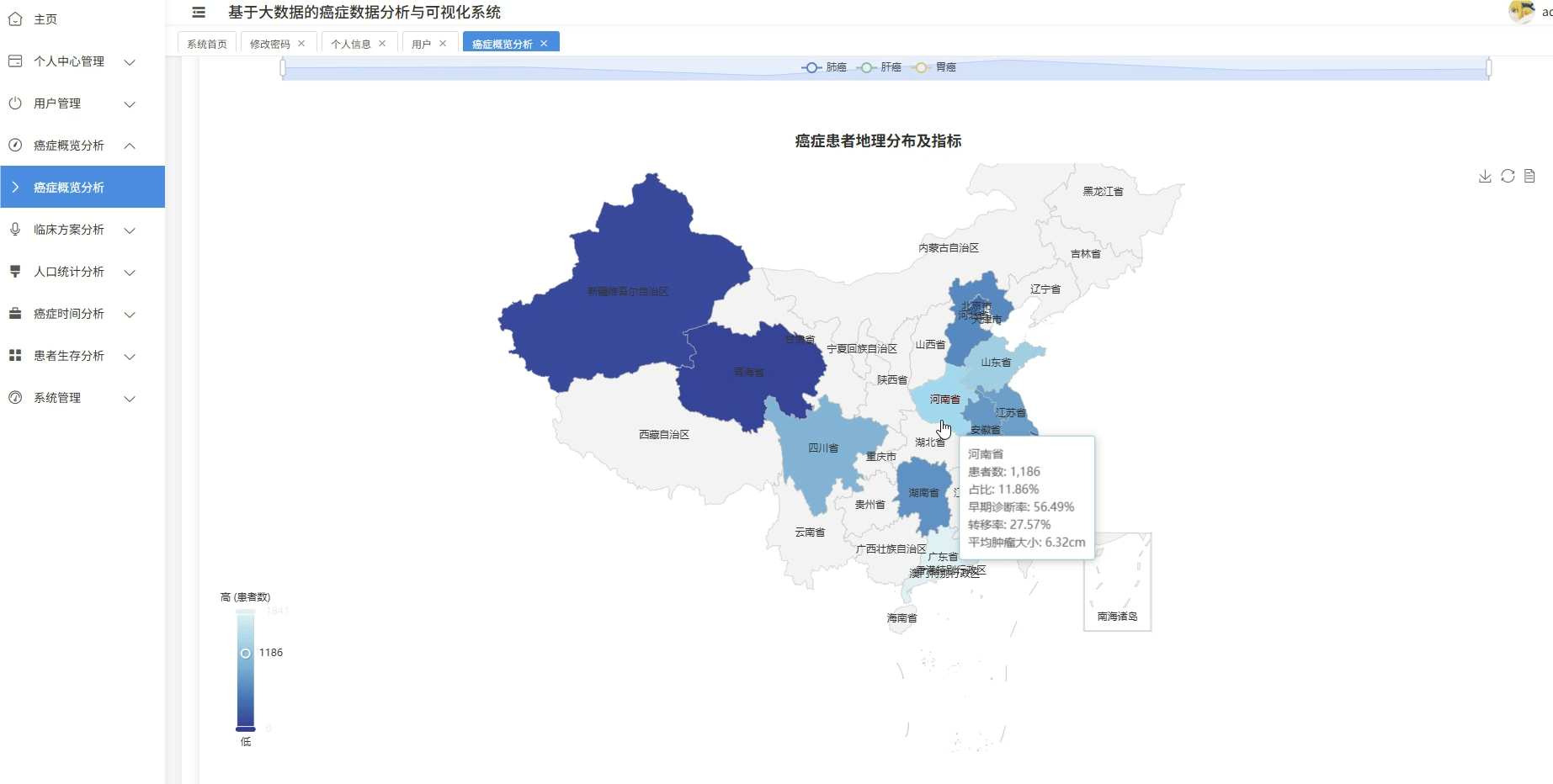
四、系统展示
系统模块展示:
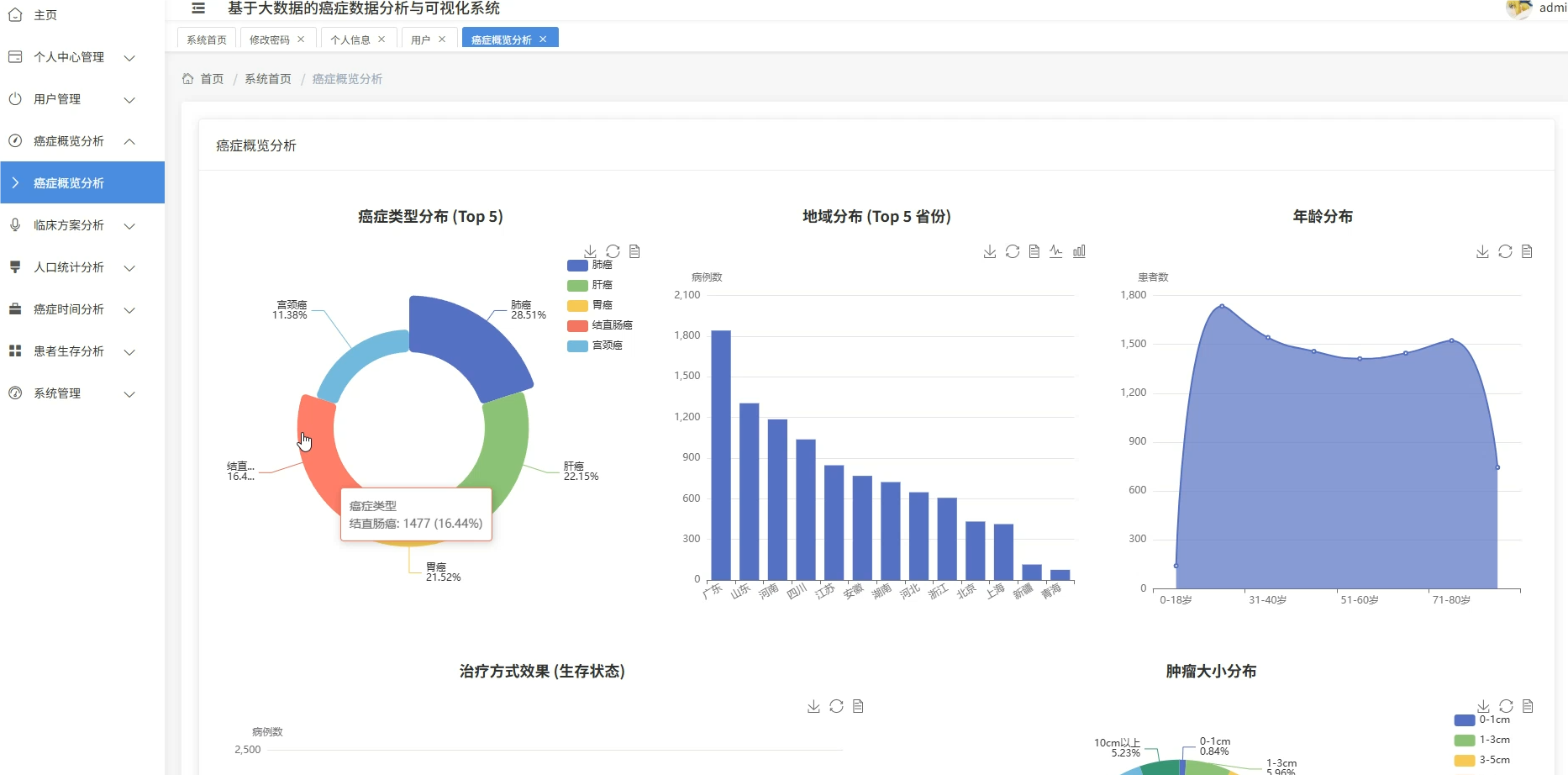
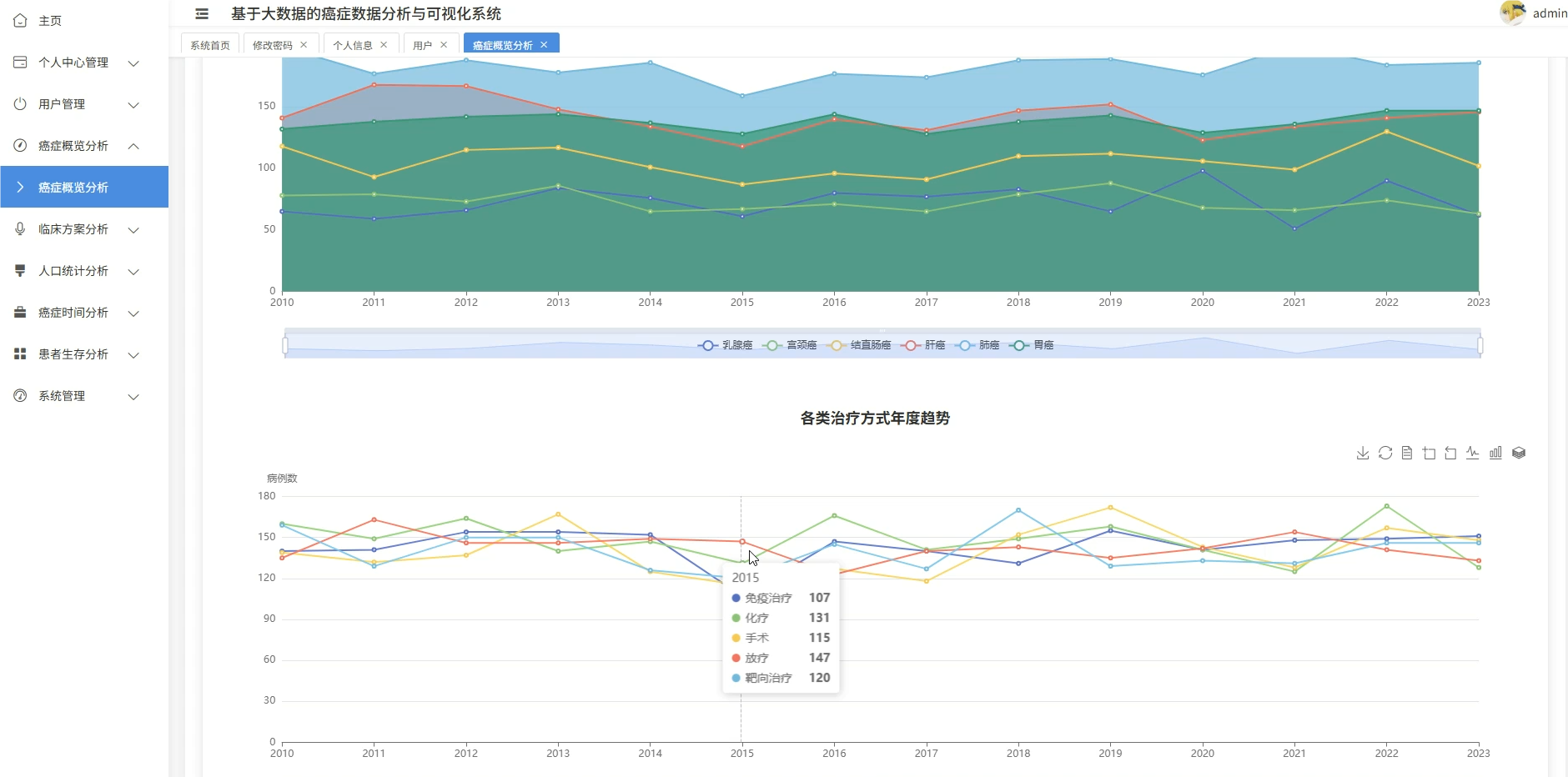
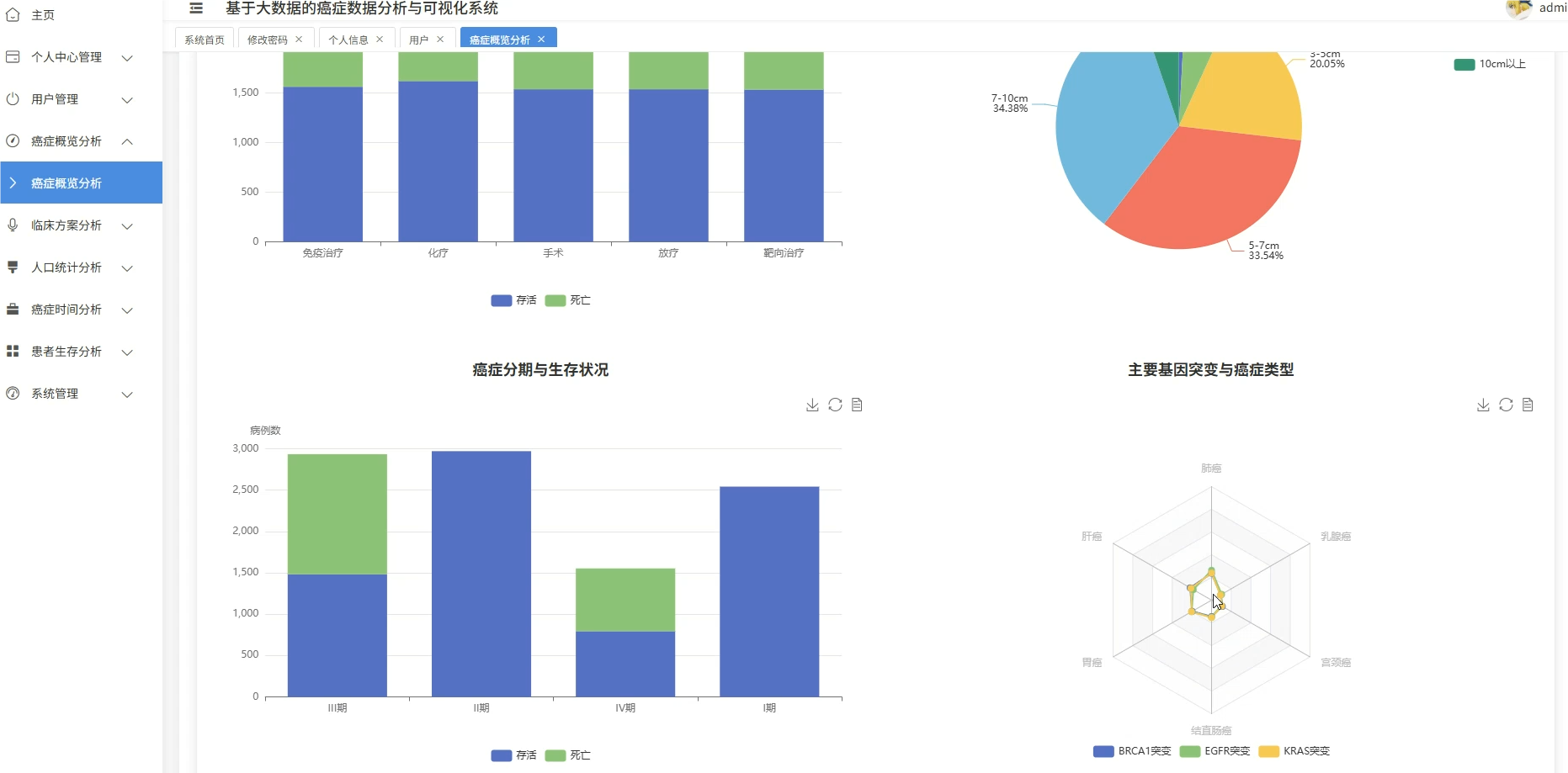
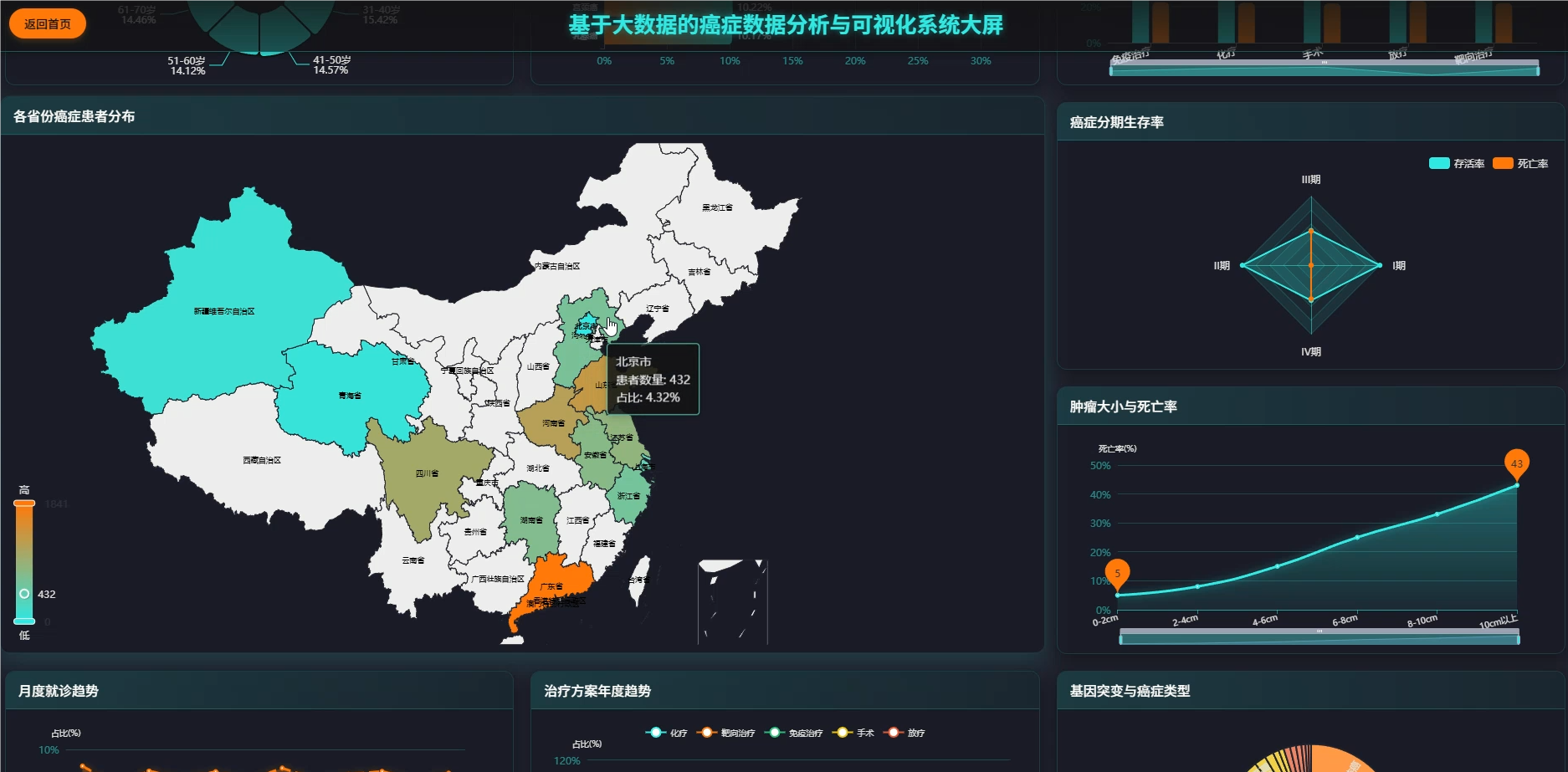
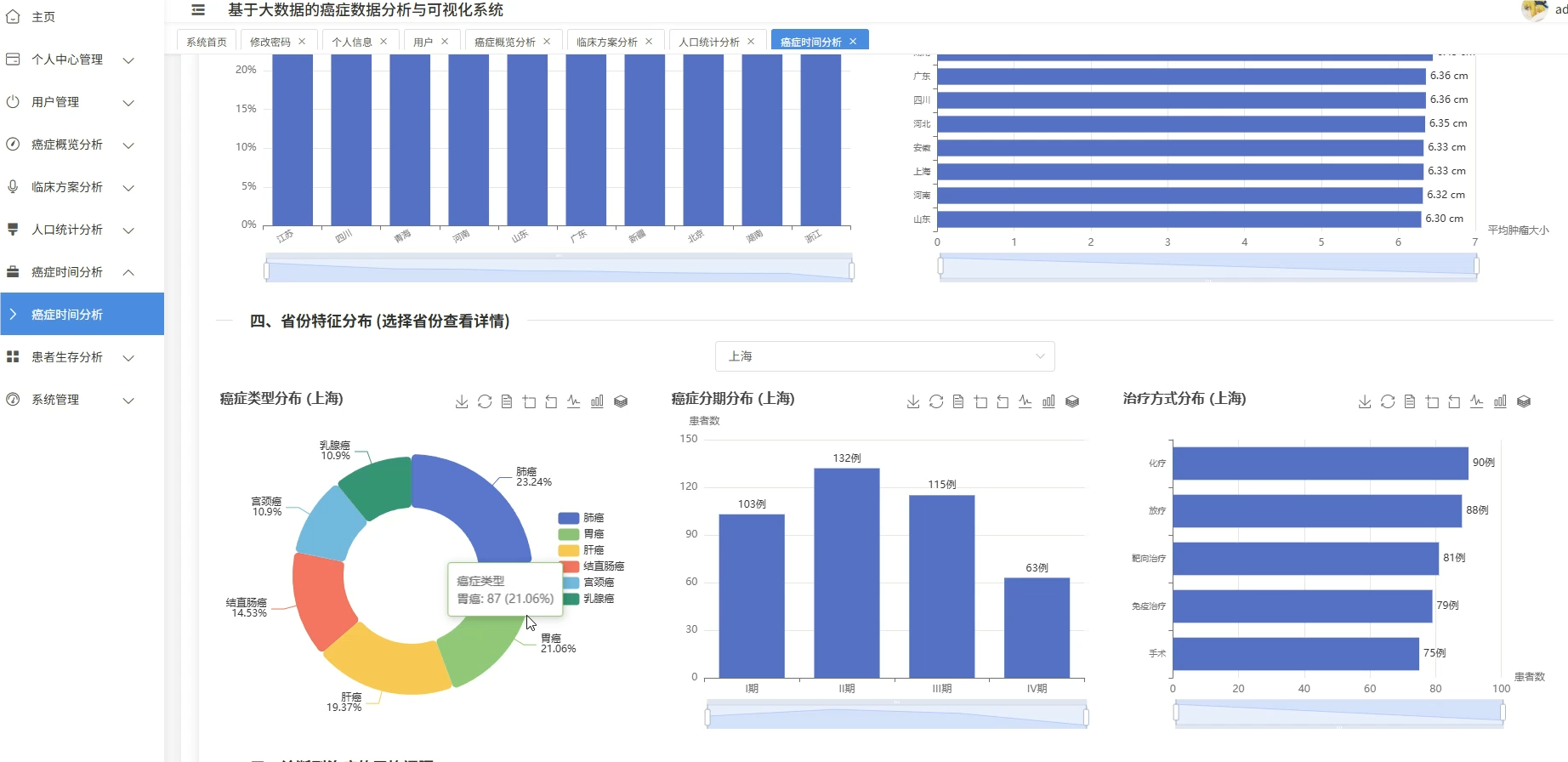
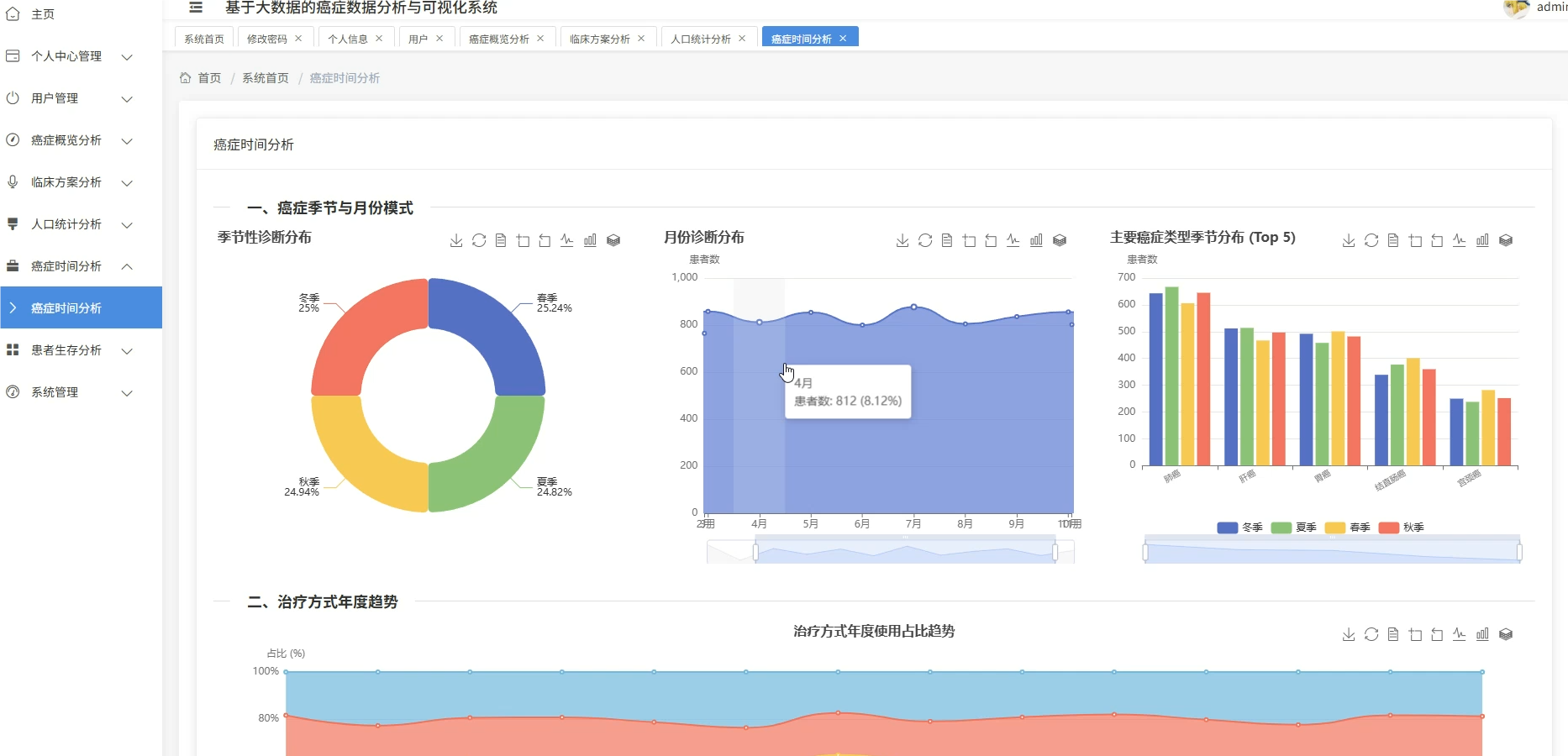
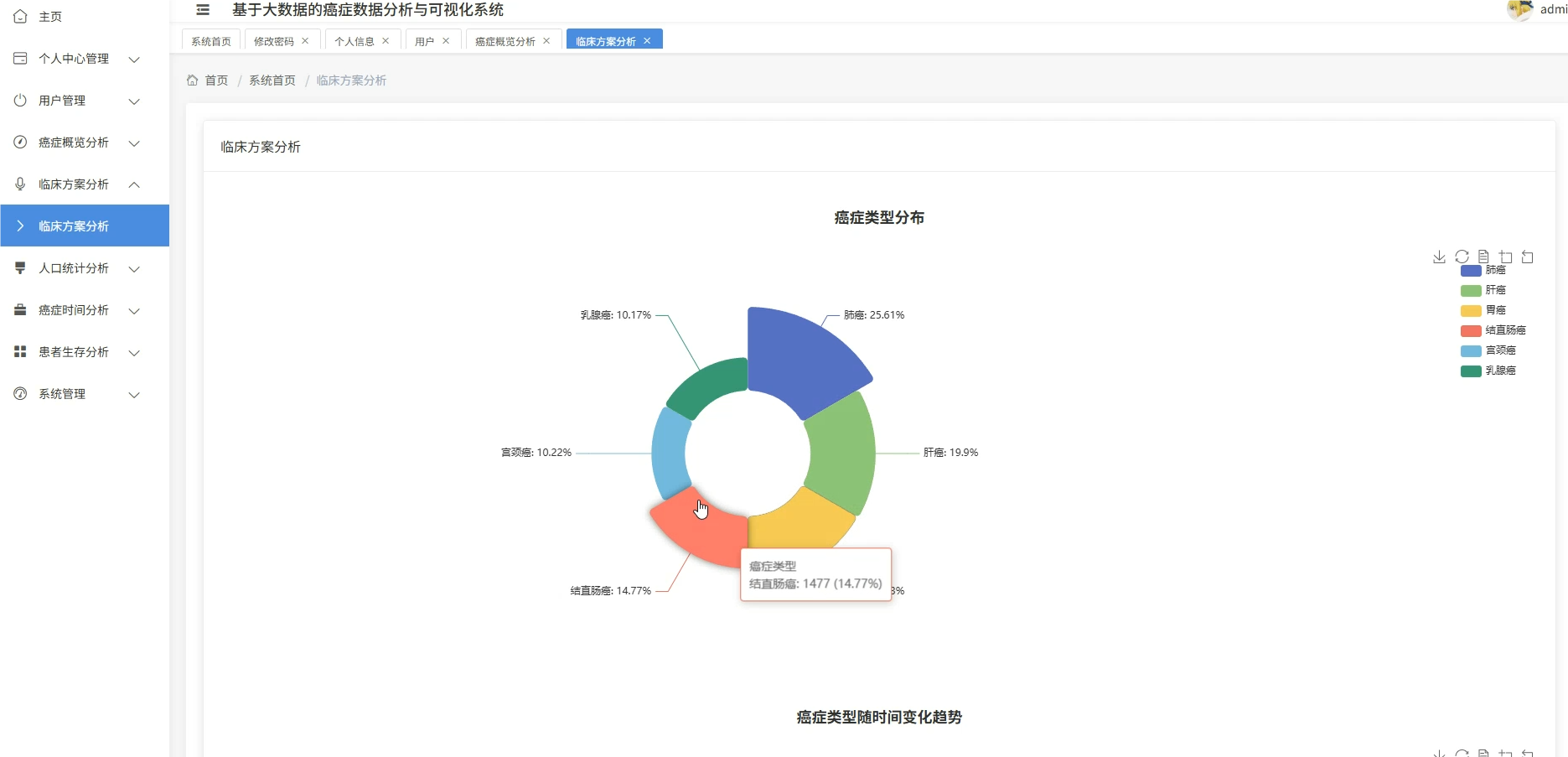
五、代码展示
bash
from pyspark.sql import SparkSession
from pyspark.sql.functions import col, when, count, avg, stddev, lag, expr, row_number, log
from pyspark.ml.feature import VectorAssembler, StringIndexer
from pyspark.ml.classification import LogisticRegression
from pyspark.ml.evaluation import BinaryClassificationEvaluator
from pyspark.sql.window import Window
spark = SparkSession.builder.appName("CancerDataAnalysis").config("spark.sql.adaptive.enabled", "true").config("spark.sql.adaptive.coalescePartitions.enabled", "true").getOrCreate()
df = spark.read.csv("cancer_data.csv", header=True, inferSchema=True)
df = df.filter(col("Age").isNotNull() & col("CancerStage").isNotNull() & col("SurvivalStatus").isNotNull())
df = df.withColumn("AgeGroup", when(col("Age") < 40, 0).when(col("Age") < 60, 1).otherwise(2))
df = df.withColumn("StageNum", when(col("CancerStage") == "I期", 1).when(col("CancerStage") == "II期", 2).when(col("CancerStage") == "III期", 3).otherwise(4))
df = df.withColumn("MetastasisFlag", when(col("Metastasis") == "是", 1).otherwise(0))
df = df.withColumn("SurvivalLabel", when(col("SurvivalStatus") == "存活", 1).otherwise(0))
assembler = VectorAssembler(inputCols=["Age", "StageNum", "MetastasisFlag", "TumorSize"], outputCol="features")
assembled_df = assembler.transform(df)
train_df, test_df = assembled_df.randomSplit([0.8, 0.2], seed=42)
lr = LogisticRegression(labelCol="SurvivalLabel", featuresCol="features", maxIter=10, regParam=0.1)
model = lr.fit(train_df)
predictions = model.transform(test_df)
evaluator = BinaryClassificationEvaluator(labelCol="SurvivalLabel", metricName="areaUnderROC")
auc = evaluator.evaluate(predictions)
predictions.select("Age", "CancerStage", "SurvivalStatus", "probability", "prediction").show(10)
type_stats = df.groupBy("TumorType").agg(count("*").alias("total_cases"), avg("Age").alias("avg_age"), stddev("TumorSize").alias("size_std"))
type_stats = type_stats.withColumn("case_ratio", col("total_cases") / df.count() * 100)
window_spec = Window.partitionBy("TumorType").orderBy("DiagnosisYear")
trend_df = df.groupBy("TumorType", "DiagnosisYear").agg(count("*").alias("yearly_count"))
trend_df = trend_df.withColumn("prev_count", lag("yearly_count", 1).over(window_spec))
trend_df = trend_df.withColumn("growth_rate", when(col("prev_count").isNotNull(), ((col("yearly_count") - col("prev_count")) / col("prev_count") * 100)).otherwise(0))
trend_df = trend_df.join(type_stats, "TumorType")
trend_df.select("TumorType", "DiagnosisYear", "yearly_count", "growth_rate", "avg_age").filter(col("growth_rate") != 0).orderBy(col("growth_rate").desc()).show(15)
treatment_stats = df.groupBy("TreatmentType").agg(
avg("FollowUpMonths").alias("avg_followup"),
count(when(col("SurvivalStatus") == "存活", 1)).alias("survival_cases"),
count("*").alias("total_cases"),
stddev("FollowUpMonths").alias("std_followup")
)
treatment_stats = treatment_stats.withColumn("survival_rate", (col("survival_cases") / col("total_cases")) * 100)
treatment_stats = treatment_stats.withColumn("mortality_rate", 100 - col("survival_rate"))
treatment_stats = treatment_stats.withColumn("effect_score", col("survival_rate") * log(col("avg_followup") + 1))
treatment_stats = treatment_stats.orderBy(col("effect_score").desc())
treatment_stats.select("TreatmentType", "survival_rate", "avg_followup", "effect_score").show()六、项目文档展示

七、项目总结
回过头看整个项目,从最初确定要做癌症数据分析这个方向,到后面一步步用Spark实现各个功能模块,整个过程算是把大数据技术栈完整地实践了一遍。系统虽然不算特别复杂,但基本覆盖了数据采集、分布式处理、机器学习建模和可视化展示这些关键环节,特别是用Spark SQL做数据清洗和用MLlib做生存风险预测这部分,确实体会到了分布式计算在处理医疗大数据时的优势。当然啦,作为毕业设计,这个系统还有很多可以改进的地方,比如可以考虑引入更复杂的深度学习模型来做图像分析,或者接入实时数据流处理功能,但在当前阶段算是达到了预期的学习目标。通过做这个课题,不仅对医疗数据分析的业务逻辑有了更深的理解,更重要的是掌握了如何用技术手段去解决实际问题,这种从需求分析到代码实现的完整经历,对自己以后从事数据开发工作还是很有帮助的。
大家可以帮忙点赞、收藏、关注、评论啦 👇🏻
💖🔥作者主页 :计算机毕设木哥🔥 💖