A Multi-scale Linear-time Encoder for Whole-Slide Image Analysis
Authors: Jagan Mohan Reddy Dwarampudi, Joshua Wong, Hien Van Nguyen, Tania Banerjee
Deep-Dive Summary:
用于全扫描切片图像分析的多尺度线性时间编码器 (MARBLE)
摘要
我们引入了多尺度自适应循环生物医学线性时间编码器(MARBLE),这是首个纯基于 Mamba 的多状态多实例学习(MIL)框架,专门用于全扫描切片图像(WSI)分析。MARBLE 并行处理多个放大倍率级别,并在线性时间状态空间模型中集成了从粗到细的推理,能够以最小的参数开销高效捕获跨尺度依赖关系。WSI 分析由于其吉像素分辨率和分层放大倍率而具有挑战性,而现有的 MIL 方法通常在单一尺度上运行,且基于 Transformer 的方法面临二次方注意力成本。通过将并行多尺度处理与线性时间序列建模相结合,MARBLE 为基于注意力的架构提供了一个可扩展且模块化的替代方案。在五个公开数据集上的实验表明,其在 AUC 上提升高达 6.9 % 6.9\% 6.9%,准确率提升 20.3 % 20.3\% 20.3%,C-index 提升 2.3 % 2.3\% 2.3%,确立了 MARBLE 作为多尺度 WSI 分析的高效且通用框架的地位。
1. 引言
多实例学习(MIL)是计算病理学中处理吉像素全扫描切片图像的核心范式。一张切片被表示为一个包含多个图像块(patches)的"包"(bag),且仅具有切片级的标签。早期的 MIL 使用最大或平均池化,后来的基于注意力的池化突出了显著区域,但仍将图像块视为独立同分布的,限制了长程空间上下文的建模。基于 Transformer 的 MIL 引入了对块嵌入的自注意力机制,分层视觉 Transformer(如 HIPT)通过金字塔结构保留了更广泛的上下文。多尺度 MIL(如 HAG-MIL 和 CS-MIL)通过分层注意力或语义过滤融合了不同放大倍率的信息。与此同时,结构化状态空间模型(SSM)实现了高效的长序列建模,例如 S4 模型提供线性时间动力学,而基于 Mamba 的变体(如 MambaMIL)已将其应用于病理学。
尽管取得了这些进展,但仍然缺乏高效且可扩展的显式多尺度推理。首先,许多流程为了稳定性和效率仅处理单一放大倍率,未利用跨尺度依赖;其次,Transformer 的多尺度融合往往导致二次方注意力成本或阻碍端到端训练的启发式方法;第三,现有的 Mamba 方法通常将切片视为单尺度序列。MARBLE 旨在统一解决这些效率、可扩展性和上下文问题。
主要贡献:
- 提出了首个全 Mamba 的 MIL 框架,引入了具有显式从粗到细融合的多尺度编码。
- 提出了一种轻量级的、标记对齐的跨级别调节机制,将粗粒度上下文信息注入细粒度表示。
- 在分类和生存分析的五个公开数据集上取得了一致的收益。
2. 方法
WSI 在 S + 1 S + 1 S+1 个放大级别提供,索引为 k ∈ { 0 , 1 , ... , S } k \in \{0, 1, \ldots , S\} k∈{0,1,...,S}, k = 0 k = 0 k=0 为最粗级别, k = S k = S k=S 为最细级别。在级别 k k k,我们将组织区域划分为非重叠的 P × P P \times P P×P 图像块,并提取 D D D 维嵌入 X ( k ) = [ x 1 ( k ) , ... , x T ( k ) ] ⊤ \mathbf{X}^{(k)} = [\mathbf{x}_1^{(k)}, \ldots , \mathbf{x}_T^{(k)}]^\top X(k)=[x1(k),...,xT(k)]⊤。
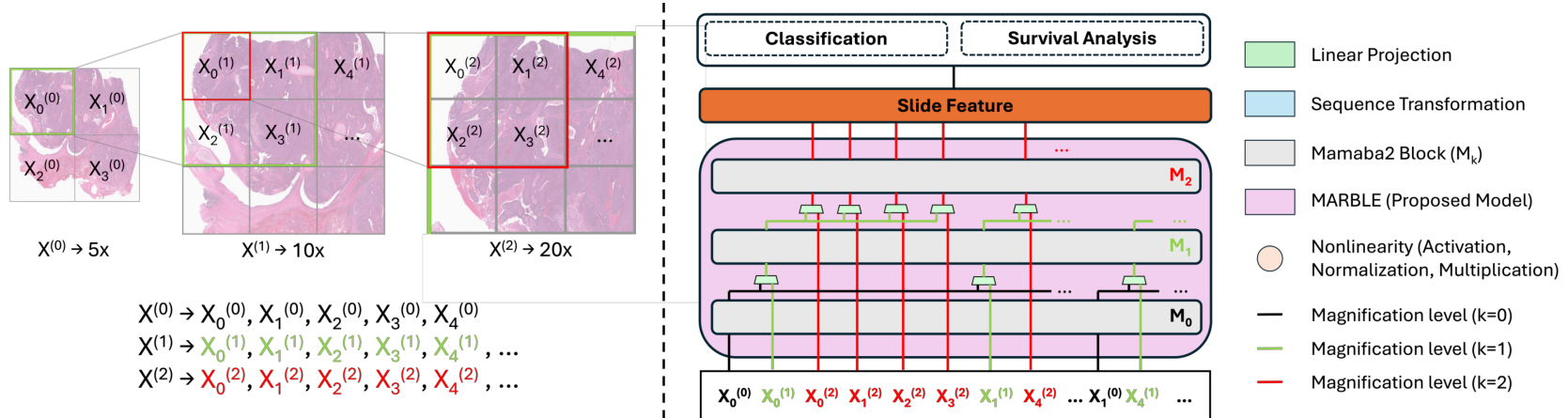
2.1 具有从粗到细融合的多尺度编码
每个级别由一个独立的序列模块进行编码,相对于标记数 T k T_k Tk 呈线性时间复杂度。每个级别编码器使用深度为 L = 1 L=1 L=1、维度 D = 1024 D=1024 D=1024 的 Mamba-2 块。在编码级别 k > 0 k > 0 k>0 之前,每个细标记会结合从级别 k − 1 k-1 k−1 提取的父级上下文进行增强:
c i ( k ) = y p k ( i ) ( k − 1 ) , x ~ i ( k ) = ϕ ( k ) ( [ x i ( k ) ∥ c i ( k ) ] ) ∈ R D , \mathbf{c}{i}^{(k)} = \mathbf{y}{p_{k}(i)}^{(k - 1)},\qquad \tilde{\mathbf{x}}{i}^{(k)} = \phi^{(k)}\big([\mathbf{x}{i}^{(k)}\parallel \mathbf{c}_{i}^{(k)}]\big)\in \mathbb{R}^{D}, ci(k)=ypk(i)(k−1),x~i(k)=ϕ(k)([xi(k)∥ci(k)])∈RD,
其中 ϕ ( k ) \phi^{(k)} ϕ(k) 是线性投影, p k ( i ) p_k(i) pk(i) 是通过空间平铺网格确定的父子映射关系。这种设计保持了线性复杂度 O ( T k D ) \mathcal{O}(T_k D) O(TkD)。
2.2 切片级预测头
分类: 仅对最细级别的表示进行注意力池化:
S = { y i ( S ) } i = 1 T S , \mathcal{S} = \{\mathbf{y}{i}^{(S)}\}{i = 1}^{T_{S}}, S={yi(S)}i=1TS,
α ( y ) = exp ( w ⊤ y ) ∑ y ′ ∈ S exp ( w ⊤ y ′ ) , z = ∑ y ∈ S α ( y ) y . \alpha (\mathbf{y}) = \frac{\exp(\mathbf{w}^{\top}\mathbf{y})}{\sum_{\mathbf{y}^{\prime}\in \mathcal{S}}\exp(\mathbf{w}^{\top}\mathbf{y}^{\prime})},\quad \mathbf{z} = \sum_{\mathbf{y}\in \mathcal{S}}\alpha (\mathbf{y})\mathbf{y}. α(y)=∑y′∈Sexp(w⊤y′)exp(w⊤y),z=y∈S∑α(y)y.
随后使用线性分类器映射到逻辑值并优化交叉熵。
生存分析: 在 z \mathbf{z} z 上附加 Cox 比例风险头以产生风险分数 r i = β ⊤ z i r_i = \beta^{\top} \mathbf{z}{i} ri=β⊤zi,并最小化负偏对数似然:
L C o x = − ∑ i : δ i = 1 ( r i − log ∑ j ∈ R ( t i ) e r j ) + λ ∥ θ ∥ 2 2 , \mathcal{L}{\mathrm{Cox}} = -\sum_{i:\delta_{i} = 1}\left(r_{i} - \log \sum_{j\in \mathcal{R}(t_{i})}e^{r_{j}}\right) + \lambda \| \pmb {\theta}\|_{2}^{2}, LCox=−i:δi=1∑ ri−logj∈R(ti)∑erj +λ∥θ∥22,
2.3 正则化与鲁棒性
我们应用了两种正则化方法:
- 随机粗路径丢弃: 在训练期间随机丢弃一部分 k = 0 k=0 k=0 级别的标记及其对应的所有子孙标记,创建随机子包。
- 扫描顺序中性: 在每个级别内随机打乱标记顺序,以防止隐含的位置偏差,保持排列不变性。
2.4 数据集与基准模型
我们在诊断分类任务(PANDA 和 TCGA-NSCLC)以及生存分析任务(KIRP, LUAD, STAD)上进行了评估。所有图像块嵌入均使用预训练的 UNI 模型提取。对比基准包括 ABMIL, CLAM, TransMIL, MambaMIL 等。
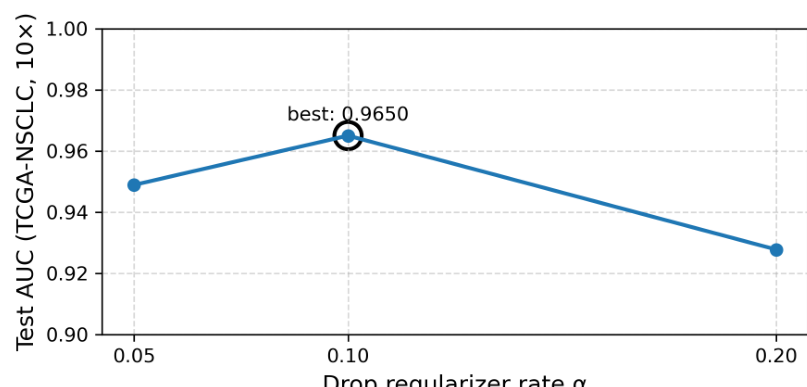
图 2:丢弃正则化率 α \alpha α 对 TCGA-NSCLC 测试 AUC 的影响。 α = 0.1 \alpha = 0.1 α=0.1 取得了最佳平衡。
3. 结果
分类结果: MARBLE 一致优于强 MIL 基准。在 PANDA 上,其准确率和 AUC 分别比最强竞争对手高出 20.25 20.25 20.25 个百分点和 6.94 6.94 6.94 个百分点。在 TCGA-NSCLC 上也表现出竞争力(见表 1)。
表 1:两个数据集上的分类性能。
| Method | PANDA Acc | PANDA AUC | TCGA-NSCLC Acc | TCGA-NSCLC AUC |
|---|---|---|---|---|
| AB-MIL | 0.4883 | 0.7797 | 0.8758 | 0.9572 |
| TransMIL | 0.4636 | 0.7728 | 0.8850 | 0.9626 |
| 2DMambaMIL | 0.5075 | 0.8184 | 0.8851 | 0.9618 |
| MARBLE (Ours) | 0.7100 | 0.8878 | 0.8966 | 0.9730 |
生存分析结果: 在 KIRP、LUAD 和 STAD 数据集上,MARBLE 均获得了最高的 C-index(见表 2)。
表 2:三个 TCGA 数据集的生存分析 C-index 比较。
| Method | KIRP | LUAD | STAD |
|---|---|---|---|
| ABMIL | 0.7824 | 0.6157 | 0.6119 |
| MambaMIL | 0.7822 | 0.5952 | 0.6244 |
| MARBLE (Ours) | 0.8184 | 0.6432 | 0.6510 |
消融研究: 表 3 显示,结合 10 × 10\times 10× 和 40 × 40\times 40× 两种分辨率的表现优于任何单一分辨率。
表 3:多分辨率消融结果。
| Method | NSCLC Acc | NSCLC AUC | STAD C-index |
|---|---|---|---|
| MARBLE (10x) | 0.8851 | 0.9650 | 0.6141 |
| MARBLE (40x) | 0.8736 | 0.9719 | 0.5961 |
| MARBLE (10x, 40x) | 0.8966 | 0.9730 | 0.6510 |
4. 结论
我们提出了 MARBLE,这是一种多实例学习框架,在线性时间状态空间主干网络上执行具有轻量级从粗到细融合的并行多倍率编码。通过在粗级父节点上调节细级标记,MARBLE 在不产生二次方注意力开销和极小参数增加的情况下捕获了跨尺度依赖。实验证明,MARBLE 显著提升了切片级任务的性能,且消融实验证实了多尺度融合的有效性。
Original Abstract: We introduce Multi-scale Adaptive Recurrent Biomedical Linear-time Encoder (MARBLE), the first \textit{purely Mamba-based} multi-state multiple instance learning (MIL) framework for whole-slide image (WSI) analysis. MARBLE processes multiple magnification levels in parallel and integrates coarse-to-fine reasoning within a linear-time state-space model, efficiently capturing cross-scale dependencies with minimal parameter overhead. WSI analysis remains challenging due to gigapixel resolutions and hierarchical magnifications, while existing MIL methods typically operate at a single scale and transformer-based approaches suffer from quadratic attention costs. By coupling parallel multi-scale processing with linear-time sequence modeling, MARBLE provides a scalable and modular alternative to attention-based architectures. Experiments on five public datasets show improvements of up to \textbf{6.9%} in AUC, \textbf{20.3%} in accuracy, and \textbf{2.3%} in C-index, establishing MARBLE as an efficient and generalizable framework for multi-scale WSI analysis.
PDF Link: 2602.02918v1
部分平台可能图片显示异常,请以我的博客内容为准
