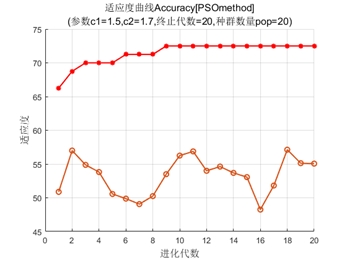
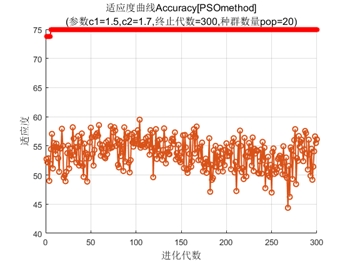
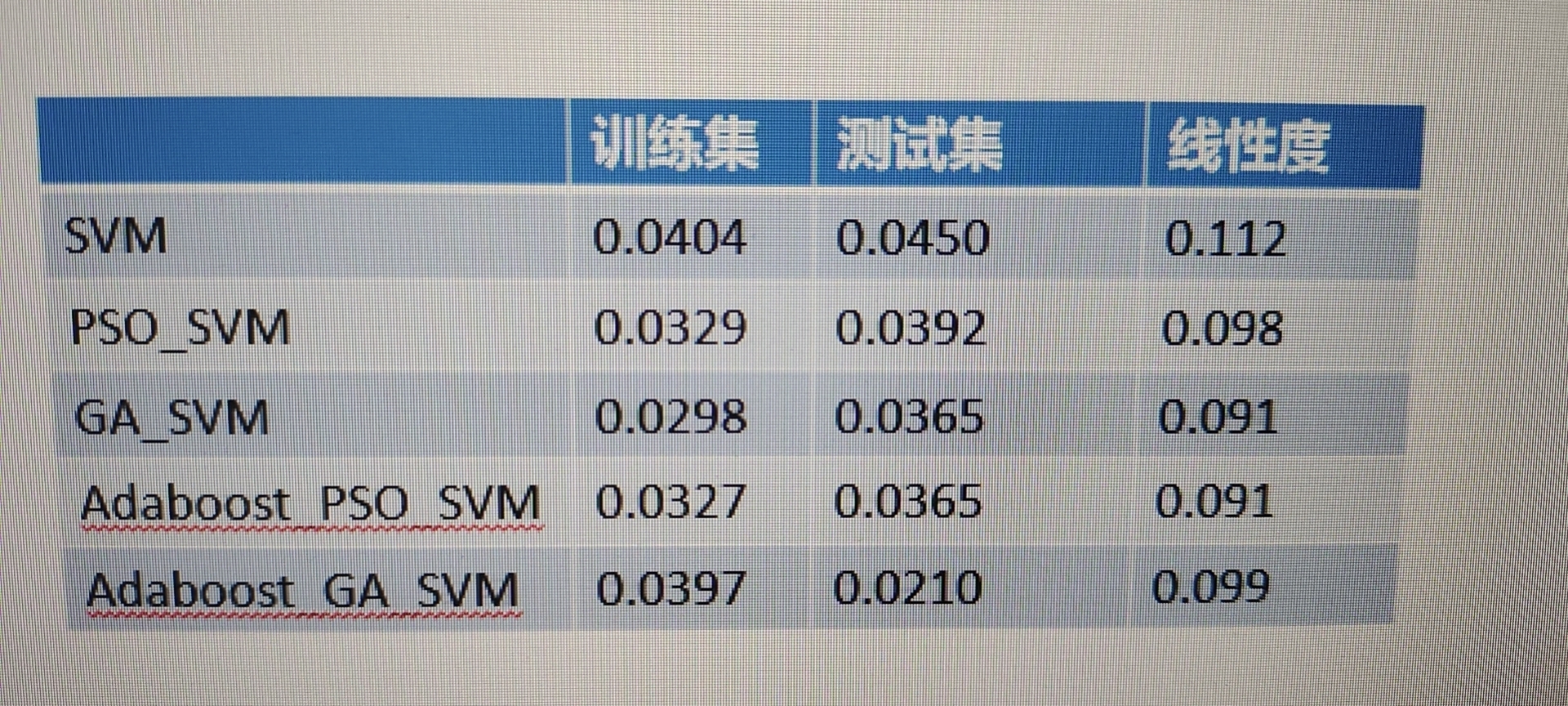
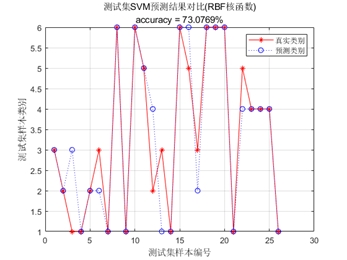
粒子群优化支持向量机 遗传算法优化支持向量机 网格搜索 遍历法 PSO-SVM GA-SVM 。 用liv-SVM工具箱,选择较好的C和G。 简单容易上手,替换数据即可,有代码解释。
在机器学习的世界里,支持向量机(SVM)以其出色的分类和回归能力备受青睐。然而,要让 SVM 发挥最佳性能,参数的选择至关重要。今天咱们就来唠唠用粒子群优化(PSO)、遗传算法(GA)以及网格搜索遍历法来优化 SVM 参数,并且使用 LIBSVM 工具箱选择较好的 C 和 G 参数。
LIBSVM 工具箱简介
LIBSVM 是一个简单易用且高效的 SVM 模式识别与回归软件包。它涵盖了多种 SVM 类型,为我们进行 SVM 模型构建提供了极大便利。在使用它之前,确保你已经安装并配置好环境。
网格搜索遍历法
网格搜索遍历法算是最直观的参数调优方法啦。它通过遍历预先设定的参数值组合,穷举所有可能性,从中找到最优的参数组合。下面来看段简单代码:
matlab
% 加载数据
load heart_scale;
x = heart_scale_inst;
y = heart_scale_label;
% 设置参数范围
c_range = 2.^(-5:2:15);
g_range = 2.^(-15:2:3);
best_acc = 0;
best_c = 0;
best_g = 0;
% 遍历所有参数组合
for i = 1:length(c_range)
for j = 1:length(g_range)
cmd = ['-v 5 -c ', num2str(c_range(i)),'-g ', num2str(g_range(j))];
acc = svmtrain(y, x, cmd);
if acc(1) > best_acc
best_acc = acc(1);
best_c = c_range(i);
best_g = g_range(j);
end
end
end
fprintf('Best C: %f, Best G: %f, Best Accuracy: %f\n', best_c, best_g, best_acc);代码分析:
- 首先加载了
heart_scale数据集,x是特征数据,y是标签数据。 - 设定
C和G的取值范围,C是惩罚参数,G是核函数参数。这里通过指数形式设定了一系列值。 - 使用两层循环遍历所有
C和G的组合。在每次循环中,构建 LIBSVM 的命令字符串cmd,其中-v 5表示进行 5 折交叉验证,-c后跟C的值,-g后跟G的值。 - 调用
svmtrain函数进行训练并获取交叉验证准确率acc。如果当前组合的准确率高于之前记录的最佳准确率,就更新最佳准确率、最佳C和最佳G。 - 最后输出找到的最佳
C、G和准确率。
粒子群优化支持向量机(PSO - SVM)
粒子群优化算法模拟鸟群觅食行为,通过粒子之间的协作与竞争找到最优解。每个粒子都有自己的位置和速度,在解空间中不断搜索。
matlab
% 适应度函数定义
function fitness = pso_svm_fitness(params, x, y)
c = params(1);
g = params(2);
cmd = ['-v 5 -c ', num2str(c),'-g ', num2str(g)];
fitness = -svmtrain(y, x, cmd)(1);
end
% 粒子群优化参数设置
n_particles = 20;
n_dimensions = 2;
max_iterations = 100;
c1 = 1.5;
c2 = 1.5;
w = 0.7;
% 初始化粒子位置和速度
particles_pos = rand(n_particles, n_dimensions);
particles_vel = rand(n_particles, n_dimensions);
pbest_pos = particles_pos;
pbest_fitness = inf(n_particles, 1);
gbest_pos = [];
gbest_fitness = inf;
% 加载数据
load heart_scale;
x = heart_scale_inst;
y = heart_scale_label;
for iter = 1:max_iterations
for i = 1:n_particles
fitness = pso_svm_fitness(particles_pos(i, :), x, y);
if fitness < pbest_fitness(i)
pbest_fitness(i) = fitness;
pbest_pos(i, :) = particles_pos(i, :);
end
if fitness < gbest_fitness
gbest_fitness = fitness;
gbest_pos = particles_pos(i, :);
end
end
% 更新粒子速度和位置
r1 = rand(n_particles, n_dimensions);
r2 = rand(n_particles, n_dimensions);
particles_vel = w * particles_vel + c1 * r1.* (pbest_pos - particles_pos) + c2 * r2.* (repmat(gbest_pos, n_particles, 1) - particles_pos);
particles_pos = particles_pos + particles_vel;
end
fprintf('Best C: %f, Best G: %f, Best Fitness: %f\n', gbest_pos(1), gbest_pos(2), -gbest_fitness);代码分析:
- 先定义了适应度函数
psosvmfitness,它接收参数params(包含C和G)、特征数据x和标签数据y。在函数内构建 LIBSVM 命令并通过svmtrain获得交叉验证准确率,取其相反数作为适应度,因为我们要最小化适应度来找到最优参数。 - 设置粒子群优化的参数,如粒子数量
nparticles*、维度n*dimensions(这里是 2,对应C和G)、最大迭代次数max_iterations等。 - 初始化粒子的位置和速度,每个粒子的位置代表一组
C和G的值。同时记录每个粒子的历史最佳位置pbestpos**和全局最佳位置gbestpos等。 - 在每次迭代中,计算每个粒子的适应度,更新粒子自身的最佳位置和全局最佳位置。
- 根据公式更新粒子的速度和位置,让粒子在解空间中不断搜索。
- 最后输出找到的最佳
C、G和适应度(转换回准确率)。
遗传算法优化支持向量机(GA - SVM)
遗传算法模拟生物进化过程,通过选择、交叉和变异等操作不断进化种群,找到最优解。
matlab
% 适应度函数定义
function fitness = ga_svm_fitness(params, x, y)
c = params(1);
g = params(2);
cmd = ['-v 5 -c ', num2str(c),'-g ', num2str(g)];
fitness = -svmtrain(y, x, cmd)(1);
end
% 遗传算法参数设置
nvars = 2;
lb = [0.01, 0.01];
ub = [100, 100];
options = gaoptimset('Generations', 100, 'PopulationSize', 50);
% 加载数据
load heart_scale;
x = heart_scale_inst;
y = heart_scale_label;
% 运行遗传算法
[x_best, fval] = ga(@(params) ga_svm_fitness(params, x, y), nvars, [], [], [], [], lb, ub, [], options);
fprintf('Best C: %f, Best G: %f, Best Fitness: %f\n', x_best(1), x_best(2), -fval);代码分析:
- 同样先定义适应度函数
gasvmfitness,逻辑和 PSO - SVM 中的适应度函数类似,将交叉验证准确率取相反数作为适应度。 - 设置遗传算法参数,如变量数量
nvars(2 个,对应C和G),变量下限lb和上限ub,以及遗传算法的选项options,这里设置了最大代数Generations和种群大小PopulationSize。 - 加载数据后,调用
ga函数运行遗传算法,它接收适应度函数句柄、变量数量等参数。 - 最后输出找到的最佳
C、G和适应度(转换回准确率)。
这几种方法都各有优劣,网格搜索简单直观但计算量大,PSO 和 GA 利用智能优化算法在一定程度上能更快找到较好解。而且这几种方法都简单易上手,你只要把数据替换成自己的,就能为你的 SVM 模型找到合适的参数啦。希望大家在实际应用中能灵活运用这些方法,让 SVM 模型发挥出最大威力!
粒子群优化支持向量机 遗传算法优化支持向量机 网格搜索 遍历法 PSO-SVM GA-SVM 。 用liv-SVM工具箱,选择较好的C和G。 简单容易上手,替换数据即可,有代码解释。