研究背景
该研究集中于通过将生物医学知识图谱(KG)与大型语言模型(LLMs)集成,以增强LLMs在生物医学应用中的表现。研究的动机源于LLMs在生成精确内容方面面临的挑战,尤其是在像生物医学这样准确性至关重要的领域。LLMs在广泛的通用文本上的传统训练并不总是能够在专业领域中转化为高准确性,这是因为模型尽管语言结构正确,却仍会生成错误信息(即"幻觉")。
研究目标
本研究的主要目标是创建一个框架,将丰富的结构化生物医学知识与LLMs的先进文本处理能力结合起来。这个框架旨在提高LLMs输出的事实正确性,使其更适合敏感的生物医学应用,而无需进行广泛的重新训练或特定领域的微调。
相关工作
LLM的限制:尽管LLM功能强大,但在处理复杂的特定领域信息 时常常生成非事实内容。
特定领域的LLM:如PubMedBERT和BioBERT等模型专门在生物医学文献上进行训练,以缓解一些问题,但需要巨大的计算资源。
LLM中的知识图谱:先前的工作已将LLM与KG整合,有效地利用了结构化领域知识,特别是在问答任务中。
方法论
数据处理
1.query->抽取实体,prompt对应为:

2.找到的graph中的三元组转为自然语言:
(Disease hypertension, ASSOCIATES_DaG, Gene VHL) → Disease hypertension associates Gene VHL
3.把检索出来的自然语言填充进Prompt,例如:

解决方案
从KG中检索出相关信息,回填Prompt,基本上走的原RAG后半段的G思路,R也是采用embedding,不过和非结构化文档的检索流程略不同。

详细流程如下:
- 实体识别:通过零样本提示(zero-shot prompting)实现的。研究人员设计了一个高效的抽取prompt,引导大型语言模型(如GPT-3.5-Turbo)从输入文本中提取疾病实体,并将结果以JSON格式返回。(替代原始NER模型)
- 实体链接:用语义相似度的方式来做。
实体embedding计算:首先,使用Embedding模型(如'all-MiniLM-L6-v2')为SPOKE知识图谱中的所有疾病概念节点计算embedding向量
将计算出的疾病embedding存储在向量数据库(如'Chroma')中,以便快速检索。
语义搜索匹配:将LLM提取的疾病实体与向量数据库中的疾病实体进行比较,选择最相似的
当然,如果零样本方法未能识别出疾病实体,采取的办法是直接拿原始query去匹配,取top 5。 - 子图查询:在得到具体的实体后,紧接着就是从KG中去查询这个实体关联的子图,这些信息通常以三元组(Subject, Predicate, Object)的形式存在,表示不同的生物医学关系。通常情况下,可以查询1~3跳内的三元组信息,这里借助图数据库可以比较容易的实现。
得到的三元组信息,LLM可能不太能比较好的理解,这里就需要将三元组转换成自然语言,以便与输入提示结合并用于后续的文本生成。举个例子:
(Disease hypertension, ASSOCIATES_DaG, Gene VHL) →Disease hypertension associates Gene VHL - 上下文剪枝:在KG-RAG框架中,Context Pruning(上下文剪枝)是一个关键步骤,就和dfs遍历时,需要剪枝来减少遍历时间一样,这里的剪枝可以减少给LLM的信息,减少token数量,同时过滤掉一些无用信心,还能提升LLM回答的精确性。
Context Pruning的具体做法还是会基于embedding来计算语义相似度,大概就是使用embedding模型计算三元组和query的cos相似度,最后选择策略:
条件一:上下文关联的余弦相似度必须大于所有提取上下文关联的相似度分布的75%分位
条件二:余弦相似度的最小值必须达到0.5
通过这个0.5 和 75%,可以有效减少给LLM的无效信息,有助于提高后续文本生成的准确性和相关性。
实验
实验设计
实验使用了多种预训练的LLM,包括Llama-2-13b、GPT-3.5-Turbo和GPT-4,利用KG-RAG框架处理不同的生物医学任务(例如,是非题、多项选择题和药物重定位)。比较这些模型在使用和不使用KG-RAG框架的情况下的表现,以评估整合KG信息的影响。
KG选用的是SPOKE,SPOKE是一个开放知识图谱,提供数据下载和开放API,整合了超过40个公开可用的生物医学知识源,涵盖了基因、蛋白质、药物、化合物、疾病等概念和概念之间的关系,
实验结论
-
引入KG-RAG后,所有测试任务中LLM输出的准确性和可靠性都显著提高。特别是,Llama-2模型在MCQ(多项选择题)数据集上的表现提高到显著的71%。
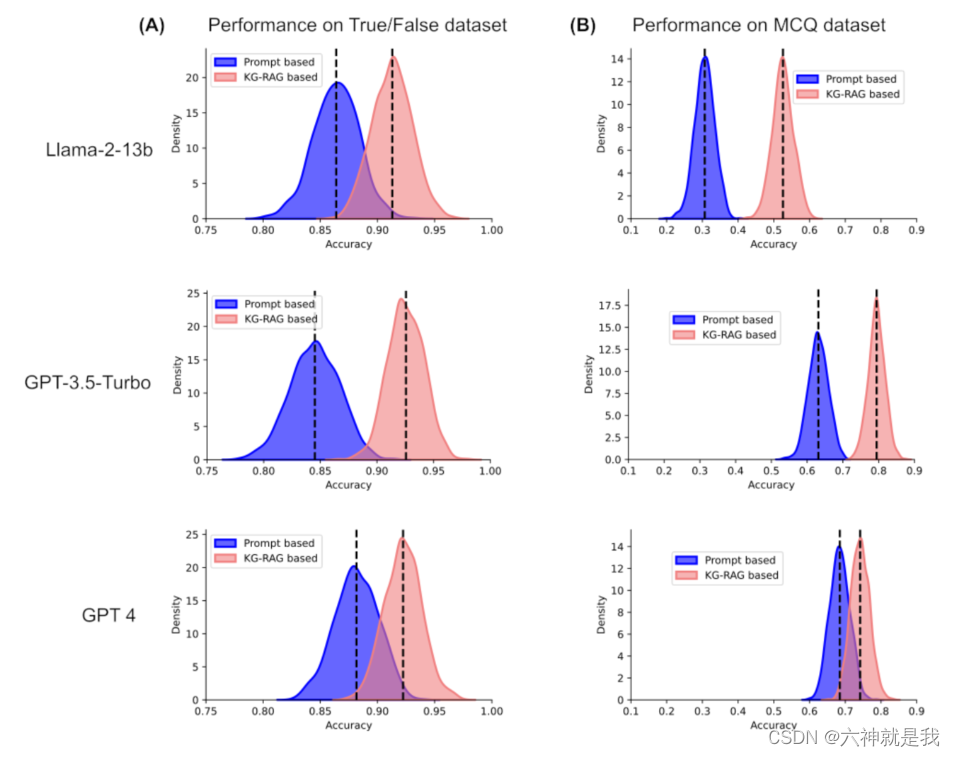
-
与KG-RAG集成的模型显示出更好的上下文利用能力,能生成更准确的生物医学文本回答,尤其是在复杂查询场景如药物重定位中。
