🍉CSDN小墨&晓末:https://blog.csdn.net/jd1813346972
个人介绍: 研一|统计学|干货分享
擅长Python、Matlab、R等主流编程软件
累计十余项国家级比赛奖项,参与研究经费10w、40w级横向
文章目录
- [1 项目任务](#1 项目任务)
- [2 数据集介绍](#2 数据集介绍)
- [3 加载相关包并读取数据](#3 加载相关包并读取数据)
- [4 探索和准备数据](#4 探索和准备数据)
- [5 划分训练集和测试集](#5 划分训练集和测试集)
- [6 拟合最小二乘线性回归模型](#6 拟合最小二乘线性回归模型)
- [7 拟合岭回归模型](#7 拟合岭回归模型)
- [8 拟合LASSO模型](#8 拟合LASSO模型)
- [9 拟合 PCR 模型](#9 拟合 PCR 模型)
- [10 拟合PLS模型](#10 拟合PLS模型)
- [11 模型评估](#11 模型评估)
- [12 完整代码](#12 完整代码)
该篇文章主要展示了利用R语言建立最小二乘线性回归、岭回归、LASSO、PCR、PLS五种模型对"College"数据集中的申请数量进行预测,并进行模型评估。
1 项目任务
使用"College"数据集中的其他变量预测收到的申请数量。
(a)将数据集分成训练集和测试集。
(b)在训练集上使用最小二乘法拟合线性模型,并报告所获得的测试误差。
©在训练集上拟合岭回归模型,通过交叉验证选择 λ。报告获得的测试误差。
(d)在训练集上拟合 LASSO 模型,通过交叉验证选择 λ。报告获得的测试误 差,以及非零系数估计值的数量。
(e)在训练集上拟合 PCR 模型,通过交叉验证选择 M。报告获得的测试误差, 以及通过交叉验证选择的 M 值。
(f)在训练集上拟合 PLS 模型,通过交叉验证选择 M。报告获得的测试误差, 以及通过交叉验证选择的 M 值。
(g)对获得的结果进行评论。我们能多准确地预测收到的大学申请数量?这 五种方法产生的测试误差有很大差别吗?
2 数据集介绍
这个数据集包含了大量关于不同学校的详细数据,主要变量包括: 1.Private:是否为私立学校。 2.Apps:申请人数。 3.Accept:被录取人数。 4.Enroll:注册人数。 5.Top10perc:排名前 10%的新生比例。 6.Top25perc:排名前 25%的新生比例。 7.F.Undergrad:全职本科生人数。 8.P.Undergrad:兼职本科生人数。 9.Outstate:州外学费。 10.Room.Board:房间和伙食费用。 11.Books:书籍费用。 12.Personal:个人费用。 13.PhD:拥有博士学位的教师比例。 14.Terminal:教师拥有终极学位的比例。 15.S.F.Ratio:学生与教师的比例。 16.perc.alumn:捐赠的校友比例。 17.Expend:学生教育支出。 18.Grad.Rate:毕业率。 此外,该数据集还公布了学校名称。
3 加载相关包并读取数据
运行程序:
library("class")
library("caret")
library("gmodels")
library('pls')
data<- read.csv("D:\\机器学习与R语言\\College.csv",header = T)
head(data) #查看前六行运行结果:
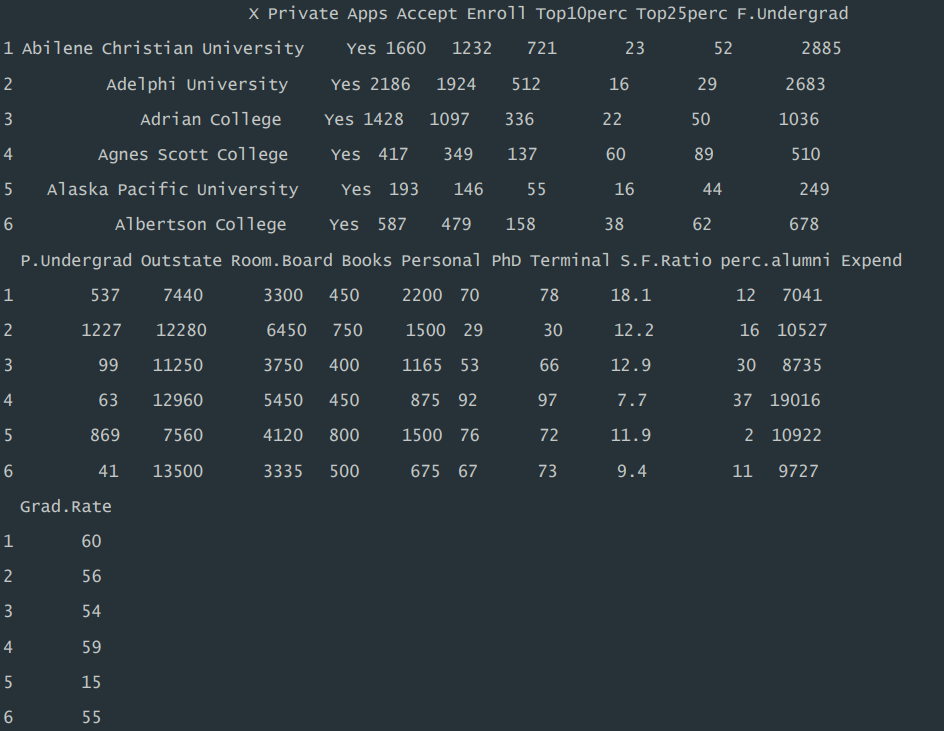
4 探索和准备数据
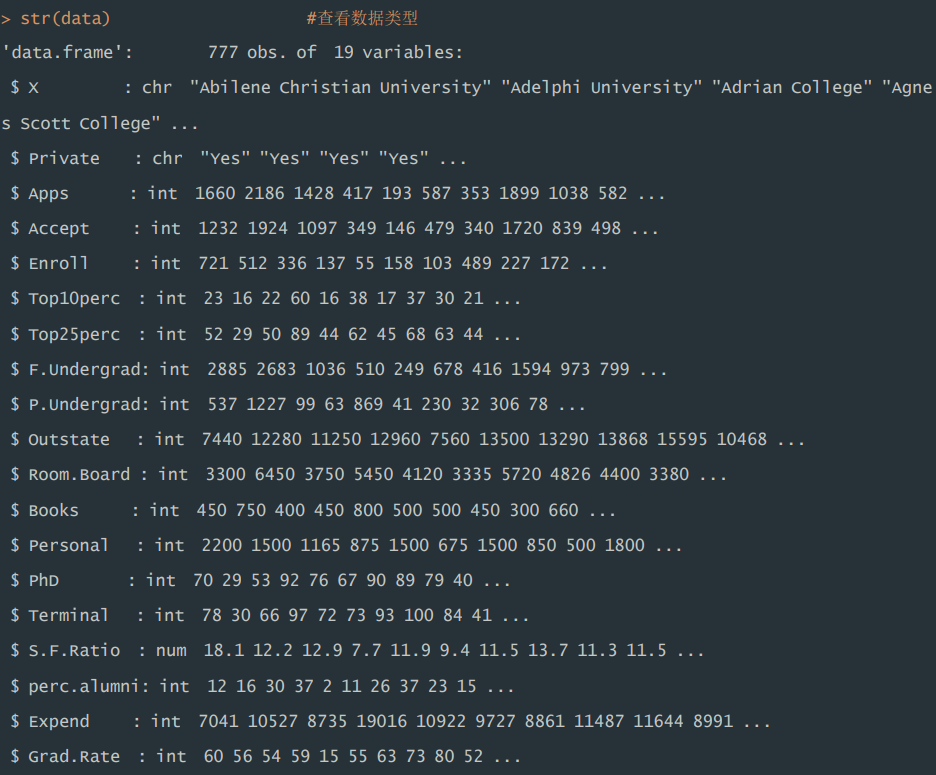
1)使用 str()发现,该数据集共有 777 个观测值,19 个属性,同时,其涉及 的数据类型有字符型(int)和整型(int),其中"Apps"为目标变量。
运行程序:
str(data) #查看数据类型运行结果:
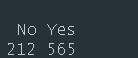
2)使用 table()函数输出数据集中"Private"变量分布情况,同时利用 ifelse ()函数将其转化为 0-1 变量,其中"Yes"转化为 1,"No"转化为 0。该数据 集中,私立学校和公立学校数量分别为 565、212。
运行程序:
table(data$Private)
data$Private_Yes=ifelse(data$Private=="Yes",1,0) #将分类变量Private转化为0-1变量,保存为Private_Yes
data=data[c(1,3:20)] ##剔除字符变量Private运行结果:
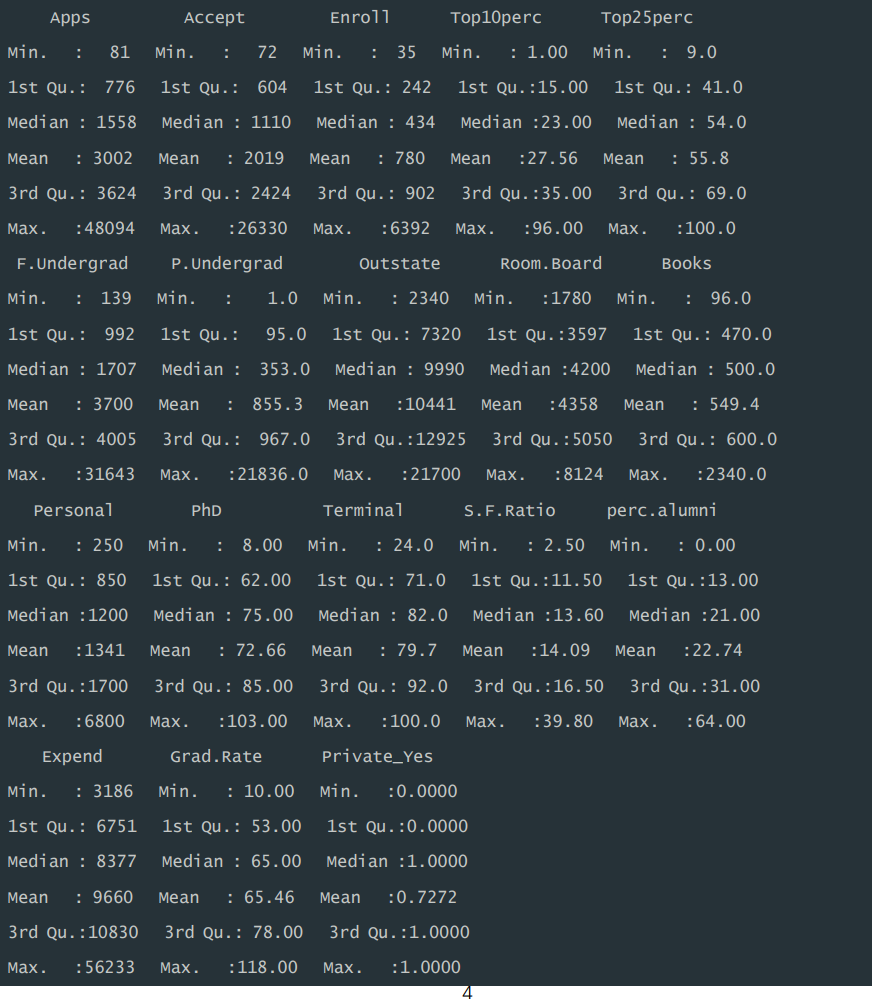
3)利用 summary()查看各变量数据特征情况,见运行结果,包括每个特 征的最小值,1/4 分位数,中位数,均值,3/4 分位数,最大值。其中"Apps"数 量分布在 81-48094 之间。
运行程序:
data<-data[c('Apps','Accept','Enroll',
'Top10perc','Top25perc','F.Undergrad','P.Undergrad',
'Outstate','Room.Board','Books','Personal',
'PhD','Terminal','S.F.Ratio','perc.alumni','Expend','Grad.Rate',
'Private_Yes')]
summary(data)运行结果:
5 划分训练集和测试集
运行程序:
set.seed(123) #设置随机种子以保证结果可重复
trainIndex=createDataPartition(data$Apps,p=0.8,
list=FALSE,
times=1)
data_train<-data[trainIndex, ] #创建训练集
data_test<-data[-trainIndex, ] #创建测试集6 拟合最小二乘线性回归模型
1)模型总结。训练集中,根据运行结果显示,线性回归方程 F 值为 537.3, p 值远小于 0.05,说明拟合的回归方程在 0.05 的显著性水平上显著成立。拟合优 度 R 2 为 0.9378,说明申请数量有 93.78%的信息可以用模型中的自变量来解释, 模型效果较好。其中,变量"Accept"、"Enroll"、"Top10perc"、"Top25perc"、 "F.Undergrad"、"Outstate"、"Expend"、"Grad.Rate"、"Private_Yes"在 0.05 的显著性水平下,对因变量"Apps"存在显著影响。
运行程序:
lm_model=lm(Apps~.,data=data_train) #拟合线性回归模型
summary(lm_model)运行结果:
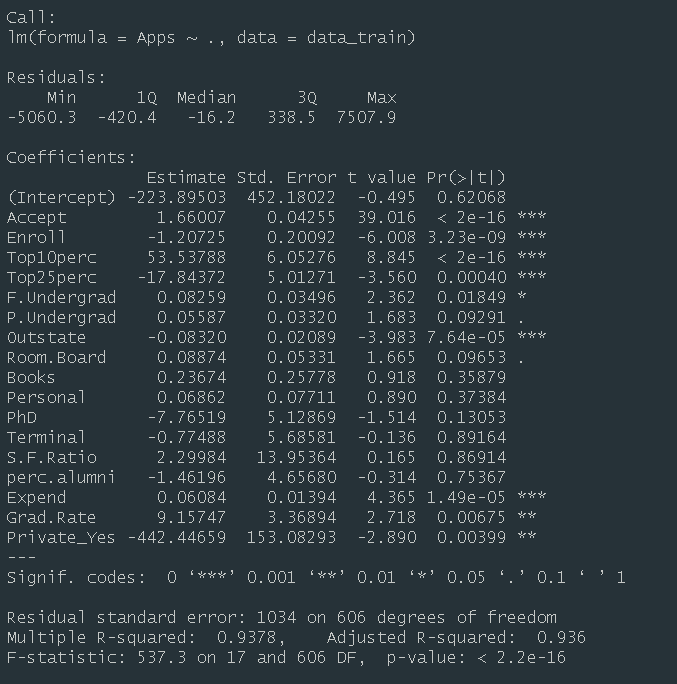
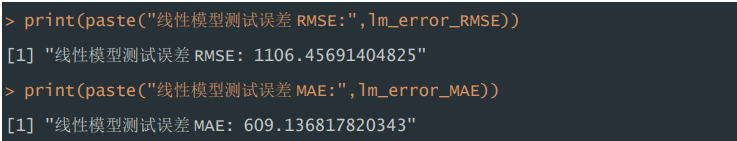
2)测试误差。根据运行结果显示,最小二乘线性回归模型,在测试集表现 中,RMSE 为 1106.46,MAE 为 609.14。
运行程序:
lm_pred=predict(lm_model,newdata=data_test) #对测试集进行预测
lm_error_RMSE=sqrt(mean((lm_pred-data_test$Apps)^2)) #计算均方根误差
lm_error_MAE=mean(abs(lm_pred-data_test$Apps)) #计算绝对平均误差
print(paste("线性模型测试误差RMSE:",lm_error_RMSE))
print(paste("线性模型测试误差MAE:",lm_error_MAE))运行结果:
7 拟合岭回归模型
1)模型总结。训练集中,在拟合岭回归模型过程中,利用交叉验证的方式 选择最优正则化λ,根据结果运行显示,对应最小 MSE 的λ值为 388.9,MSE 为1785806,在该 λ 值下,模型中非零系数的数量为 17(不包含截距项),且在最小 MSE 的一个标准误差内的最大 λ 值为 1430.4,MSE 为 2405994,该 λ 值 下,模型中非零系数的数量为 17(不包含截距项)。
运行程序:
ridge_model=cv.glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps,alpha=0) #交叉验证拟合岭回归模型
print(ridge_model) #输出模型运行结果:
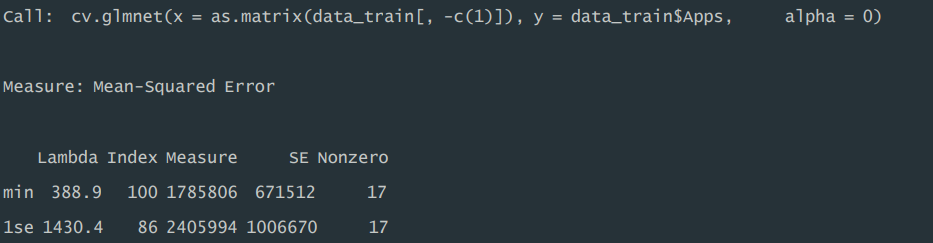
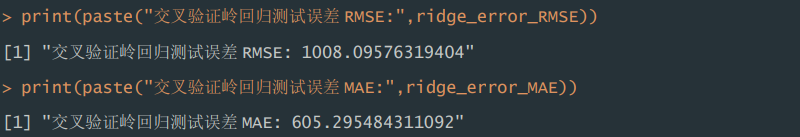
2)测试误差。根据运行结果显示,交叉验证的岭回归模型,在测试集表现 中,RMSE 为 1108.10,MAE 为 605.30。
运行程序:
ridge_model <- glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps, alpha = 0, lambda = best_lambda)
ridge_pred <- predict(ridge_model, newx = as.matrix(data_test[,-c(1)]), s = best_lambda)
ridge_error_RMSE=sqrt(mean((ridge_pred-data_test$Apps)^2)) #计算均方根误差
ridge_error_MAE=mean(abs(ridge_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证岭回归测试误差RMSE:",ridge_error_RMSE))
print(paste("交叉验证岭回归测试误差MAE:",ridge_error_MAE))运行结果:
8 拟合LASSO模型
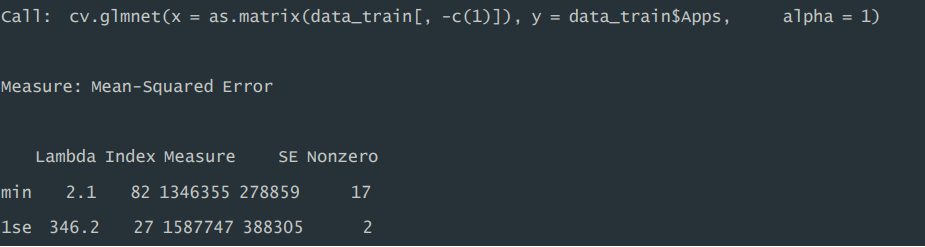
1)模型总结。训练集中,在拟合 LASSO 回归模型过程中,利用交叉验证 的方式选择最优正则化λ,根据结果运行显示,对应最小 MSE 的λ值为 2.1,MSE 为 1346355,在该 λ 值下,模型中非零系数的数量为 17(不包含截距 项),且在最小 MSE 的一个标准误差内的最大 λ 值为 346.2,MSE 为 1587747,该 λ 值下,模型中非零系数的数量为 2(不包含截距项)。
运行程序:
lasso_model=cv.glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps,alpha=1) #交叉验证拟合lasso回归模型
print(lasso_model) #输出模型运行结果:
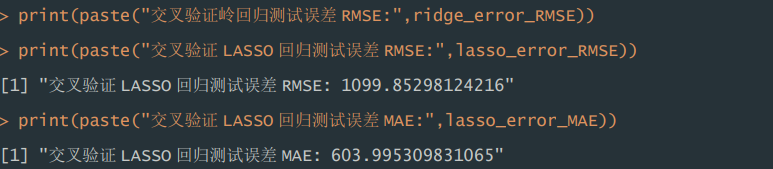
2)测试误差。根据运行结果显示,交叉验证的 LASSO 回归模型,在测试 集表现中,RMSE 为 1099.85,MAE 为 604.00。
运行程序:
best_lambda <- lasso_model$lambda.min
print(paste("最优lambda:",best_lambda))
# 使用最佳λ在训练集上重新拟合模型
lasso_model <- glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps, alpha = 1, lambda = best_lambda)
lasso_pred=predict(lasso_model,newx = as.matrix(data_test[,-c(1)]), s = best_lambda) #对测试集进行预测
lasso_error_RMSE=sqrt(mean((lasso_pred-data_test$Apps)^2)) #计算均方根误差
lasso_error_MAE=mean(abs(lasso_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证LASSO回归测试误差RMSE:",lasso_error_RMSE))
print(paste("交叉验证LASSO回归测试误差MAE:",lasso_error_MAE))运行结果:
9 拟合 PCR 模型
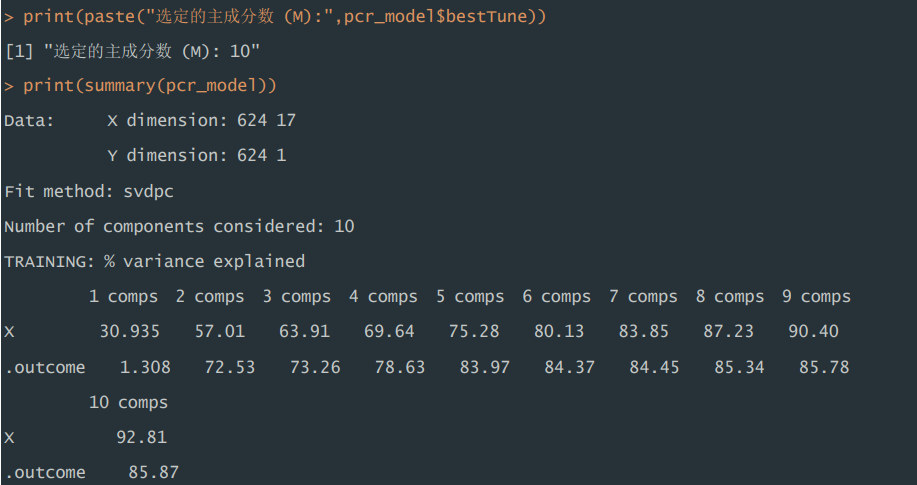
1)模型总结。训练集中,在拟合 PCR 模型过程中,利用交叉验证的方式 选择最优主成分数量 M 为 10,当使用 10 个主成分时,可以解释原始数据中 85.87%的方差,解释能力较强。
运行程序:
##拟合PCR模型
pcr_model=train(Apps~.,data=data_train,method="pcr",
preProcess=c("center","scale"),tuneLength=10) #交叉验证拟合主成分回归模型
##选定的主成分数M
print(paste("选定的主成分数 (M):",pcr_model$bestTune))
print(summary(pcr_model))运行结果:
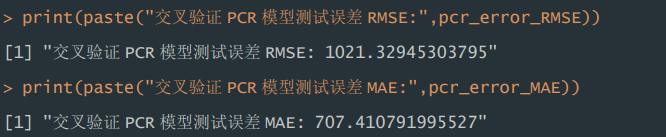
2)测试误差。根据运行结果显示,交叉验证的 PCR 模型,在测试集表现 中,RMSE 为 1021.33,MAE 为 707.41。
运行程序:
pcr_pred=predict(pcr_model,newdata=data_test)
pcr_error_RMSE=sqrt(mean((pcr_pred-data_test$Apps)^2)) #计算均方根误差
pcr_error_MAE=mean(abs(pcr_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证PCR模型测试误差RMSE:",pcr_error_RMSE))
print(paste("交叉验证PCR模型测试误差MAE:",pcr_error_MAE))运行结果:
10 拟合PLS模型
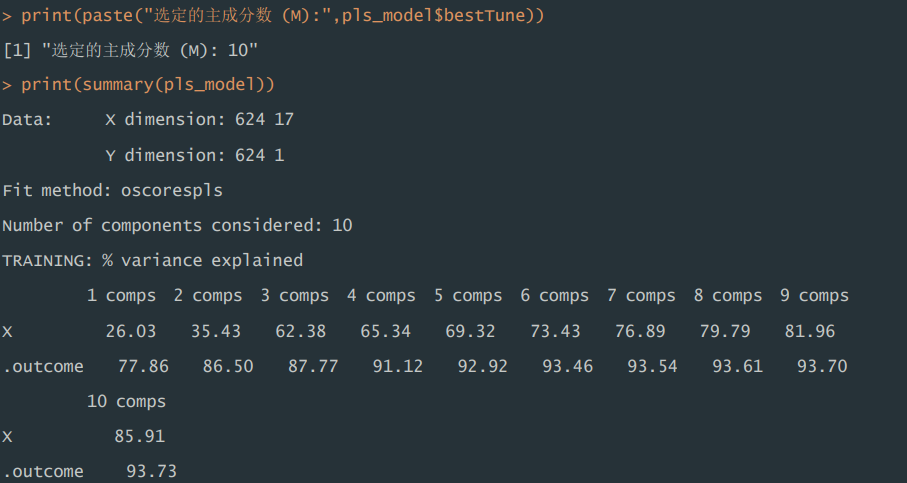
1)模型总结。训练集中,在拟合 PLS 回归模型过程中,利用交叉验证的 方式选择最优主成分数量 M 为 10,当使用 10 个主成分时,可以解释原始数据 中 93.73%的方差,解释能力较强。
运行程序:
pls_model=train(Apps~.,data=data_train,method="pls",
preProcess=c("center","scale"),tuneLength=10) #交叉验证拟合偏最小二乘回归
##选定的主成分数M
print(paste("选定的主成分数 (M):",pls_model$bestTune))
print(summary(pls_model))运行结果:
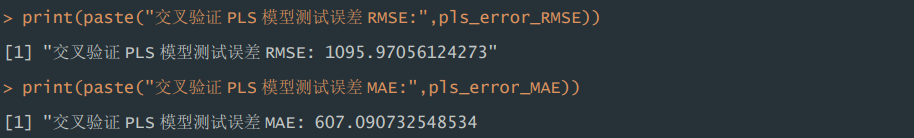
2)测试误差。根据运行结果显示,交叉验证的 PCR 模型,在测试集表现 中,RMSE 为 1095.97,MAE 为 607.09。
运行程序:
pls_pred=predict(pls_model,newdata=data_test)
pls_error_RMSE=sqrt(mean((pls_pred-data_test$Apps)^2)) #计算均方根误差
pls_error_MAE=mean(abs(pls_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证PLS模型测试误差RMSE:",pls_error_RMSE))
print(paste("交叉验证PLS模型测试误差MAE:",pls_error_MAE))运行结果:
11 模型评估
本实验在训练集过程中,利用训练集分别拟合了最小二乘线性回归模型、交 叉验证的岭回归模型、交叉验证的 LASSO 模型、PCR 模型、PLS 回归模型五 个模型,并在测试集进行测试,以 RMSE、MAE 指标评估模型。结果见表 1 所 示。
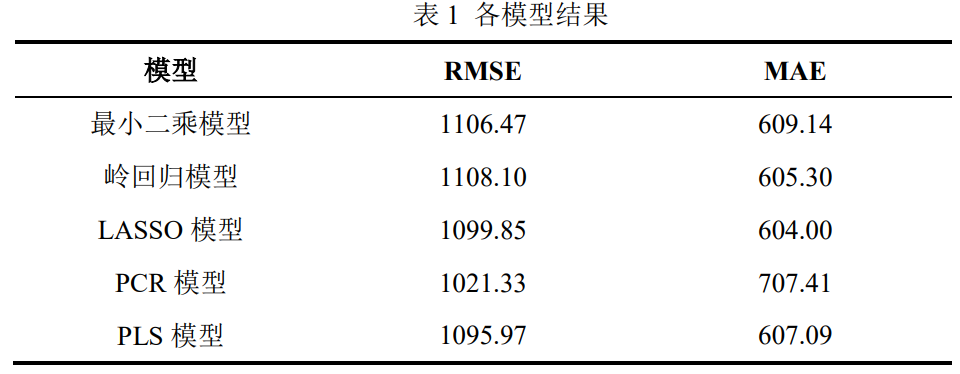
根据表 1 可以看出,五种模型在测试集表现中,RMSE 集中在 1100 附近, 其中 PCR 模型的 RMSE 指标最低,MAE 大多集中在 600 附近,PCR 为 707,五 种模型总体上性能相似。
12 完整代码
##加载相关包并读取数据
library("class")
library("caret")
library("gmodels")
library('pls')
data<- read.csv("D:\\机器学习与R语言\\College.csv",header = T)
head(data) #查看前六行
str(data) #查看数据类型
table(data$Private)
data$Private_Yes=ifelse(data$Private=="Yes",1,0) #将分类变量Private转化为0-1变量,保存为Private_Yes
data=data[c(1,3:20)] ##剔除字符变量Private
data<-data[c('Apps','Accept','Enroll',
'Top10perc','Top25perc','F.Undergrad','P.Undergrad',
'Outstate','Room.Board','Books','Personal',
'PhD','Terminal','S.F.Ratio','perc.alumni','Expend','Grad.Rate',
'Private_Yes')]
summary(data)
set.seed(123) #设置随机种子以保证结果可重复
trainIndex=createDataPartition(data$Apps,p=0.8,
list=FALSE,
times=1)
data_train<-data[trainIndex, ] #创建训练集
data_test<-data[-trainIndex, ] #创建测试集
##最小二乘线性回归
lm_model=lm(Apps~.,data=data_train) #拟合线性回归模型
summary(lm_model)
lm_pred=predict(lm_model,newdata=data_test) #对测试集进行预测
lm_error_RMSE=sqrt(mean((lm_pred-data_test$Apps)^2)) #计算均方根误差
lm_error_MAE=mean(abs(lm_pred-data_test$Apps)) #计算绝对平均误差
print(paste("线性模型测试误差RMSE:",lm_error_RMSE))
print(paste("线性模型测试误差MAE:",lm_error_MAE))
##拟合岭回归模型
ridge_model=cv.glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps,alpha=0) #交叉验证拟合岭回归模型
print(ridge_model) #输出模型
# 使用最佳λ在训练集上重新拟合模型
ridge_model <- glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps, alpha = 0, lambda = best_lambda)
ridge_pred <- predict(ridge_model, newx = as.matrix(data_test[,-c(1)]), s = best_lambda)
ridge_error_RMSE=sqrt(mean((ridge_pred-data_test$Apps)^2)) #计算均方根误差
ridge_error_MAE=mean(abs(ridge_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证岭回归测试误差RMSE:",ridge_error_RMSE))
print(paste("交叉验证岭回归测试误差MAE:",ridge_error_MAE))
##LASSO回归
lasso_model=cv.glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps,alpha=1) #交叉验证拟合lasso回归模型
print(lasso_model) #输出模型
# 选择最佳λ(最小交叉验证误差的λ)
best_lambda <- lasso_model$lambda.min
print(paste("最优lambda:",best_lambda))
# 使用最佳λ在训练集上重新拟合模型
lasso_model <- glmnet(as.matrix(data_train[,-c(1)]),data_train$Apps, alpha = 1, lambda = best_lambda)
lasso_pred=predict(lasso_model,newx = as.matrix(data_test[,-c(1)]), s = best_lambda) #对测试集进行预测
lasso_error_RMSE=sqrt(mean((lasso_pred-data_test$Apps)^2)) #计算均方根误差
lasso_error_MAE=mean(abs(lasso_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证LASSO回归测试误差RMSE:",lasso_error_RMSE))
print(paste("交叉验证LASSO回归测试误差MAE:",lasso_error_MAE))
##拟合PCR模型
pcr_model=train(Apps~.,data=data_train,method="pcr",
preProcess=c("center","scale"),tuneLength=10) #交叉验证拟合主成分回归模型
##选定的主成分数M
print(paste("选定的主成分数 (M):",pcr_model$bestTune))
print(summary(pcr_model))
pcr_pred=predict(pcr_model,newdata=data_test)
pcr_error_RMSE=sqrt(mean((pcr_pred-data_test$Apps)^2)) #计算均方根误差
pcr_error_MAE=mean(abs(pcr_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证PCR模型测试误差RMSE:",pcr_error_RMSE))
print(paste("交叉验证PCR模型测试误差MAE:",pcr_error_MAE))
##拟合PLS回归模型
pls_model=train(Apps~.,data=data_train,method="pls",
preProcess=c("center","scale"),tuneLength=10) #交叉验证拟合偏最小二乘回归
##选定的主成分数M
print(paste("选定的主成分数 (M):",pls_model$bestTune))
print(summary(pls_model))
pls_pred=predict(pls_model,newdata=data_test)
pls_error_RMSE=sqrt(mean((pls_pred-data_test$Apps)^2)) #计算均方根误差
pls_error_MAE=mean(abs(pls_pred-data_test$Apps)) #计算绝对平均误差
print(paste("交叉验证PLS模型测试误差RMSE:",pls_error_RMSE))
print(paste("交叉验证PLS模型测试误差MAE:",pls_error_MAE))