前言
随着多模态大模型的发展,其不仅限于文字处理,更能够在图像、视频、音频方面进行识别与理解。医疗领域中,医生们往往需要对各种医学图像进行处理,以辅助诊断和治疗。如果将多模态大模型与图像诊断相结合,那么这会极大地提升诊断效率。
项目目标
训练一个医疗多模态大模型,用于图像诊断。
刚好家里老爷子近期略感头疼,去医院做了脑部CT,诊断患有垂体瘤,我将尝试使用多模态大模型进行进一步诊断。
实现过程
1. 数据集准备
为了训练模型,需要准备大量的医学图像数据。通过搜索我们找到以下训练数据:
数据名称 :MedTrinity-25M
数据地址 :https://github.com/UCSC-VLAA/MedTrinity-25M
数据简介 :MedTrinity-25M数据集是一个用于医学图像分析和计算机视觉研究的大型数据集。
数据来源 :该数据集由加州大学圣克鲁兹分校(UCSC)提供,旨在促进医学图像处理和分析的研究。
数据量 :MedTrinity-25M包含约2500万条医学图像数据,涵盖多种医学成像技术,如CT、MRI和超声等。
数据内容 :
该数据集有两份,分别是 25Mdemo 和 25Mfull 。
25Mdemo (约162,000条)数据集内容如下:
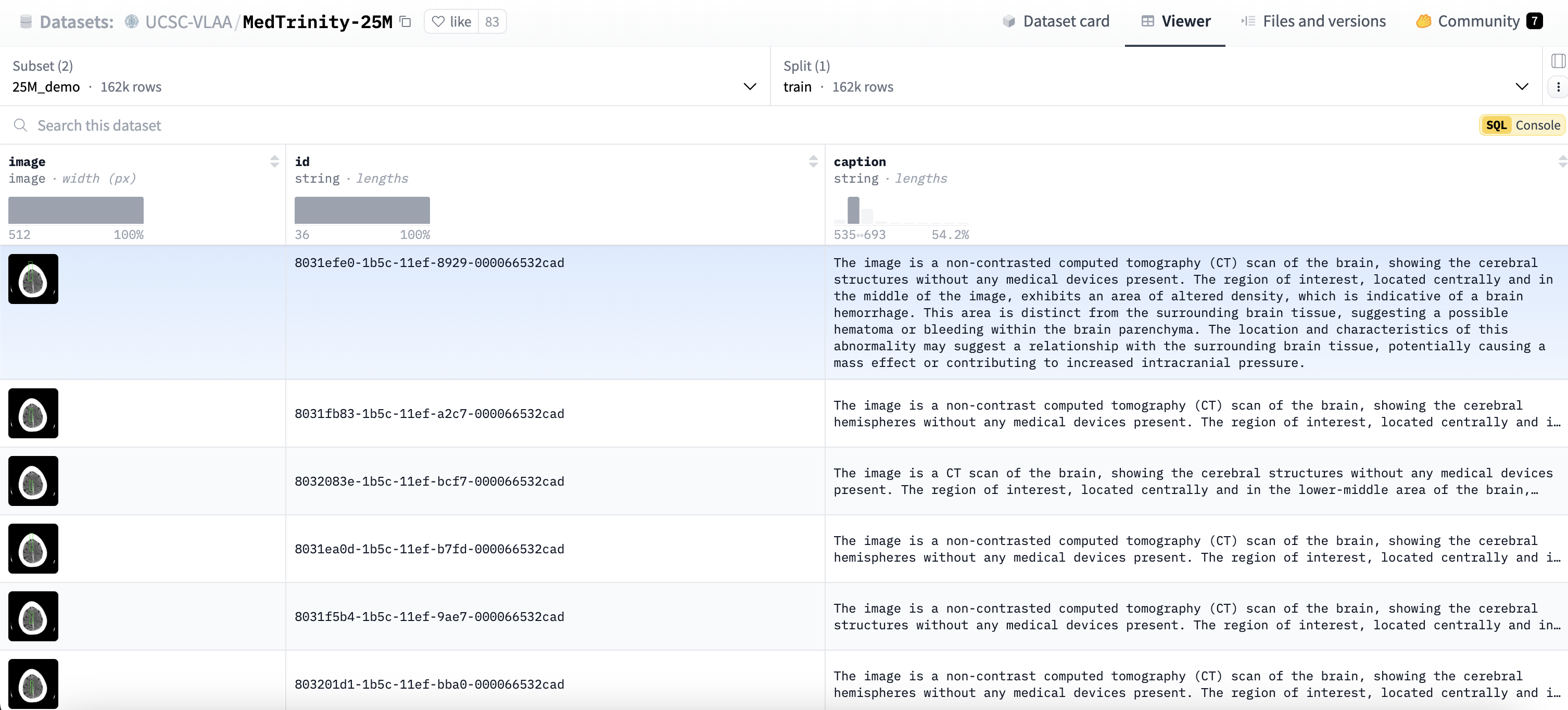
25Mfull (约24,800,000条)数据集内容如下:
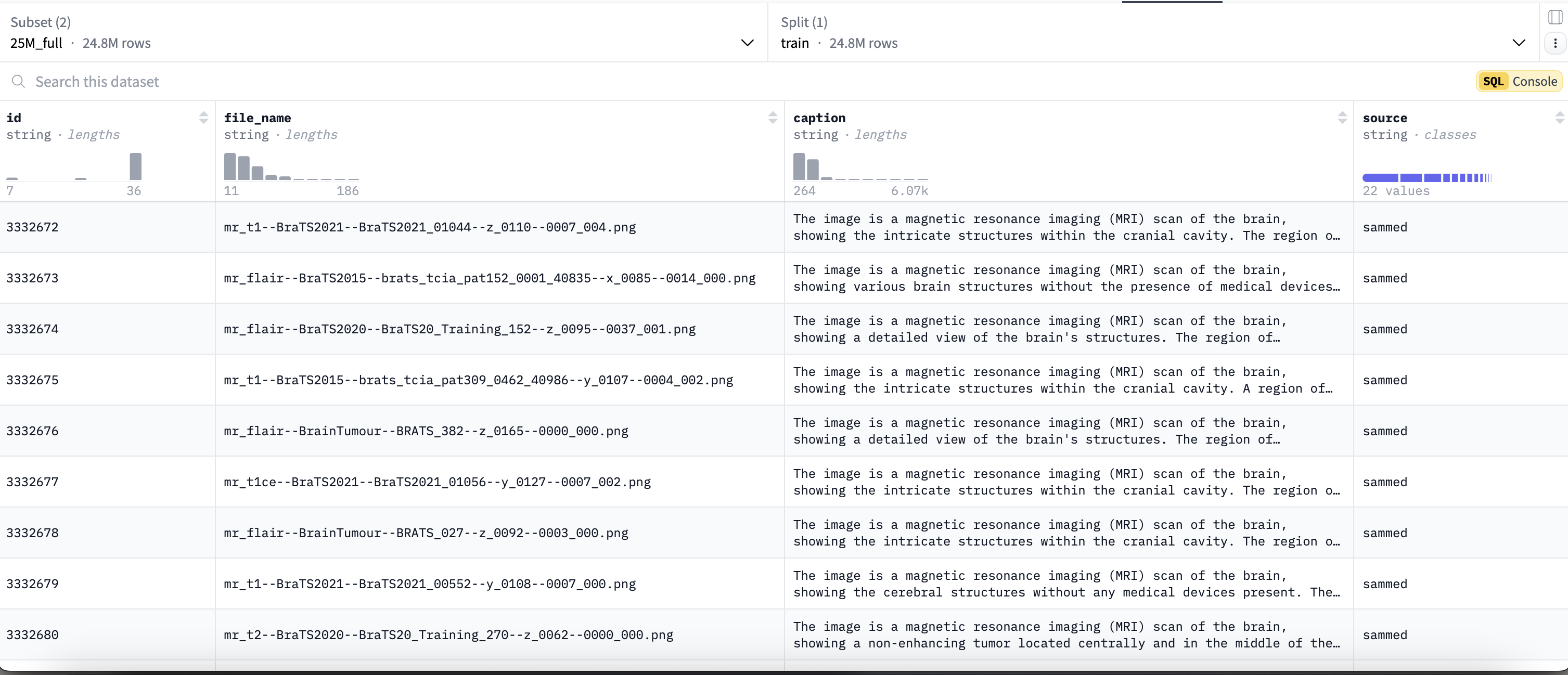
2. 数据下载
2.1 安装Hugging Face的Datasets库
bash
pip install datasets2.2 下载数据集
python
from datasets import load_dataset
# 加载数据集
ds = load_dataset("UCSC-VLAA/MedTrinity-25M", "25M_demo", cache_dir="cache")执行结果:
说明:
- 以上方法是使用HuggingFace的Datasets库下载数据集,下载的路径为当前脚本所在路径下的cache文件夹。
- 使用HuggingFace下载需要能够访问https://huggingface.co/ 并且在网站上申请数据集读取权限才可以。
- 如果没有权限访问HuggingFace,可以关注一起AI技术 公众号后,回复 "MedTrinity"获取百度网盘下载地址。
2.3 预览数据集
python
# 查看训练集的前1个样本
print(ds['train'][:1]) 运行结果:
bash
{
'image': [<PIL.JpegImagePlugin.JpegImageFile image mode=RGB size=512x512 at 0x15DD6D06530>],
'id': ['8031efe0-1b5c-11ef-8929-000066532cad'],
'caption': ['The image is a non-contrasted computed tomography (CT) scan of the brain, showing the cerebral structures without any medical devices present. The region of interest, located centrally and in the middle of the image, exhibits an area of altered density, which is indicative of a brain hemorrhage. This area is distinct from the surrounding brain tissue, suggesting a possible hematoma or bleeding within the brain parenchyma. The location and characteristics of this abnormality may suggest a relationship with the surrounding brain tissue, potentially causing a mass effect or contributing to increased intracranial pressure.'
]
}使用如下命令对数据集的图片进行可视化查看:
python
# 可视化image内容
from PIL import Image
import matplotlib.pyplot as plt
image = ds['train'][0]['image'] # 获取第一张图像
plt.imshow(image)
plt.axis('off') # 不显示坐标轴
plt.show()运行结果:

3. 数据预处理
由于后续我们要通过LLama Factory进行多模态大模型微调,所以我们需要对上述的数据集进行预处理以符合LLama Factory的要求。
3.1 LLama Factory数据格式
查看LLama Factory的多模态数据格式要求如下:
json
[
{
"messages": [
{
"content": "<image>他们是谁?",
"role": "user"
},
{
"content": "他们是拜仁慕尼黑的凯恩和格雷茨卡。",
"role": "assistant"
},
{
"content": "他们在做什么?",
"role": "user"
},
{
"content": "他们在足球场上庆祝。",
"role": "assistant"
}
],
"images": [
"mllm_demo_data/1.jpg"
]
}
]3.2 实现数据格式转换脚本
python
from datasets import load_dataset
import os
import json
from PIL import Image
def save_images_and_json(ds, output_dir="mllm_data"):
"""
将数据集中的图像和对应的 JSON 信息保存到指定目录。
参数:
ds: 数据集对象,包含图像和标题。
output_dir: 输出目录,默认为 "mllm_data"。
"""
# 创建输出目录
if not os.path.exists(output_dir):
os.makedirs(output_dir)
# 创建一个列表来存储所有的消息和图像信息
all_data = []
# 遍历数据集中的每个项目
for item in ds:
img_path = f"{output_dir}/{item['id']}.jpg" # 图像保存路径
image = item["image"] # 假设这里是一个 PIL 图像对象
# 将图像对象保存为文件
image.save(img_path) # 使用 PIL 的 save 方法
# 添加消息和图像信息到列表中
all_data.append(
{
"messages": [
{
"content": "<image>图片中的诊断结果是怎样?",
"role": "user",
},
{
"content": item["caption"], # 从数据集中获取的标题
"role": "assistant",
},
],
"images": [img_path], # 图像文件路径
}
)
# 创建 JSON 文件
json_file_path = f"{output_dir}/mllm_data.json"
with open(json_file_path, "w", encoding='utf-8') as f:
json.dump(all_data, f, ensure_ascii=False) # 确保中文字符正常显示
if __name__ == "__main__":
# 加载数据集
ds = load_dataset("UCSC-VLAA/MedTrinity-25M", "25M_demo", cache_dir="cache")
# 保存数据集中的图像和 JSON 信息
save_images_and_json(ds['train'])运行结果:
4. 模型下载
本次微调,我们使用阿里最新发布的多模态大模型:Qwen2-VL-2B-Instruct 作为底座模型。
模型说明地址:https://modelscope.cn/models/Qwen/Qwen2-VL-2B-Instruct
使用如下命令下载模型
bash
git lfs install
# 下载模型
git clone https://www.modelscope.cn/Qwen/Qwen2-VL-2B-Instruct.git5. 环境准备
5.1 机器环境
硬件:
- 显卡:4080 Super
- 显存:16GB
软件:
- 系统:Ubuntu 20.04 LTS
- python:3.10
- pytorch:2.1.2 + cuda12.1
5.2 准备虚拟环境
bash
# 创建python3.10版本虚拟环境
conda create --name train_env python=3.10
# 激活环境
conda activate train_env
# 安装依赖包
pip install streamlit torch torchvision
# 安装Qwen2建议的transformers版本
pip install git+https://github.com/huggingface/transformers6. 准备训练框架
下载并安装LLamaFactory框架的具体步骤,请见【课程总结】day24(上):大模型三阶段训练方法(LLaMa Factory)中 准备训练框架 部分内容,本章不再赘述。
6.1 修改LLaMaFactory源码以适配transformer
由于Qwen2-VL使用的transformer的版本为4.47.0.dev0,LLamaFactory还不支持,所以需要修改LLaMaFactory的代码,具体方法如下:
第一步 :在 llamafactory 源码中,找到 check_dependencies() 函数,这个函数位于 src/llamafactory/extras/misc.py 文件的第 82 行。
第二步 :修改 check_dependencies() 函数并保存
python
# 原始代码
require_version("transformers>=4.41.2,<=4.45.2", "To fix: pip install transformers>=4.41.2,<=4.45.2")
# 修改后代码
require_version("transformers>=4.41.2,<=4.47.0", "To fix: pip install transformers>=4.41.2,<=4.47.0")第三步:重新启动LLaMaFactory服务
bash
llamafactory-cli webui这个过程可能会提示 ImportError: accelerate>=0.34.0 is required for a normal functioning of this module, but found accelerate==0.32.0.
如遇到上述问题,可以重新安装accelerate,如下:
bash
# 卸载旧的 accelerate
pip uninstall accelerate
# 安装新的 accelerate
pip install accelerate==0.34.07. 测试当前模型
第一步:启动LLaMa Factory后,访问http://0.0.0.0:7860
第二步:在web页面配置模型路径为 4.步骤 下载的模型路径,并点击加载模型
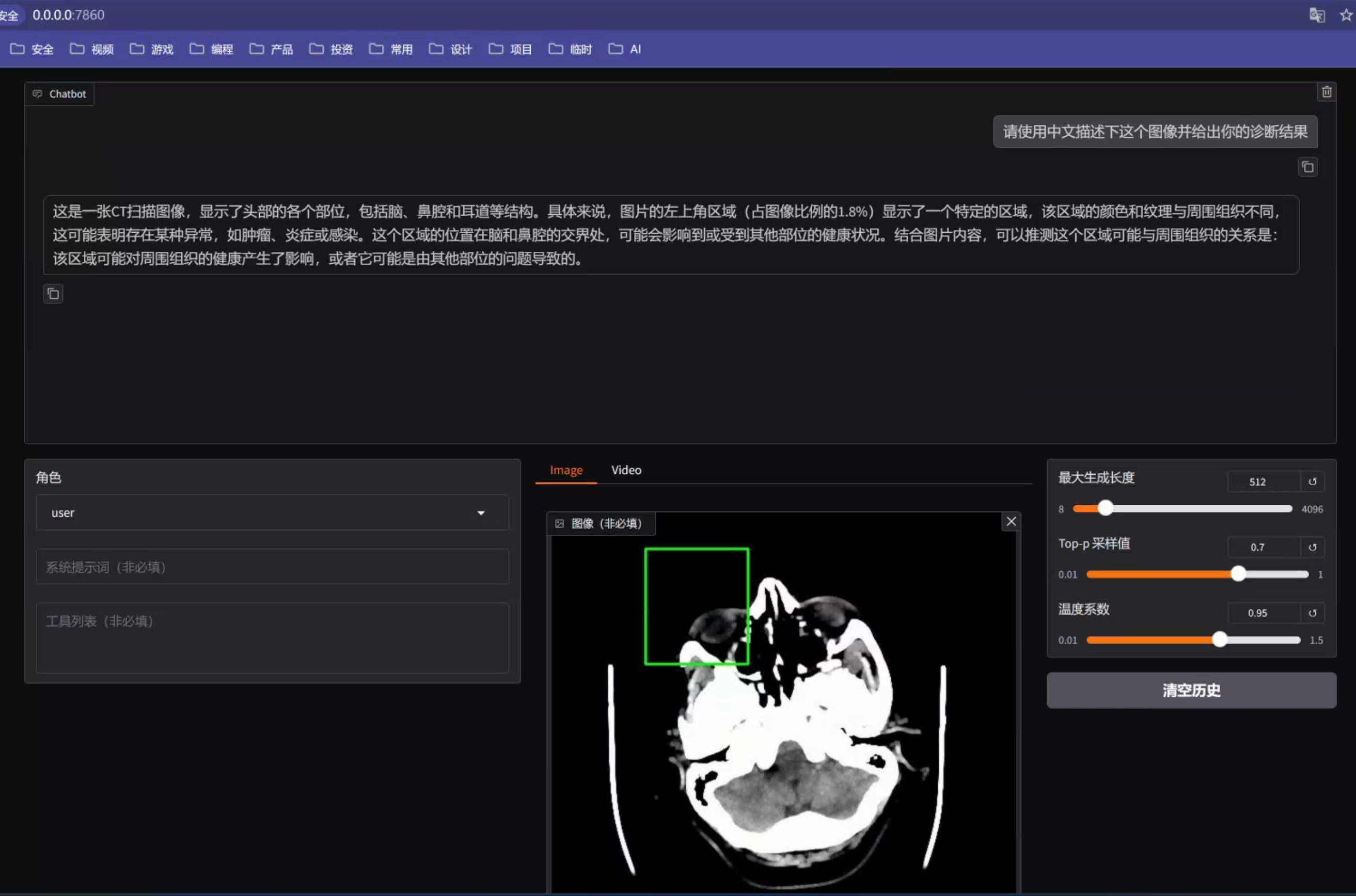
第三步:上传一张CT图片并输入问题:"请使用中文描述下这个图像并给出你的诊断结果"
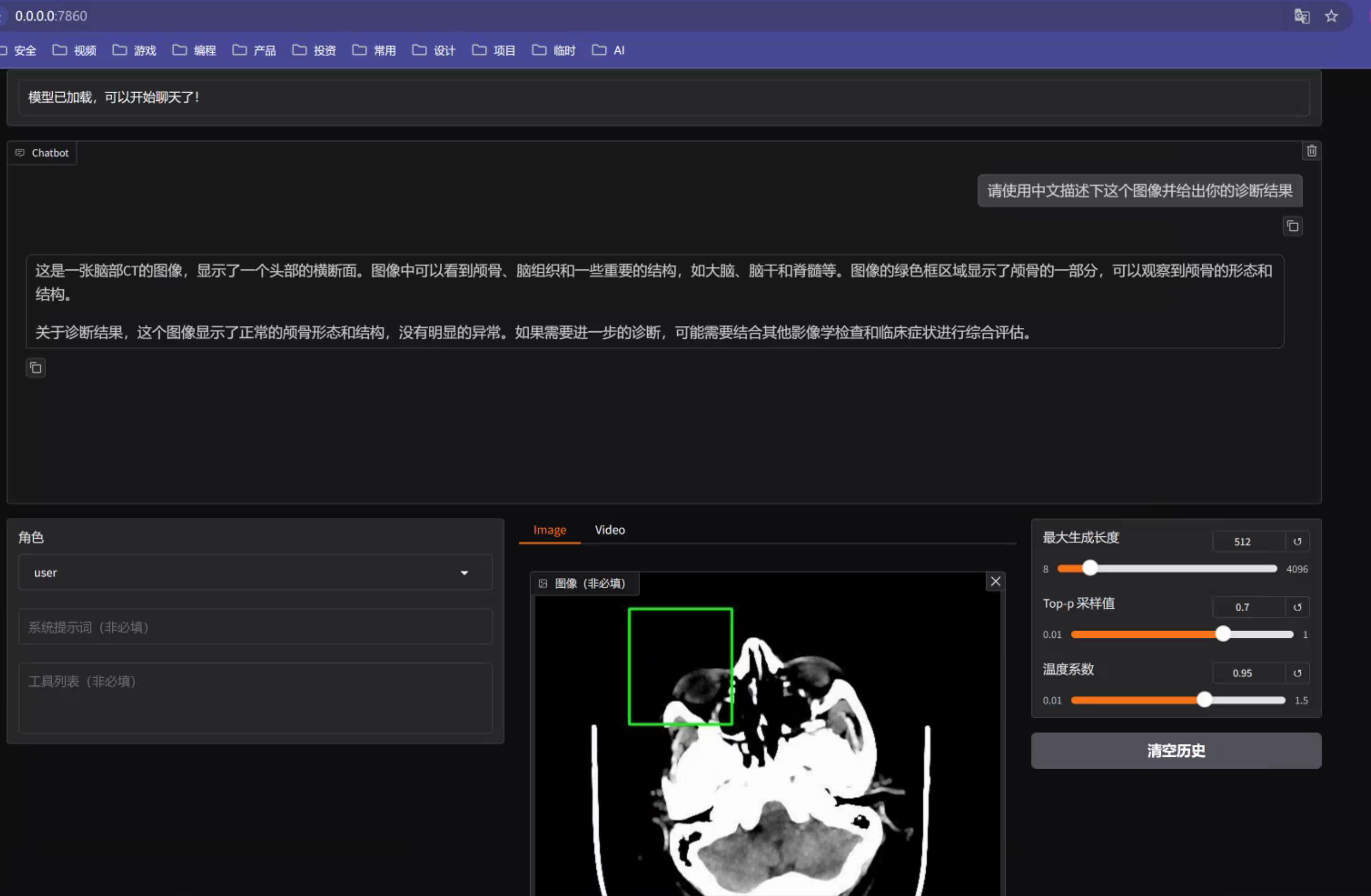
由上图可以看到,模型能够识别到这是一个CT图像,显示了大概的位置以及相应的器官,但是并不能给出是否存在诊断结果。
8. 模型训练
8.1 数据准备
第一步:将 3.2步骤 生成的mllm_data文件拷贝到LLaMaFactory的data目录下
第二步:将 4.步骤 下载的底座模型Qwen2-VL 拷贝到LLaMaFactory的model目录下
第三步:修改 LLaMaFactory data目录下的dataset_info.json,增加自定义数据集:
json
"mllm_med": {
"file_name": "mllm_data/mllm_data.json",
"formatting": "sharegpt",
"columns": {
"messages": "messages",
"images": "images"
},
"tags": {
"role_tag": "role",
"content_tag": "content",
"user_tag": "user",
"assistant_tag": "assistant"
}
},8.2 配置训练参数
访问LLaMaFactory的web页面,配置微调的训练参数:
- Model name:
Qwen2-VL-2B-Instruct - Model path:
models/Qwen2-VL-2B-Instruct - Finetuning method:
lora - Stage :
Supervised Fine-Tuning - Dataset:
mllm_med - Output dir:
saves/Qwen2-VL/lora/Qwen2-VL-sft-demo1
配置参数中最好将
save_steps设置大一点,否则训练过程会生成非常多的训练日志,导致硬盘空间不足而训练终止。
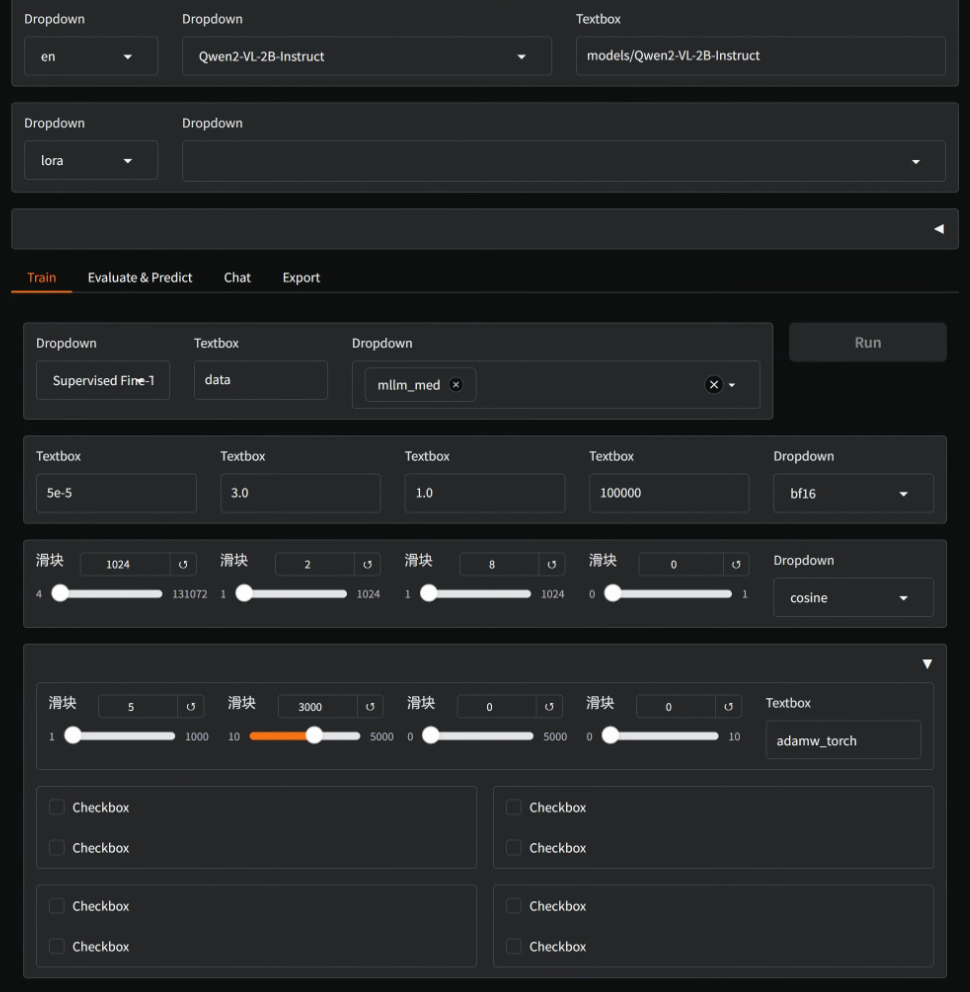
点击Preview Command预览命令行无误后,点击Run按钮开始训练。
训练参数:
python
llamafactory-cli train \
--do_train True \
--model_name_or_path models/Qwen2-VL-2B-Instruct \
--preprocessing_num_workers 16 \
--finetuning_type lora \
--template qwen2_vl \
--flash_attn auto \
--dataset_dir data \
--dataset mllm_med \
--cutoff_len 1024 \
--learning_rate 5e-05 \
--num_train_epochs 3.0 \
--max_samples 100000 \
--per_device_train_batch_size 2 \
--gradient_accumulation_steps 8 \
--lr_scheduler_type cosine \
--max_grad_norm 1.0 \
--logging_steps 5 \
--save_steps 3000 \
--warmup_steps 0 \
--optim adamw_torch \
--packing False \
--report_to none \
--output_dir saves/Qwen2-VL-2B/full/Qwen2-VL-sft-demo1 \
--bf16 True \
--plot_loss True \
--ddp_timeout 180000000 \
--include_num_input_tokens_seen True \
--lora_rank 8 \
--lora_alpha 16 \
--lora_dropout 0 \
--lora_target all训练过程 :

训练的过程中,可以通过
watch -n 1 nvidia-smi实时查看GPU显存的消耗情况。
经过35小时的训练,模型训练完成,损失函数如下:
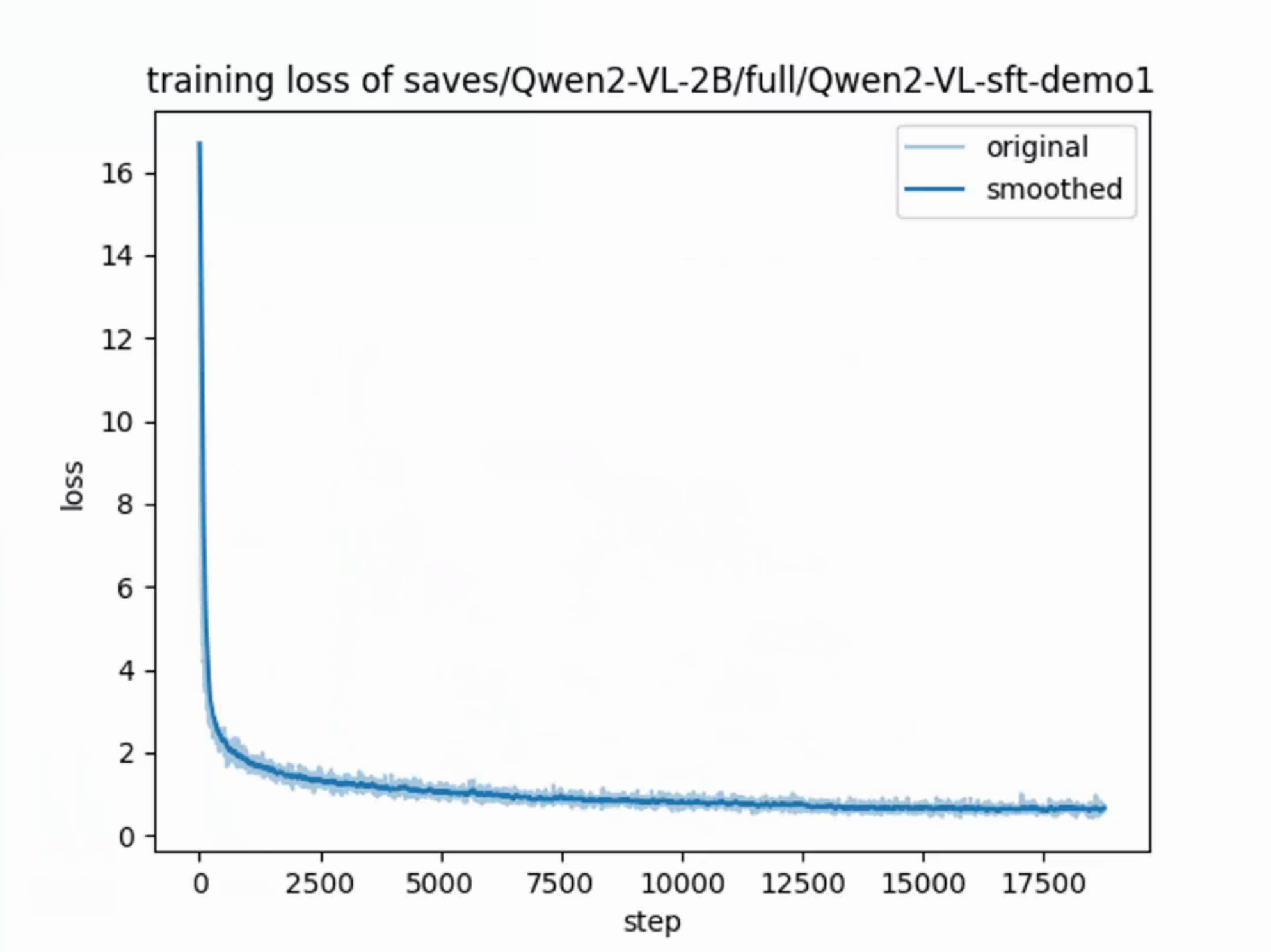
损失函数一般降低至1.2左右,太低会导致模型过拟合。
8.3 合并导出模型
接下来,我们将 Lora补丁 与 原始模型 合并导出:
- 切换到
Expert标签下 - Model path: 选择Qwen2-VL的基座模型,即:
models/Qwen2-VL-2B-Instruct - Checkpoint path: 选择lora微调的输出路径,即
saves/Qwen2-VL/lora/Qwen2-VL-sft-demo1 - Export path:设置一个新的路径,例如:
Qwen2-VL-sft-final - 点击
开始导出按钮
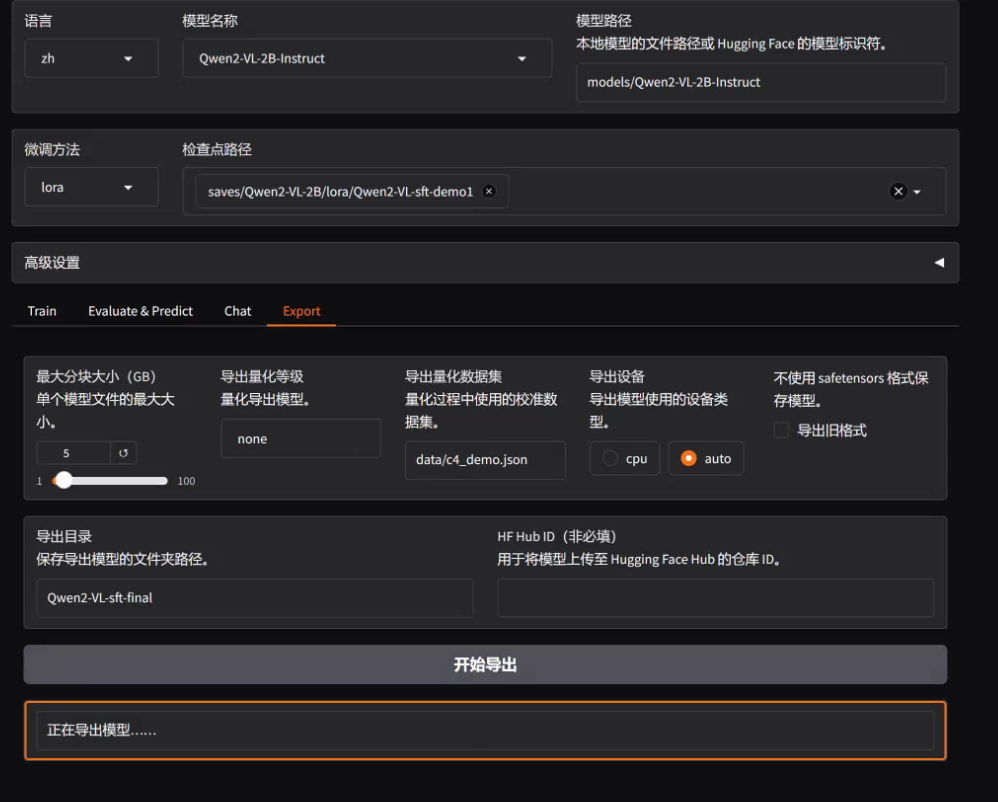
导出完毕后,会在LLaMaFactory的根目录下生成一个 Qwen2-VL-sft-final 的文件夹。
9. 模型验证
9.1 模型效果对比
第一步:在LLaMa Factory中卸载之前的模型
第二步 :在LLaMa Factory中加载导出的模型,并配置模型路径为 Qwen2-VL-sft-final
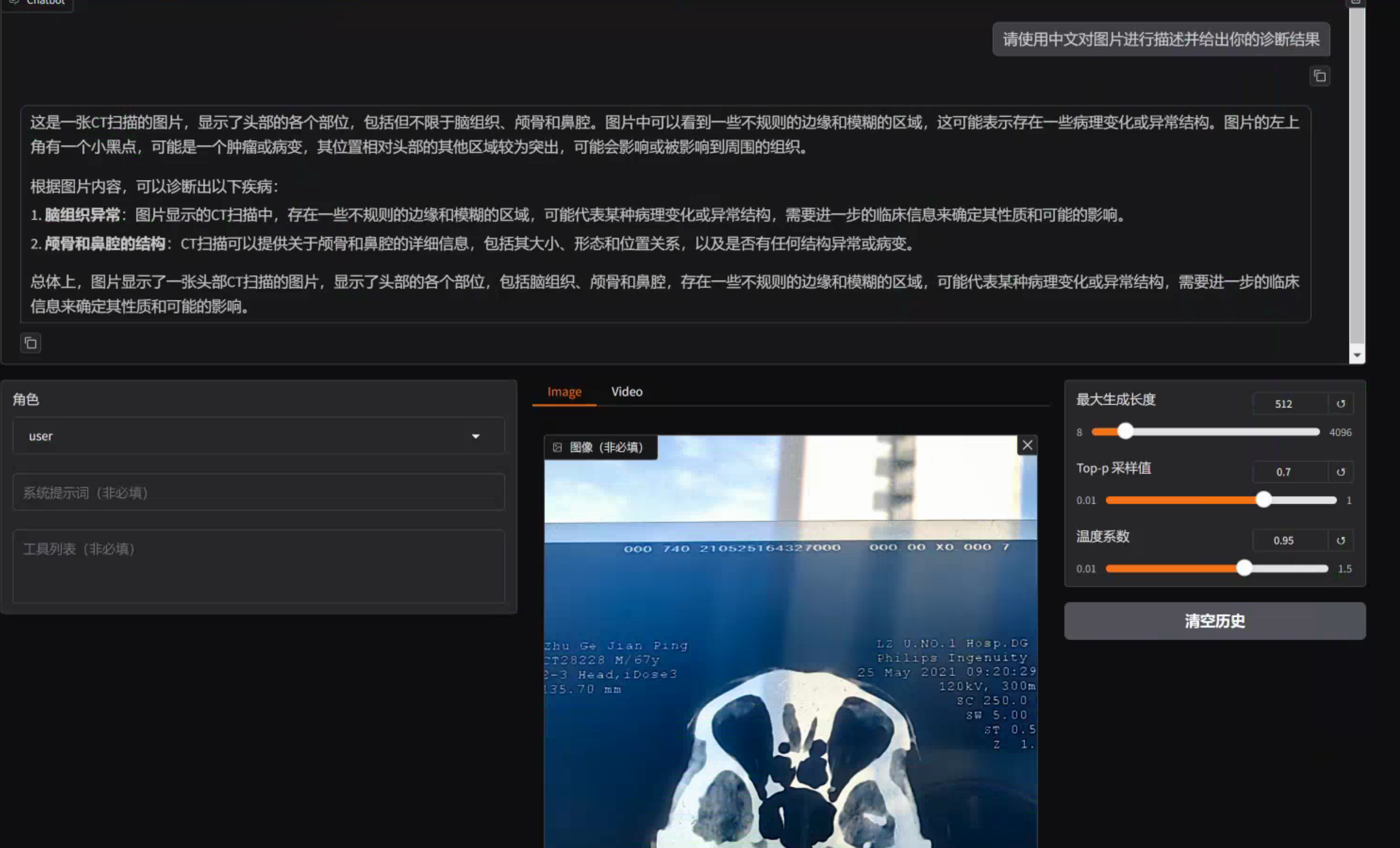
第三步:加载模型并上传之前的CT图片提问同样的问题
可以看到,经过微调后的模型,可以给出具体区域存在的可能异常问题。
9.2 实际诊断
接下来,我将使用微调后的模型,为家里老爷子的CT片做诊断,看看模型给出的诊断与大夫的异同点。
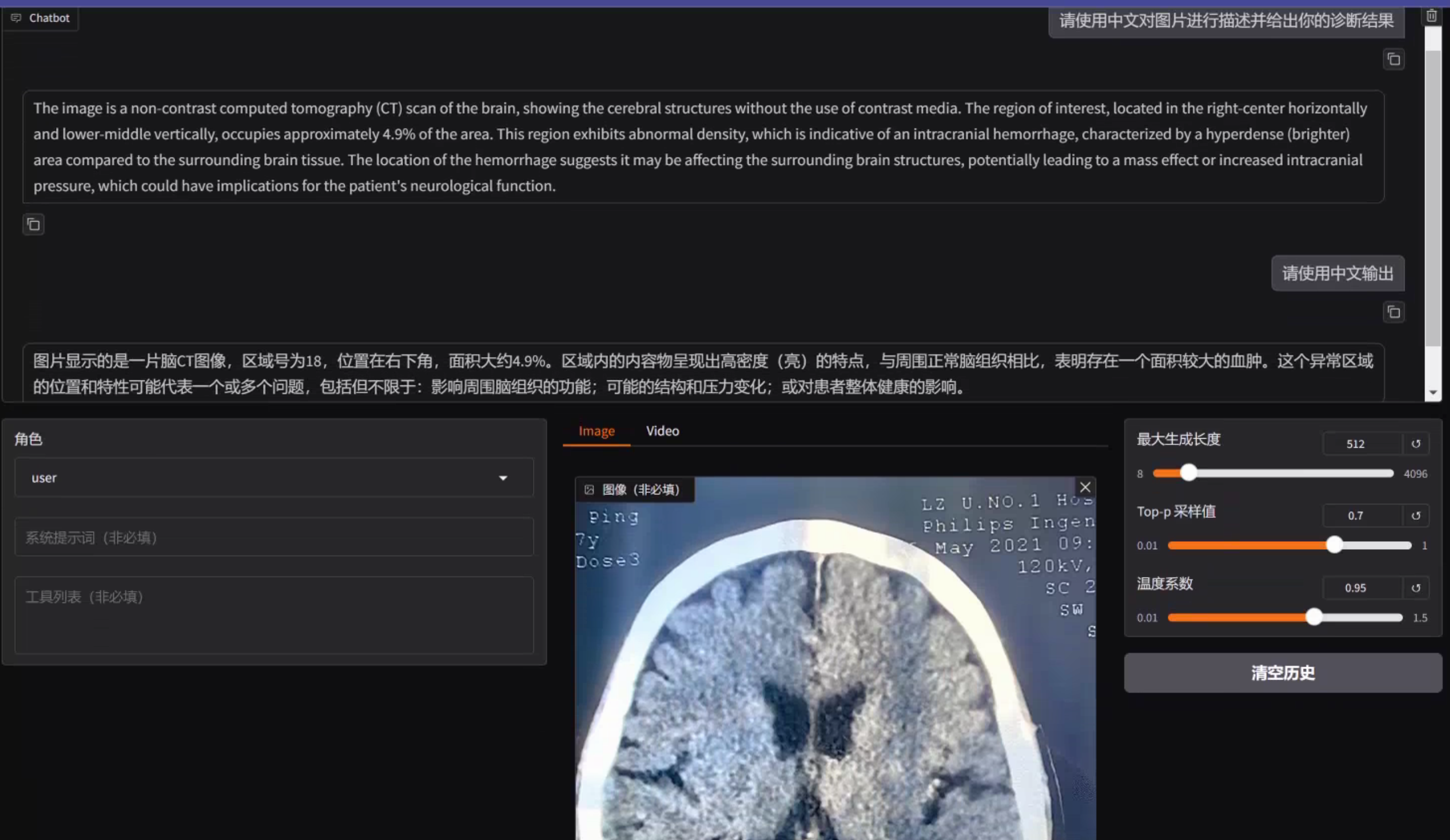
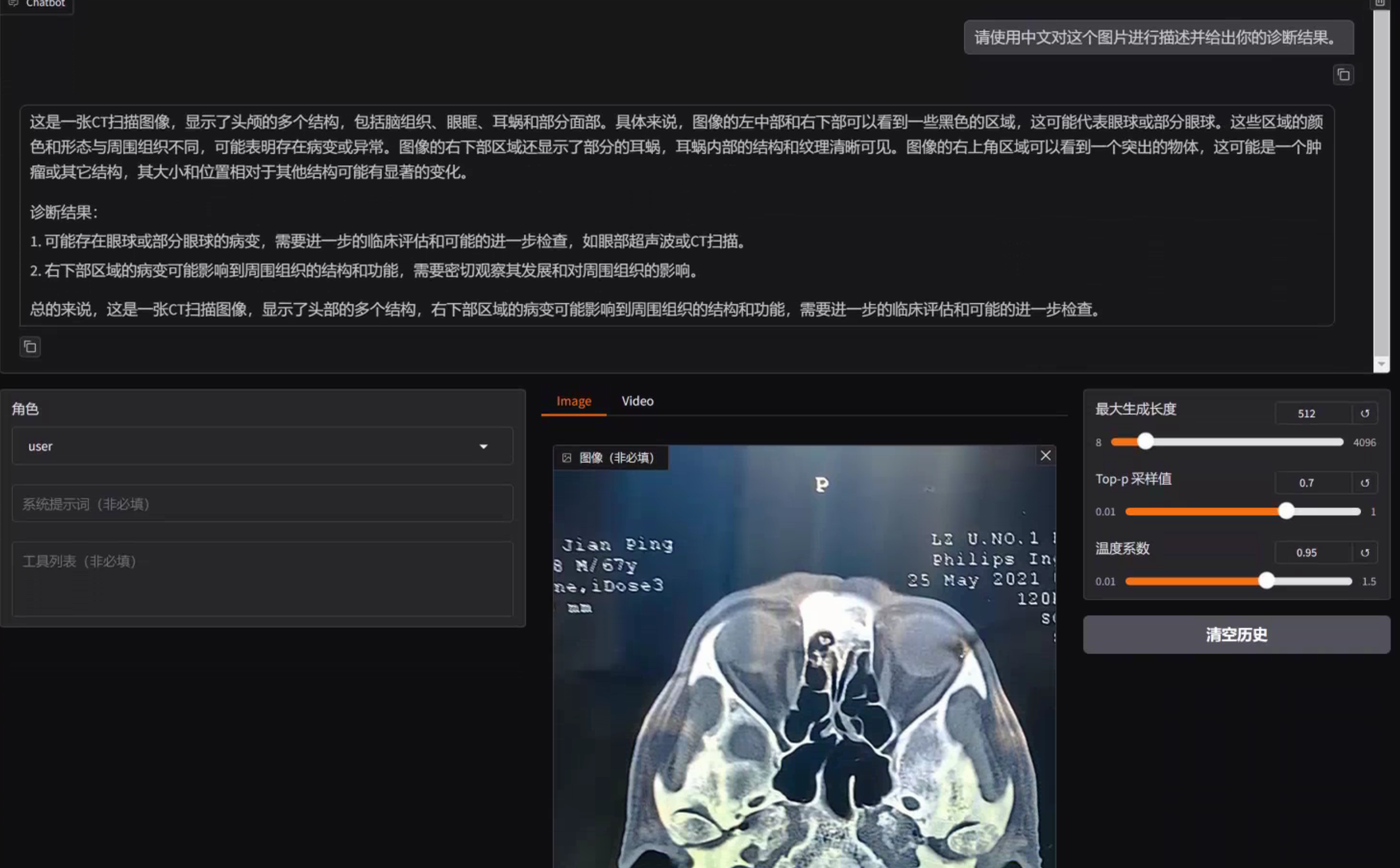
我总计测试了CT片上的52张局部结果,其中具有代表性的为上述三张,可以看到模型还是比较准确地诊断出:脑部有垂体瘤,可能会影响到眼部。这与大夫给出的诊断和后续检查方案一致。
不足之处
训练集:
- 多模态:本次训练只是采用了MedTrinity-25Mdemo数据集,如果使用MedTrinity-25Mfull数据集,效果应该会更好。
- 中英文:本次训练集中使用的MedTrinity-25Mdemo数据集,只包含了英文数据,如果将英文标注翻译为中文,提供中英文双文数据集,相信效果会更好。
- 对话数据集 :本次训练只是使用了多模态数据集,如果增加中文对话(如:中文医疗对话数据-Chinese-medical-dialogue),相信效果会更好。
前端页面:
- 前端页面 :本次实践曾使用streamlit构建前端页面,以便图片上传和问题提出,但是在加载微调后的模型时,会出现:
ValueError: No chat template is set for this processor问题,所以转而使用LLaMaFactory的web页面进行展示。 - 多个图片推理 :在Qwen2-VL的官方指导文档中,提供了
Multi image inference方法,本次未进行尝试,相信将多个图片交给大模型进行推理,效果会更好。
内容小结
- Qwen2-VL-2B作为多模态大模型,具备有非常强的多模态处理能力,除了能够识别图片内容,还可以进行相关的推理。
- 我们可以通过
LLaMaFactory对模型进行微调,使得其具备医疗方面的处理能力。 - 微调数据集采用开源的MedTrinity-25M数据集,该数据集有两个版本:25Mdemo和25Mfull。
- 训练前需要对数据集进行预处理,使得其适配LLaMaFactory的微调格式。
- 经过微调后的多模态大模型,不但可以详细地描述图片中的内容,还可以给出可能的诊断结果。