
Scikit-bio:用于生物组学数据分析的基础Python库
研究论文
● 期刊:Nature Methods, IF[32.1]
● DOI:10.1038/s41592-025-02981-z
● 原文链接:
https://www.nature.com/articles/s41592-025-02981-z
● 发表日期:2025-12-11
● 第一作者:Matthew Aton
● 通讯作者:诸奇赟 (qiyun.zhu@asu.edu)
● 主要单位:亚利桑那州立大学坦佩分校、美国加利福尼亚大学圣地亚哥分校、美国科罗拉多大学博尔德分校、瑞士苏黎世联邦理工学院、美国北亚利桑那大学弗拉格斯塔夫分校、美国博尔德 Gutz Analytics 有限责任公司、加拿大滑铁卢大学、美国威斯康星大学麦迪逊分校、美国史蒂文斯理工学院
正文Main
现代生物学研究以多种"组学"数据类型为特征,这些数据旨在描述生物实体的整体,如基因组学、转录组学、蛋白质组学、代谢组学和宏基因组学。它们为复杂生物系统提供了前所未有的洞察,但也带来了持续的分析挑战,包括高维度(特征数量远多于样本数量)、稀疏性(大多数特征为零)以及组成性(样本内特征相互依赖)。尽管存在这些挑战,组学数据也带来了一个独特机会:生物特征之间可通过基于知识的、通常呈树状结构的图(如系统发育树和功能分类)相互连接,这些结构可被用来增强分析效果。这些特点使得通用数据分析方法难以胜任组学数据的处理。
为满足这一需求,我们推出了 scikit-bio,一个面向组学数据分析的 Python 生物信息学库(图 1)。scikit-bio 提供超过 500 个公开函数、类和方法,构成了一套全面的数据结构与算法,旨在应对组学固有的基本分析挑战与机遇。虽然 scikit-bio 也支持基础序列分析,但其真正优势在于对"样本×特征"表格及相关树结构的分析,而这两种数据表示正是众多组学研究的共通之处。这一独特定位使 scikit-bio 既区别于 BioPython 等通用工具包,也不同于 ScanPy等 domain-specific 软件包。scikit-bio 还充当桥梁,将最初多诞生于 R 语言的组学方法,与通常在 Python 中实现的高级机器学习框架连接起来(附表 1)。
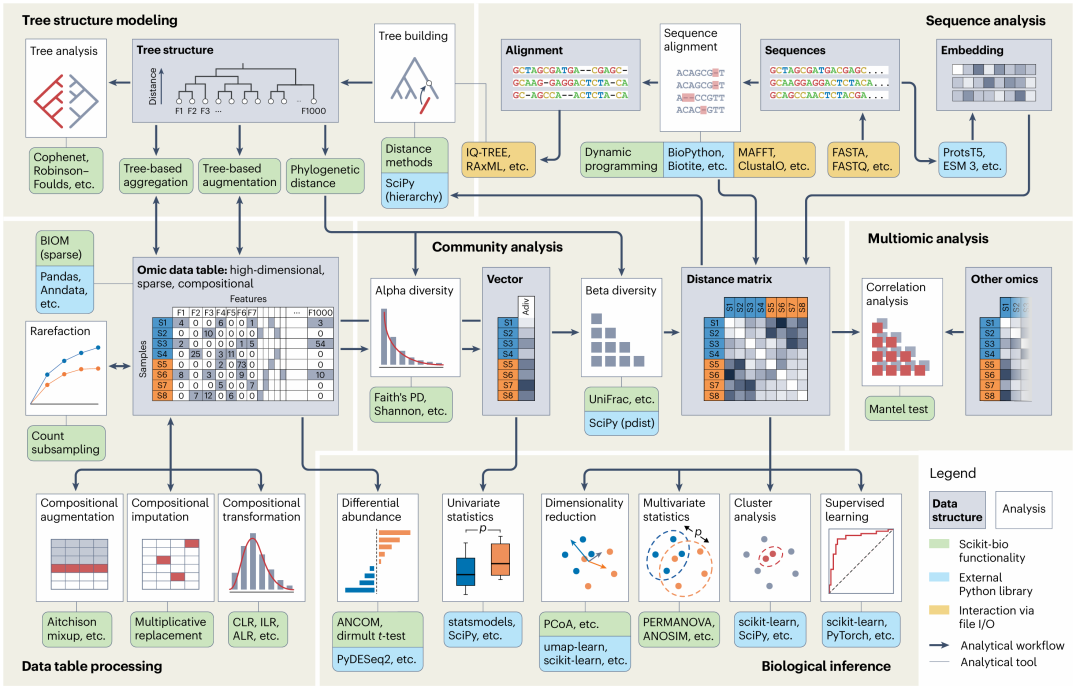
图1 | Scikit-bio在生物组学数据分析中的能力及其与Python科学计算生态系统中其他工具的交互关系
Fig. 1 | Scikit-bio's capacities for biological omic data analysis and its interactions with other tools in the Python scientific computing ecosystem
带插图的方框表示数据结构(浅灰背景)或分析流程(白色背景);圆角框则表示具体的分析工具,并按以下方式分类:
-
绿色:已在 scikit-bio 中实现的功能;
-
蓝色:由外部 Python 库提供的功能,可在 Python 框架内与 scikit-bio 联用------例如,scikit-bio 生成的距离矩阵可输入 SciPy 进行层次聚类、输入 scikit-learn 做 k 近邻分类,或输入 umap-learn 进行 UMAP 降维;
-
黄色:由非 Python 程序提供的功能,通过文件读写(I/O)与 scikit-bio 交互。例如,scikit-bio 可读取 RAxML 构建的系统发育树,或读取 MAFFT 生成的多序列比对结果。
为应对组学数据的稀疏性挑战,scikit-bio 内置了 Biological Observation Matrix(BIOM)格式,一种专为表格数据设计的稀疏矩阵结构。BIOM 由团队成员发起并维护,已成为微生物组研究的事实标准。针对数据的组成性,scikit-bio 提供多种变换方法,如中心对数比(CLR)以及处理零值的多重替换法,并实现了考虑组成性的差异丰度检验,例如微生物组组成分析(ANCOM)。同时,库内集成了丰富的多样性度量,可计算样本内(α)与样本间(β)生物多样性;后者生成距离矩阵,可直接用于主坐标分析(PCoA)等非欧嵌入技术,或置换多因素方差分析(PERMANOVA)等多变量检验,以评估组学特征与表型的关联。此外,scikit-bio 实现了 Mantel 检验,用于跨组学相关性评估。
在树结构方面,库提供构建、遍历与操作树的全套功能,可计算特征或类群间距离,并将树整合到群落建模中(如 Faith 系统发育多样性 PD 与 UniFrac 度量),从而把先验生物学知识纳入高维数据分析,使原本难以解析的问题变得可解。
多项实现处于 Python 生态的领先或独有地位(附表 1),并针对当代及新兴研究中动辄数十万样本的超大规模数据做了性能优化(如对 >10 万份样本的 PCoA 分析)。
scikit-bio 与 Python 科学计算栈无缝衔接:API 设计遵循现代风格与原则,大量功能基于 NumPy 数组、SciPy 稀疏矩阵和 Pandas DataFrame 等高效结构,用户无需复杂转换即可直接调用;分析结果可立即对接 statsmodels(统计建模)、scikit-learn(机器学习)、matplotlib(可视化)等外部库。灵活的 I/O 体系使其能通过文件与非 Python 生信流程互操作。
自 2014 年发布以来,scikit-bio 已支撑大量研究与工具开发,尤其是广受欢迎的微生物组分析平台 QIIME 1/2 的众多核心功能。尽管起源于微生物组领域,其抽象设计充分利用了组学数据的共性;作为独立库,它采用模块化架构,用户可按需选取组件,插入各种组学工作流。其应用已扩展至单细胞分析、结构变异检测、代谢组学、表观基因组学等诸多领域,相关论文被引用数万次(附表 2、3)。
项目坚持工业级质量标准:单元测试覆盖率 98%,配套完整 doctests 与严格代码风格检查;在线文档详述数学背景、生物学应用与示例代码,并提供结构化教程。scikit-bio 还被选为生物信息学教材的基础,降低跨学科门槛,促进协作。
scikit-bio 采用社区驱动模式,迄今已有 80 余名开发者贡献代码,确保库功能随科学计算前沿持续演进。团队致力于不断扩展与优化,以支持更新、更广泛的研究场景。我们相信,这一基础软件库将继续惠及全球科研社区。
作者简介
诸奇赟 (通讯作者)

诸奇赟是一位计算生物学家,专长于系统发生学、比较基因组学、宏基因组学和多组组学。他的研究兴趣包括开发计算工具和资源以提升微生物组数据分析,研究各种宿主和环境微生物组及其对人类健康的影响,以及微生物与宿主的进化研究,重点关注生物体之间的垂直和水平遗传联系。他领导或做出了多项微生物组研究项目的重要贡献,涵盖病理学、营养学和人类学等多个主题,来源多样,包括宿主肠道、癌症组织、建筑环境、海洋等。他致力于通过微生物世界的多层次------基因、基因组、谱系、群落、宿主和环境------扩展对微生物世界的理解。
信息来源:
https://search.asu.edu/profile/3737104
翻译:杨海飞,青岛农业大学,基因组所联培硕士在读
审核:朱志豪,广东医科大学,基因组所联合博士后
排版:曾美尹,中国农科院基因组所硕士在读
终审:刘永鑫,中国农科院基因组所,研究员/博导
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
iMeta高引 fastp PhyloSuite ImageGP2iNAP2 ggClusterNet2
iMeta工具 SangerBox2 美吉2024 OmicStudioWekemo OmicShare
iMeta综述 高脂饮食菌群 发酵中药 口腔菌群 微塑料 癌症 宿主代谢
10000+:扩增子EasyAmplicon 比较基因组JCVI 序列分析SeqKit2 维恩图EVenn