文章目录
介绍
单细胞技术能够进行高分辨率的细胞研究,但因数据复杂性而面临识别差异特征的难题。
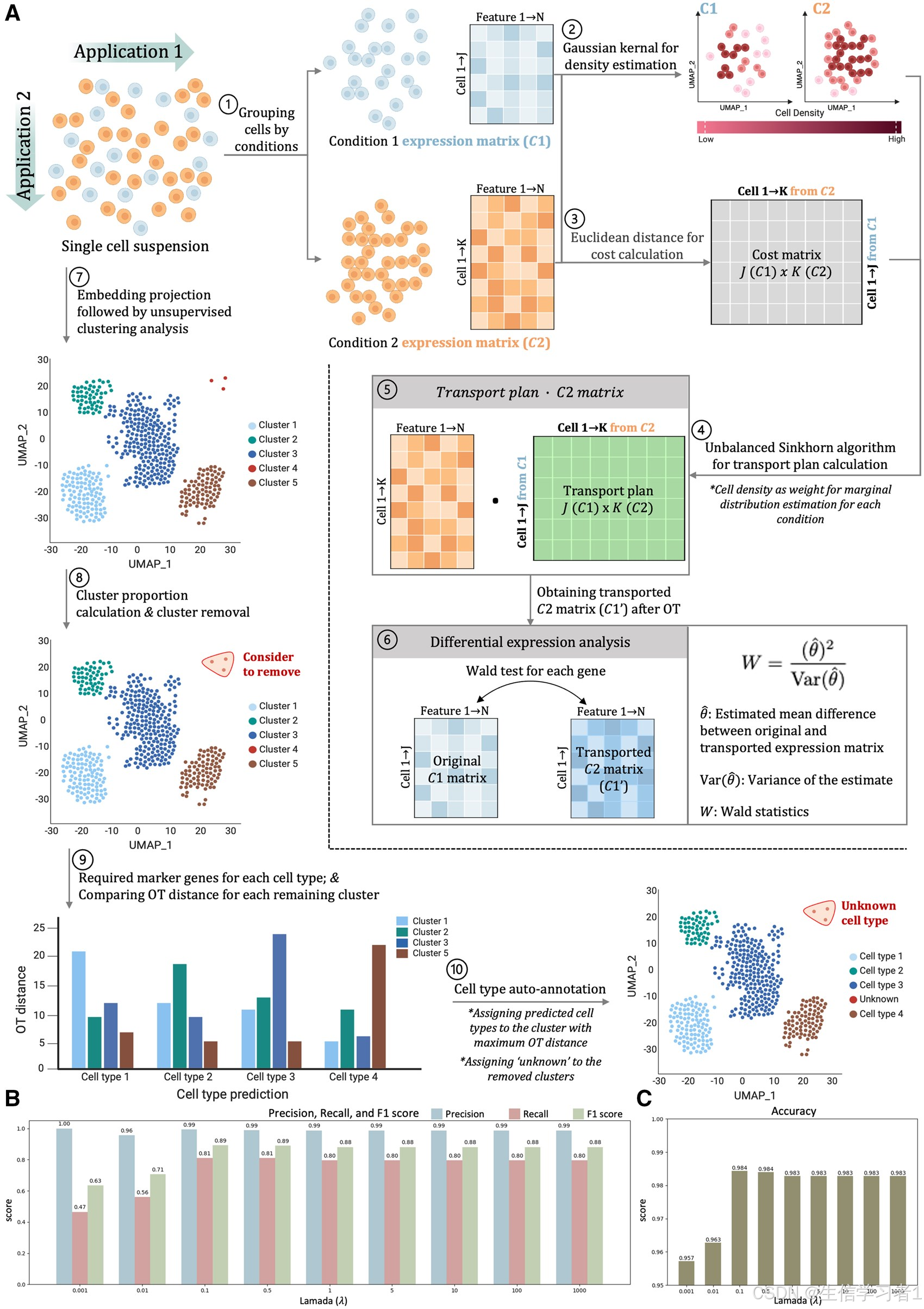
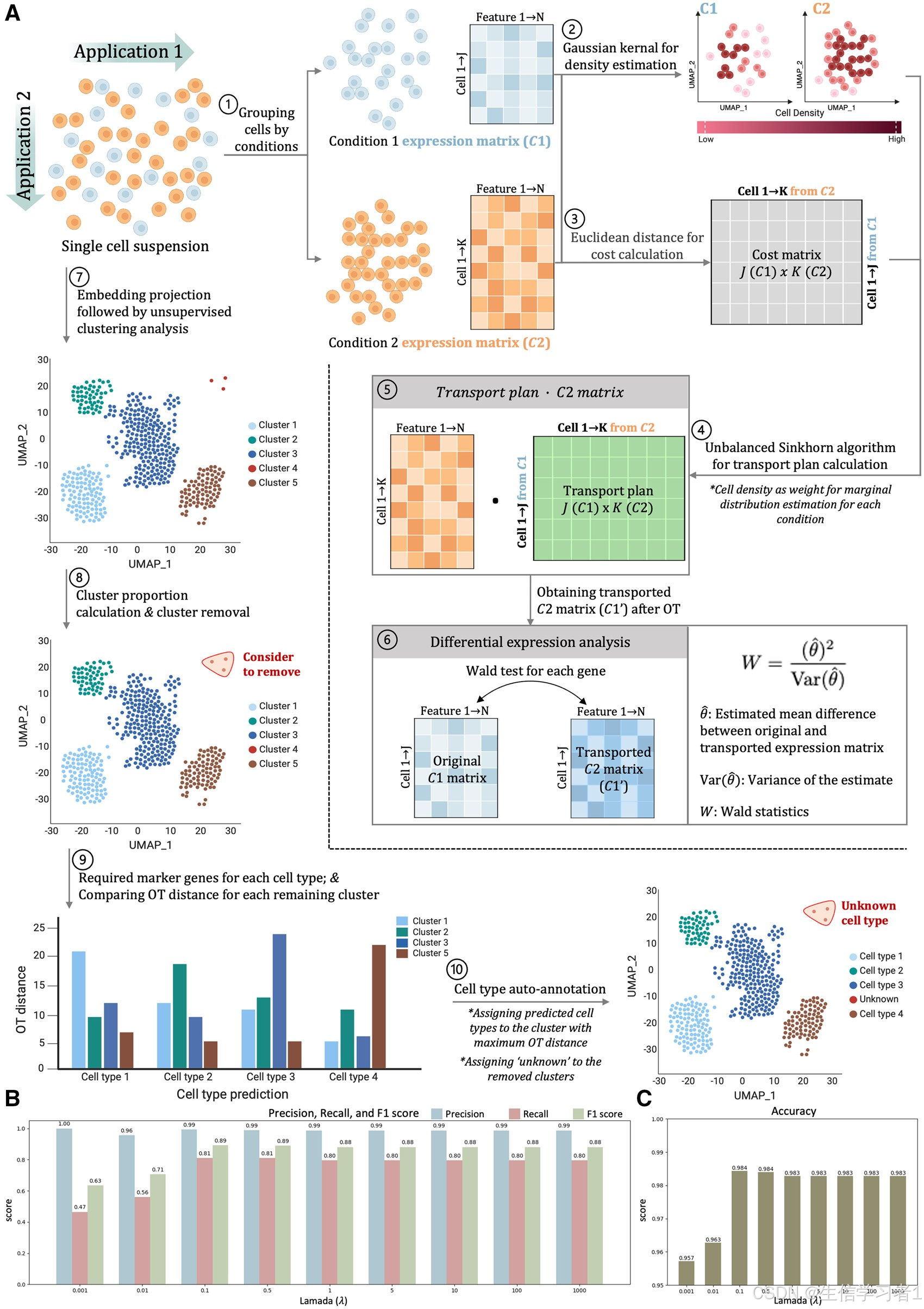
我们提出了 OTMODE 这一非参数方法,它采用了不平衡的辛普朗算法和瓦尔德检验,旨在提升单细胞多组学数据中的差异特征识别能力。在模拟实验中,OTMODE 表现出了卓越的性能(平均 F1 分数为 90%;平均 AUC 分数为 92%),并且效率很高(处理 5000 个细胞仅需 2.2 秒)。在实际应用中,它在检测有意义的过程方面比其他最先进的方法更具敏感性,并且能够通过识别自动注释工具中可能存在的错误注释簇来评估注释准确性。此外,OTMODE 与 Scanpy 兼容性极佳,为研究人员提供了一个用户友好的解决方案。
单细胞组学技术的迅速发展为疾病和实验操作所引起的扰动提供了前所未有的分辨率。这些扰动会导致染色质状态和转录活动的改变,而这些变化可以通过单细胞技术检测到。具体而言,单细胞 RNA 测序(scRNA-seq)揭示了组织内的转录异质性,揭示了不同的细胞状态(洛戈尼等,2021 年)。单细胞 ATAC 测序(scATAC-seq)为染色质可及性的研究提供了一个独特的视角,将表观遗传调控与单个细胞的转录动态联系起来(布恩罗斯特罗等人,2015 年)。
为了检测这些变化,传统的方法,如 t 检验和威尔科克森秩和检验,由于单细胞数据的高稀疏性而存在灵敏度降低的问题(Stegle 等人,2015 年)。为了解决诸如随机失活和特征性双峰表达分布等问题,专门的方法,如 Monocle3(Qiu 等人,2017 年)和 MAST(Finak 等人,2015 年)已被开发出来,用于检测不同条件下的差异表达基因。然而,这些方法存在严重的局限性:(1)偏向于高表达基因,而对低表达水平的基因不敏感(Soneson 和 Robinson,2018 年,Kharchenko,2021 年,Wu 等人,2025 年);(2)与主流工具(如 Seurat(Hao 等人,2024 年)和 Scanpy(Wolf 等人,2018 年))的互操作性较差。此外,最近的 Memento 方法在图谱级别的数据集上表现出高灵敏度和计算效率(Kim 等人,2024 年),然而,其核心的高斯分布假设对于小数据集尚未得到验证。
伪批量分析方法,例如 DESeq2(Love 等人,2014 年),通过将样本内细胞的基因表达进行汇总来规避稀疏性问题(Hafemeister 和 Halbritter,2023 年),但会掩盖细胞异质性(Hu 和 Chikina,2024 年),并且会增加分析的复杂性。针对转录组数据开发的模型在单细胞 ATAC 测序数据上的表现往往不尽如人意,这是由于其基础数据分布存在根本差异(Zhao 等人,2024 年)。总的来说,这些方法上的缺陷阻碍了对差异特征的准确识别,并可能延缓生物学发现的进程。
在区分特征识别方面,另一个关键挑战在于缺乏标准化的标注流程(Heumos 等人,2023 年;Nouri 等人,2023 年)。单细胞标注是通过特定簇的差异表达基因(DEGs)和标准细胞标志物来确定细胞类型的(克拉克等人,2021 年)。然而,高维数据的复杂性掩盖了有意义的生物学簇与技术误差之间的区别(范登邦和迪埃兹,2020 年)。尽管如今已经开发出了自动标注方法,如 CellTypist(多明吉·孔德等人,2022 年),但这些方法很大程度上依赖于参考数据库的质量,并且有时会导致标注的模糊性(帕斯奎尼等人,2021 年),这就需要专家的监督参与。这种对研究人员经验的主观依赖可能会危及后续分析的有效性。因此,需要一个定量指标来评估标注的准确性。
为解决上述挑战,我们引入了 OTMODE 这一基于 Python 的框架,该框架利用最优传输(OT)理论来检测单细胞数据中的差异特征,并提高注释的准确性。OT 理论提供了一种基于原则的方法来比较概率分布,通过找到将一个分布转换为另一个分布的最有效方式来实现。这使得 OT 特别适合单细胞数据分析,因为它自然地处理了固有的稀疏性,通过关注分布的实际支持而非要求在整个特征空间中进行密集表示而无需特定于维度的假设。此外,OT 的强大泛化能力使其能够轻松扩展到高维空间,而无需特定于维度的假设(阿尔特舒勒等人,2017 年;吉内瓦伊等人,2018 年;韦德和巴赫,2019 年),使其非常适合应用于通常包含数万个特征的现代单细胞数据集。由于 OT 的几何特性保留了高维数据的内在结构,其捕捉复杂分布差异的能力使其非常适合单细胞测量的异质性。
在此,我们展示了 OTMODE 的有效性:(1)在单细胞多组学数据中,能够灵敏且稳健地识别不同条件下的差异特征;(2)通过一种新的度量标准,能够通过量化正标记和负标记对每个细胞类型的总体贡献来实现注释的改进;(3)与 Scanpy 和 scverse 生态系统(Virshup 等人,2023 年)无缝集成,以方便后续分析。我们在模拟和真实的大规模单细胞 RNA 测序和单细胞 ATAC 测序数据集上验证了 OTMODE(补充数据 1,可在生物信息学在线网站的补充数据中获取),证明了细胞类型的显著改善。
代码
https://github.com/Eggong/OTMODE
参考
- OTMODE: an optimal transport theory-based framework for identifying differential features in single-cell multi-omics data
- https://github.com/Eggong/OTMODE