温馨提示:文末有 CSDN 平台官方提供的学长 QQ 名片 :)
1. 项目简介
肺癌是全球范围内导致癌症死亡的主要原因之一,早期检测和诊断对于提高患者生存率至关重要。随着深度学习技术的迅猛发展,基于CT影像的肺癌检测识别成为了研究热点。本文介绍了一种基于深度学习的CT影像肺癌检测识别系统。该系统使用TensorFlow和Keras深度学习框架,通过迁移学习技术,以EfficientNetB3为基础模型,构建了一个卷积神经网络。通过对数据集进行扩充,利用扩充后的数据集进行模型训练,并进行模型性能评估,并利用 Flask + Bootrap + Ajax 搭建交互式分析框架,实现脑部 MRI 扫描影像上传和在线预测,模型给出是否包含脑部肿瘤及肿瘤类型,整体准确率达到90.27%。
B站详情与代码下载:基于深度学习的CT影像肺癌检测识别_哔哩哔哩_bilibili
基于深度学习的CT影像肺癌检测识别
2. 肺癌CT影像数据读取
利用 TensorFlow 的 ImageDataGenerator 创建训练、验证和测试数据的生成器:
python
def create_gens(train_df, valid_df, test_df, batch_size):
"""
定义一个函数create_gens,用于创建训练、验证和测试数据的生成器。
参数:
train_df -- DataFrame,包含训练数据的文件路径和标签
valid_df -- DataFrame,包含验证数据的文件路径和标签
test_df -- DataFrame,包含测试数据的文件路径和标签
batch_size -- 整数,表示每次迭代读取的样本数量
返回值:
train_gen -- 训练数据的生成器
valid_gen -- 验证数据的生成器
test_gen -- 测试数据的生成器
"""
# 设置图像的大小和通道数
img_size = (224, 224) # 图像的大小
channels = 3 # 图像的通道数
img_shape = (img_size[0], img_size[1], channels) # 图像的形状
# 计算测试数据的批量大小和步数
ts_length = len(test_df) # 测试数据的数量
test_batch_size = max(sorted([ts_length // n for n in range(1, ts_length + 1) if ts_length % n == 0 and ts_length / n <= 80])) # 测试数据的批量大小
test_steps = ts_length // test_batch_size # 测试数据的步数
# 定义一个函数,用于对图像进行预处理
def scalar(img):
return img # 这里只是一个示例,实际的预处理函数可能会更复杂
# 创建训练数据的生成器
tr_gen = ImageDataGenerator(preprocessing_function=scalar, horizontal_flip=True) # 使用ImageDataGenerator创建生成器,可以进行数据增强
train_gen = tr_gen.flow_from_dataframe(train_df, x_col='filepaths', y_col='labels', target_size=img_size, class_mode='categorical',
color_mode='rgb', shuffle=True, batch_size=batch_size) # 从DataFrame中读取数据,生成训练数据
# 创建验证数据的生成器
valid_gen = tr_gen.flow_from_dataframe(valid_df, x_col='filepaths', y_col='labels', target_size=img_size, class_mode='categorical',
color_mode='rgb', shuffle=True, batch_size=batch_size) # 从DataFrame中读取数据,生成验证数据
# 创建测试数据的生成器
test_gen = tr_gen.flow_from_dataframe(test_df, x_col='filepaths', y_col='labels', target_size=img_size, class_mode='categorical',
color_mode='rgb', shuffle=False, batch_size=test_batch_size) # 从DataFrame中读取数据,生成测试数据
return train_gen, valid_gen, test_gen # 返回三个生成器:训练、验证和测试数据的生成器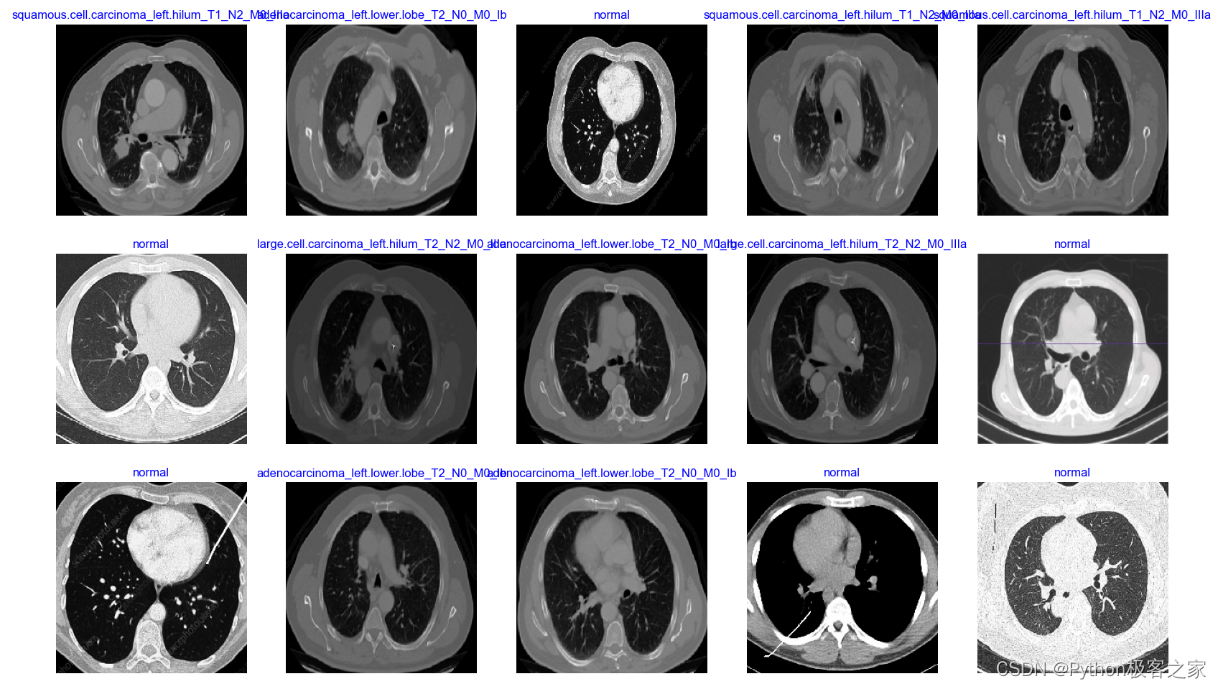
3. 构建肺癌检测的卷积神经网络
在构建深度学习模型时,可以选择多种现有的神经网络架构,如VGG16、ResNet、InceptionV3等,或者使用更先进的模型如EfficientNetB3。这些模型可以通过迁移学习的方式进行微调,以适应特定的脑部肿瘤检测任务。
python
# 创建一个预训练的EfficientNetB3模型,不包括顶部的全连接层,使用ImageNet的权重,输入形状为img_shape,池化层使用最大池化
base_model = tf.keras.applications.efficientnet.EfficientNetB3(
include_top=False, weights="efficientnetb3_notop.h5", input_shape=img_shape, pooling='max'
)
# 创建一个顺序模型,包含以下层:
# 1. 预训练的base_model
# 2. BatchNormalization层,用于标准化批量数据的激活值
# 3. 全连接层,有256个单元,使用L2和L1正则化以及ReLU激活函数
# 4. Dropout层,丢弃率为0.45,用于减少过拟合
# 5. 另一个全连接层,输出单元数为类别数量,使用softmax激活函数,用于多分类任务
model = Sequential([
base_model,
BatchNormalization(axis=-1, momentum=0.99, epsilon=0.001),
Dense(256, kernel_regularizer=regularizers.l2(l=0.016), activity_regularizer=regularizers.l1(0.006),
bias_regularizer=regularizers.l1(0.006), activation='relu'),
Dropout(rate=0.45, seed=123),
Dense(class_count, activation='softmax')
])
# 编译模型,使用Adamax优化器,学习率为0.001,损失函数为交叉熵,评估指标为准确率
model.compile(Adamax(learning_rate=0.001), loss='categorical_crossentropy', metrics=['accuracy'])
# 打印模型的摘要信息,显示每一层的结构和参数数量
model.summary()4. 模型训练
python
# 创建回调函数列表
checkpoint = tf.keras.callbacks.ModelCheckpoint('efficientnetb3_base_best_weights.h5', monitor='val_accuracy', verbose=1, mode='max',save_best_only=True)
early = tf.keras.callbacks.EarlyStopping(monitor="accuracy", mode="max",restore_best_weights=True, patience=5)
callbacks_list = [checkpoint,early]
history = model.fit(
train_gen,
validation_data=valid_gen,
epochs=25,
shuffle=True,
verbose=True,
callbacks=callbacks_list
)Epoch 1/25
16/16 [==============================] - ETA: 0s - loss: 8.7303 - accuracy: 0.6134
Epoch 1: val_accuracy improved from -inf to 0.42857, saving model to efficientnetb3_base_best_weights.h5
16/16 [==============================] - 115s 5s/step - loss: 8.7303 - accuracy: 0.6134 - val_loss: 10.0141 - val_accuracy: 0.4286
Epoch 2/25
16/16 [==============================] - ETA: 0s - loss: 7.6084 - accuracy: 0.8728
Epoch 2: val_accuracy improved from 0.42857 to 0.51111, saving model to efficientnetb3_base_best_weights.h5
16/16 [==============================] - 67s 4s/step - loss: 7.6084 - accuracy: 0.8728 - val_loss: 8.8468 - val_accuracy: 0.5111
Epoch 3/25
16/16 [==============================] - ETA: 0s - loss: 7.0149 - accuracy: 0.9380
Epoch 3: val_accuracy improved from 0.51111 to 0.54921, saving model to efficientnetb3_base_best_weights.h5
16/16 [==============================] - 70s 4s/step - loss: 7.0149 - accuracy: 0.9380 - val_loss: 8.2572 - val_accuracy: 0.5492
......
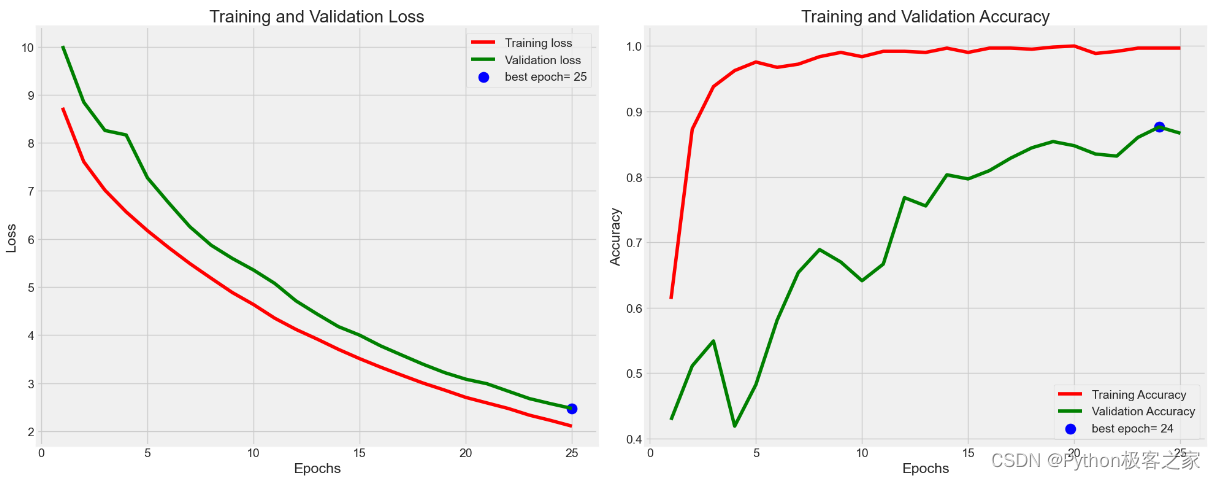
5. 模型预测评估
利用训练集和验证集完成模型的训练和验证后,利用测试集进行预测评估,测试集预测准确率达到 90.28%
python
ts_length = len(test_df)
test_batch_size = test_batch_size = max(sorted([ts_length // n for n in range(1, ts_length + 1) if ts_length%n == 0 and ts_length/n <= 80]))
test_steps = ts_length // test_batch_size
train_score = model.evaluate(train_gen, steps= test_steps, verbose= 1)
valid_score = model.evaluate(valid_gen, steps= test_steps, verbose= 1)
test_score = model.evaluate(test_gen, steps= test_steps, verbose= 1)
print("Train Loss: ", train_score[0])
print("Train Accuracy: ", train_score[1])
print('-' * 20)
print("Validation Loss: ", valid_score[0])
print("Validation Accuracy: ", valid_score[1])
print('-' * 20)
print("Test Loss: ", test_score[0])
print("Test Accuracy: ", test_score[1])
Train Loss: 2.6708874702453613 Train Accuracy: 1.0 -------------------- Validation Loss: 3.06054425239563 Validation Accuracy: 0.8500000238418579 -------------------- Test Loss: 2.887510299682617 Test Accuracy: 0.9027777910232544
绘制模型性能评估结果的混淆矩阵:
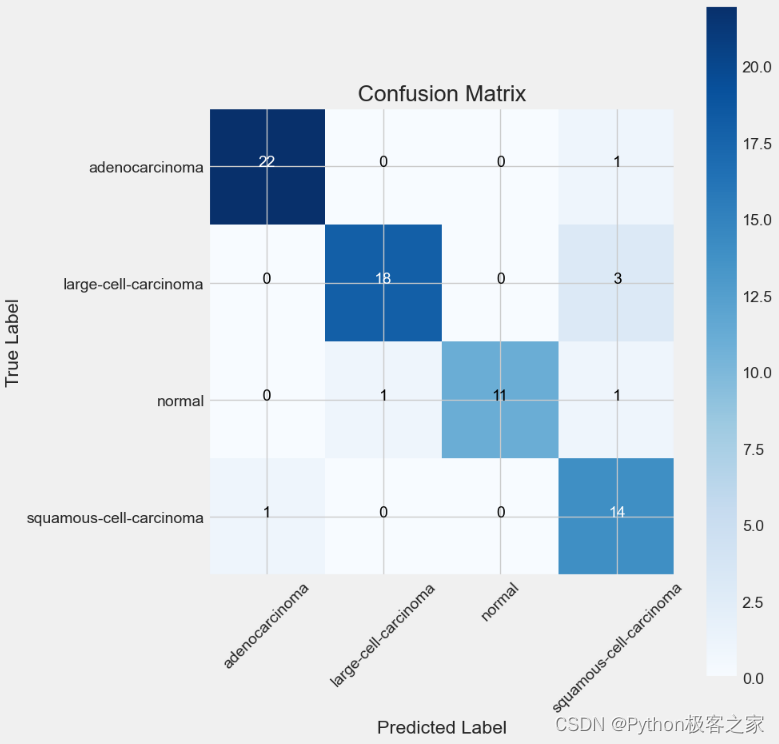
6. CT影像肺癌检测识别系统
6.1 系统首页
6.2 肺癌在线检测
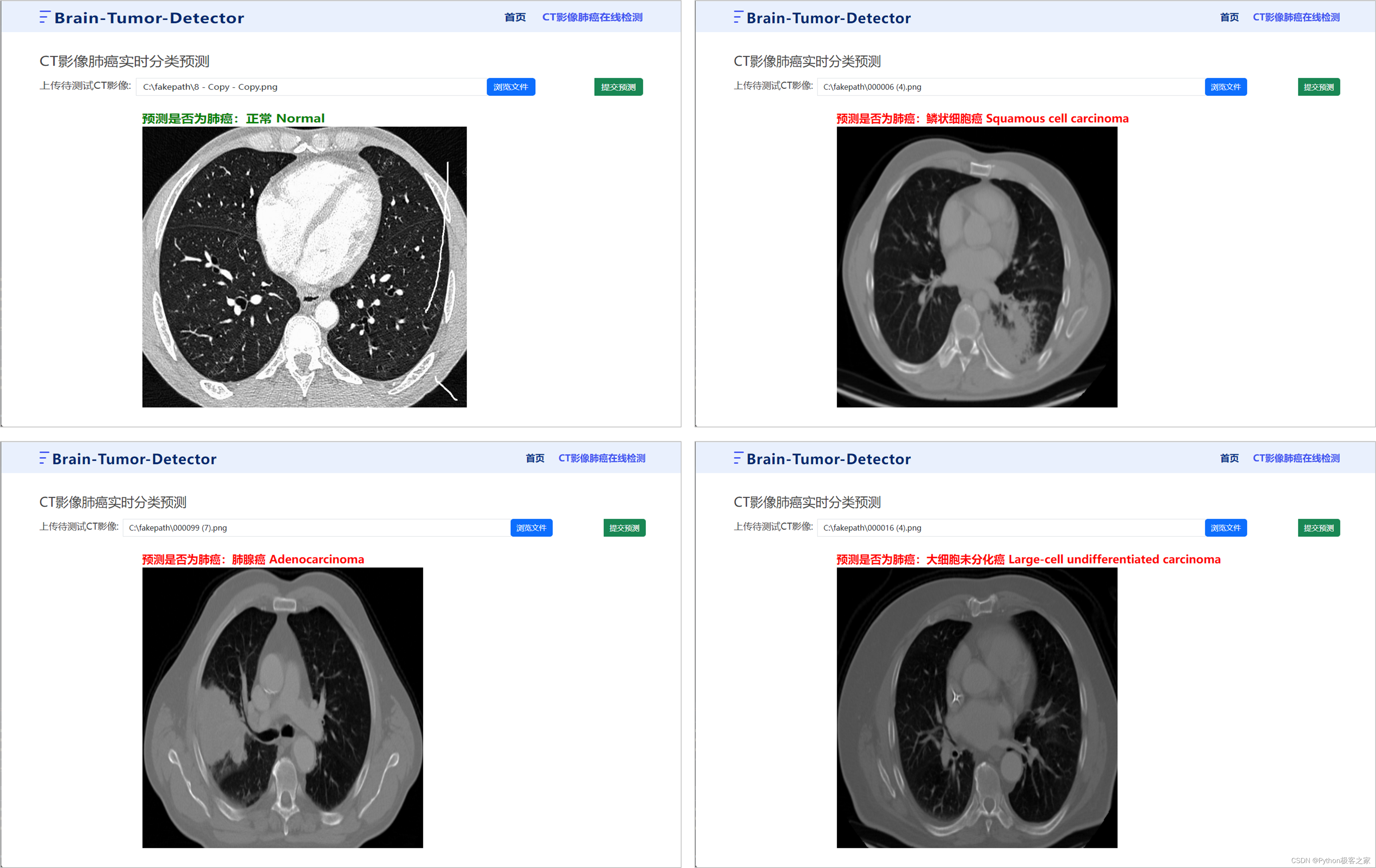
通过上传肺癌的CT影像,点击提交预测,后台加载训练好的模型,预测是否换有肺癌及其肺癌的具体类型:
7. 总结
本文介绍了一种基于深度学习的CT影像肺癌检测识别系统。该系统使用TensorFlow和Keras深度学习框架,通过迁移学习技术,以EfficientNetB3为基础模型,构建了一个卷积神经网络。通过对数据集进行扩充,利用扩充后的数据集进行模型训练,并进行模型性能评估,并利用 Flask + Bootrap + Ajax 搭建交互式分析框架,实现脑部 MRI 扫描影像上传和在线预测,模型给出是否包含脑部肿瘤及肿瘤类型,整体准确率达到90.27%。
欢迎大家点赞、收藏、关注、评论啦 ,由于篇幅有限,只展示了部分核心代码。技术交流、源码获取 认准下方 CSDN 官方提供的学长 QQ 名片 :)
精彩专栏推荐订阅:
