小罗碎碎念
今天分析Nature Medicine病理AI系列的第二篇文章------《Towards a general-purpose foundation model for computational pathology》
昨天分享的模型是CONCH,今天分享的模型是UNI,都来自于哈佛大学Faisal Mahmood课题组。
这篇文章介绍了一个名为UNI的新型自监督模型,它在大规模病理学图像数据集上预训练,能够在多种计算病理学任务中实现卓越的性能。

| 作者角色 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Richard J. Chen | Department of Pathology, Brigham and Women's Hospital, Harvard Medical School | 哈佛医学院,布莱根妇女医院,病理学系 |
| 第一作者 | Tong Ding | Harvard John A. Paulson School of Engineering and Applied Sciences, Harvard University | 哈佛大学,约翰·A·保尔森工程与应用科学学院 |
| 第一作者 | Ming Y. Lu | Electrical Engineering and Computer Science, Massachusetts Institute of Technology (MIT) | 麻省理工学院(MIT),电气工程与计算机科学系 |
| 通讯作者 | Faisal Mahmood | Department of Pathology, Brigham and Women's Hospital, Harvard Medical School | 哈佛医学院,布莱根妇女医院,病理学系 |
研究背景
- 背景介绍: 这篇文章的研究背景是定量评估组织图像对于计算病理学(CPath)任务至关重要,需要从全切片图像(WSIs)中客观表征组织形态特征。然而,WSIs的高分辨率和组织形态特征的多样性带来了大规模数据标注的挑战。
- 研究内容: 该问题的研究内容包括通过使用大规模预训练模型来解决这些挑战,特别是通过自我监督学习在大规模组织学切片集合上进行预训练。
- 文献综述: 该问题的相关工作包括使用自然图像数据集进行迁移学习和自我监督学习的方法,但这些方法在多样化的组织类型上的应用和评估仍有限。
研究方法
这篇论文提出了一个名为UNI的通用自我监督模型。具体来说:
- 模型架构: UNI基于视觉变换器(ViT)架构,使用了DINOv2自我监督学习方法进行预训练。
- 数据集: 使用了Mass-100K数据集,包含来自20个主要组织类型的超过1亿个组织补丁,总计约77TB的数据。
- 预训练方法: 使用DINOv2方法进行自我监督学习,包括自蒸馏损失和掩码图像建模损失。
- 评估方法: 在34个代表性的CPath任务上评估UNI的性能,包括ROI级分类、分割和图像检索等。
实验设计
- 数据收集: Mass-100K数据集由来自MGH和BWH的内部组织学切片以及GTEx联盟的组织学切片组成。
- 样本选择: 数据集中的每个WSI被采样约800个组织补丁,用于预训练和微调。
- 参数配置: 使用4x8 80GB NVIDIA A100 GPU 节点进行多GPU、多节点训练,所有下游实验在单个24GB NVIDIA 3090 GPU上进行。
结果与分析
- 预训练扩展性: 在不同数据规模上的预训练结果显示,随着数据量的增加,UNI在OT-43和OT-108任务上的性能显著提高。
- 模型性能: UNI在34个CPath任务中的表现优于现有的最先进模型,特别是在分辨率无关的组织分类、少样本类原型和疾病亚型分类方面表现出色。
- 弱监督学习: 在弱监督滑动分类任务中,UNI在多个任务中表现出色,特别是在罕见癌症类型分类和高诊断复杂性任务中。
- 高分辨率图像处理: UNI在不同分辨率下的表现稳健,能够编码与大多数图像分辨率无关的语义有意义的表示。
一、UNI框架
1-1:数据集分布
Mass-100K是一个大规模且多样化的预训练数据集,包含10亿个组织病理学补丁,这些补丁来自超过100,000张诊断性全玻片图像(WSIs),涵盖了20个主要器官类型。
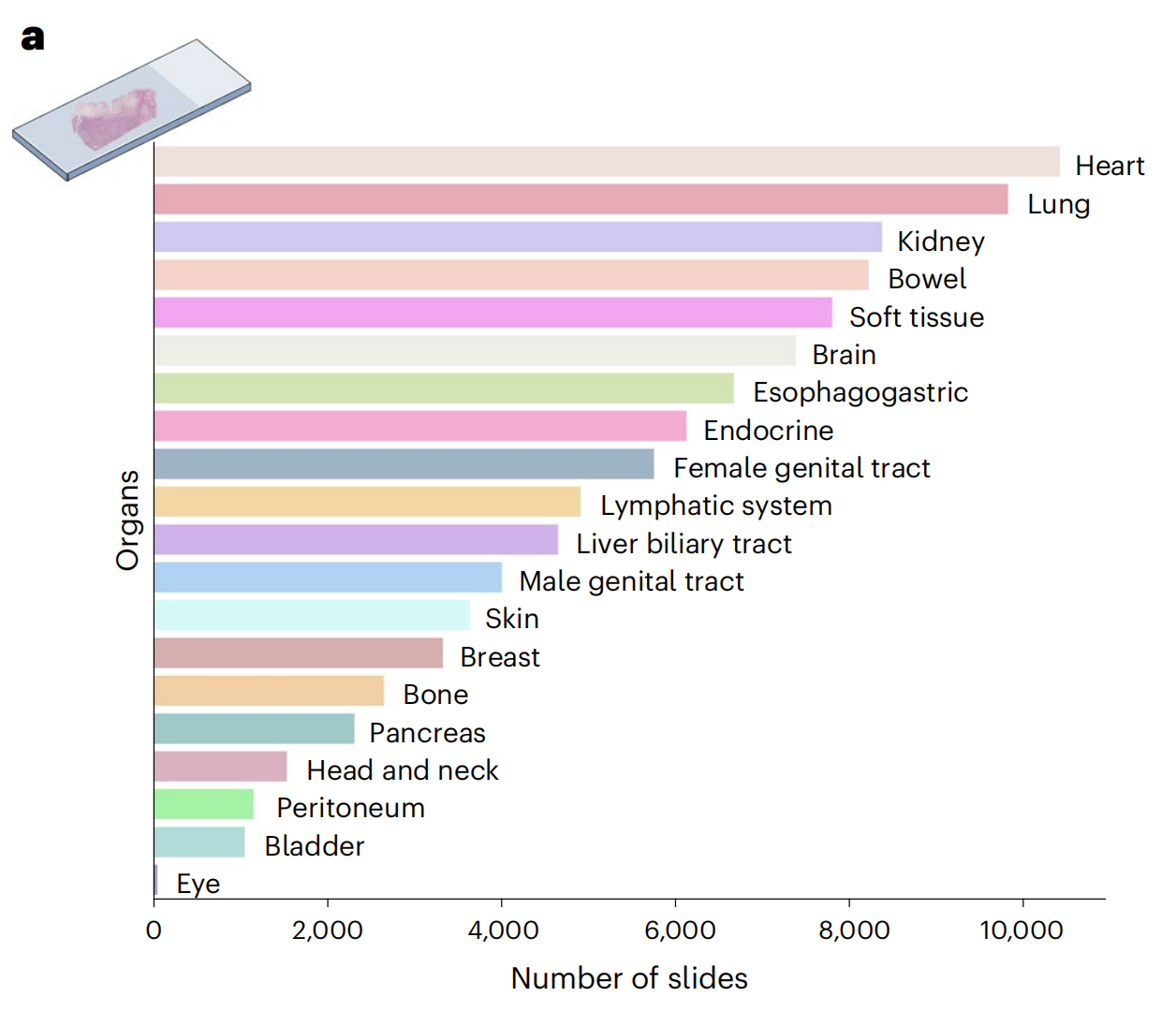
简单列表分析一下
| 器官/系统 | 切片数量描述 | 研究重要性描述 |
|---|---|---|
| 心脏 (Heart) | 接近10000片 | 心脏在研究中的重要性 |
| 肺 (Lung) | 略低于心脏,切片数量也很高 | 肺部研究的广泛性 |
| 肾脏 (Kidney) | 切片数量较多 | 肾脏在生理和病理研究中的重要性 |
| 肠 (Bowel) | 切片数量较多 | 消化系统在健康和疾病研究中的重要性 |
| 软组织 (Soft tissue) | 切片数量较多 | 可能包括多种不同类型的软组织样本 |
| 大脑 (Brain) | 切片数量较多 | 神经系统研究的重要性 |
| 食道胃 (Esophagogastric) | 切片数量较多 | 可能涉及食道和胃部的疾病研究 |
| 内分泌系统 (Endocrine) | 切片数量较多 | 可能包括甲状腺、胰腺等内分泌器官 |
| 女性生殖系统 (Female genital tract) | 切片数量较多 | 可能涉及妇科相关研究 |
| 淋巴系统 (Lymphatic system) | 切片数量较多 | 可能涉及免疫学研究 |
| 肝胆系统 (Liver biliary tract) | 切片数量较多 | 可能涉及肝脏和胆道的研究 |
| 男性生殖系统 (Male genital tract) | 切片数量较多 | 可能涉及男科相关研究 |
| 皮肤 (Skin) | 切片数量较多 | 可能涉及皮肤病学研究 |
| 乳腺 (Breast) | 切片数量较多 | 可能涉及乳腺癌等研究 |
| 骨骼 (Bone) | 切片数量较多 | 可能涉及骨科和骨病研究 |
| 胰腺 (Pancreas) | 切片数量较多 | 可能涉及糖尿病等研究 |
| 头颈部 (Head and neck) | 切片数量较多 | 可能涉及头颈癌等研究 |
| 腹膜 (Peritoneum) | 切片数量较少 | 可能涉及腹膜相关疾病研究 |
| 膀胱 (Bladder) | 切片数量较少 | 可能涉及泌尿系统疾病研究 |
| 眼睛 (Eye) | 切片数量最少 | 可能涉及眼科疾病研究 |
1-2:预训练方法
UNI使用DINOv2自监督训练算法在Mass-100K上进行预训练。DINOv2结合了掩膜图像建模目标(mask image modeling objective)和自蒸馏目标(self-distillation objective)。
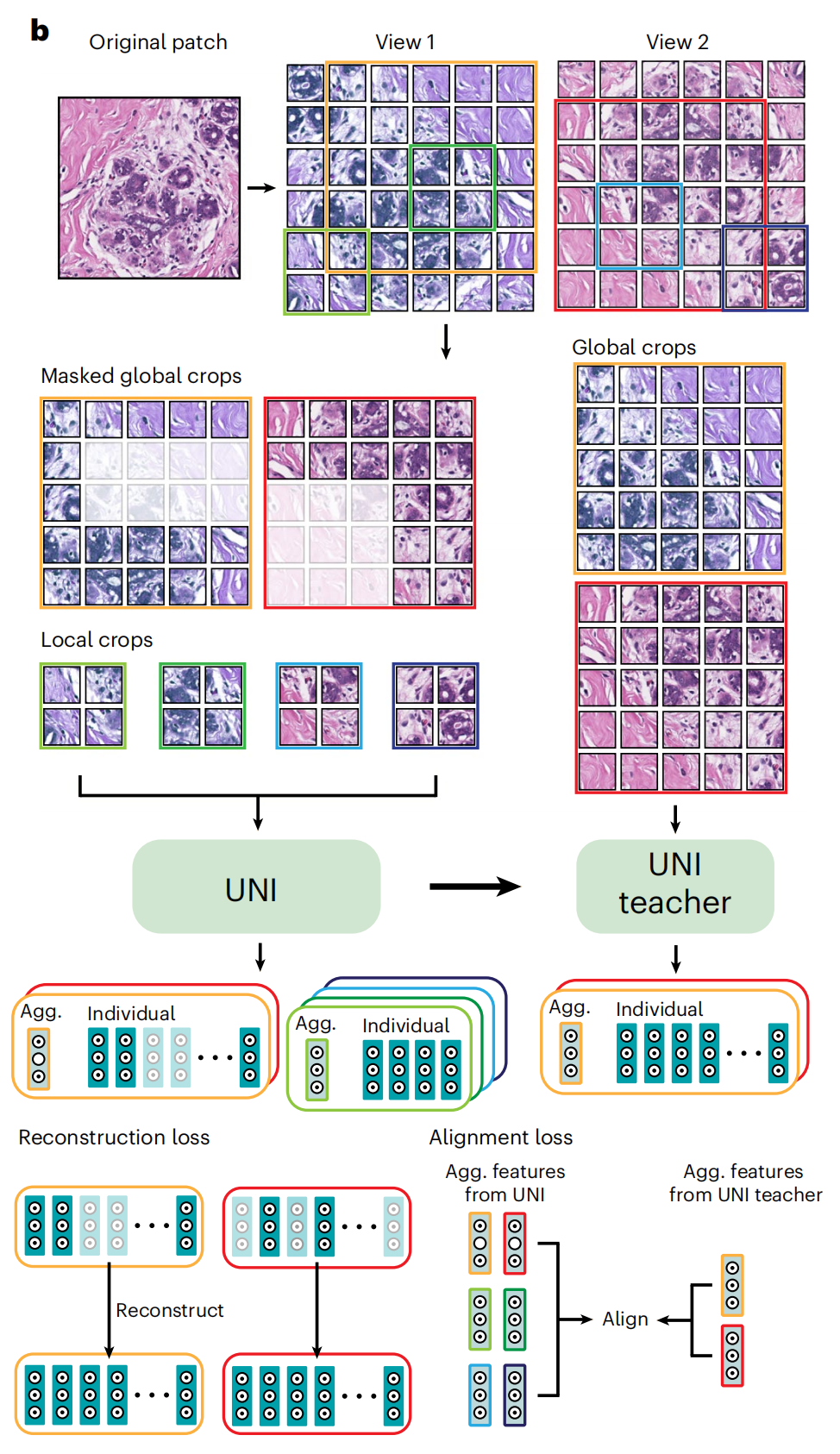
以下是对图中各部分的详细分析:
-
Original Patch: 图像的最上方显示了一个原始的组织切片图像。
-
View 1 和 View 2: 原始图像被分割成多个小块(patches),这些小块被用来生成两个不同的视图。
-
Masked Global Crops: 从两个视图中,一些全局的图像块被遮盖(masked),这意味着这些区域在训练过程中不会被模型直接看到。这通常用于训练模型去预测或重建这些遮盖的部分。
-
Global Crops: 未被遮盖的全局图像块,这些将被用于训练模型。
-
Local Crops: 其实这就是被遮盖的那一部分。
-
UNI 和 UNI Teacher: 这两个模块代表了模型的两个不同部分。UNI 是一个学生模型,而 UNI Teacher 是一个教师模型,用于指导学生模型的学习过程。
-
Aggregation (Agg.) 和 Individual: 在 UNI 模块中,聚合特征和个体特征被用来计算重建损失(Reconstruction loss)。这表明模型需要重建遮盖的图像块。
-
Alignment Loss: 在 UNI Teacher 模块中,对齐损失(Alignment loss)被用来确保学生模型的特征与教师模型的特征对齐。这有助于提高学生模型的性能。
-
Reconstruct: 在 UNI 模块的底部,重建过程将聚合特征和个体特征结合起来,尝试重建原始的遮盖图像块。
-
Align: 在 UNI Teacher 模块的底部,对齐过程确保学生模型的输出与教师模型的输出保持一致。
整体来看,这个框架通过遮盖某些图像区域并训练模型去重建这些区域,从而学习到图像的内在特征。同时,通过教师-学生模型的结构,可以提高学习效率和模型性能。这种自监督学习方法在没有大量标注数据的情况下尤其有用。
1-3:性能比较
在34个临床任务中,UNI的表现优于其他预训练编码器。这些任务涵盖了解剖病理学的多个方面。

任务分类
图中的任务被分为四类,用不同颜色表示:
- ROI分类(蓝色)
- ROI检索(绿色)
- ROI分割(黄色)
- 切片分类(紫色)
模型比较
图中比较了四个模型:
- UNI(红色)
- REMEDIS(青色)
- CTransPath(灰色)
- ResNet-50 (t)(黄色)
具体任务
| 任务名称 | 缩写 | 描述 |
|---|---|---|
| 前列腺腺癌组织分类 | AGGC | PRAD tissue class. |
| 食管癌 | ESCA | - |
| 结直肠癌 | CRC | - |
| 结直肠癌组织分类 | HunCRC | CRC tissue class. |
| 肿瘤浸润淋巴细胞检测 | TCGA | TIL det. |
| 结直肠癌微卫星不稳定性筛查 | TCGA | CRC MSI screening |
| 乳腺癌亚型分类 | BACH | BRCA subtyping |
| 肾细胞癌组织分类 | TCGA+ | RCC tissue class. |
| 乳腺癌转移检测 | CAMELYON16 | Breast metastasis det. |
| 非小细胞肺癌亚型分类 | TCGA+ | NSCLC subtyping |
| 肾细胞癌亚型分类 | DHMC | RCC subtyping |
| 结直肠癌筛查 | HunCRC | CRC screening |
| 乳腺癌C亚型分类 | BRACS | BRCA C-subtyping |
| 乳腺癌F亚型分类 | BRACS | BRCA F-subtyping |
| 前列腺ISUP分级 | PANDA | Prostate ISUP grading |
| 胶质瘤IDH1筛查 | TCGA+ | Glioma IDH1 screening |
| 胶质瘤组织分子亚型分类 | TCGA+ | Glioma histo-molecular subtyping |
| 脑肿瘤C亚型分类 | EBRAINS | Brain tumor C-subtyping |
| OncoTree分类 | OT108 | OncoTree class. |
| 泛癌分类 | OP43 | Pan-cancer class. |
| 心脏移植评估 | BWH-EMB | Heart transplant assess. |
这个表格总结了不同癌症类型和相关病理分析任务的名称、缩写以及描述。
1-4:评估任务
这张图展示了一个病理图像分析的流程,分为三个主要部分:Patch-level classification(块级分类)、Retrieval and prototyping(检索和原型化)、Slide-level classification(切片级分类)。

以下是对每个部分的详细分析:
Patch-level classification(块级分类)
- 输入:原始组织切片图像。
- UNI模型:图像首先通过UNI模型处理。
- 分类器:处理后的图像被送入分类器,进行块级分类,输出为BRCA(乳腺癌)。
Patch-level segmentation(块级分割)
- UNI模型:同样使用UNI模型处理图像。
- 分割网络:处理后的图像被送入分割网络,进行块级分割,输出分割后的图像。
Retrieval and prototyping(检索和原型化)
- 输入:多个组织切片图像。
- UNI模型:每个图像都通过UNI模型处理。
- 检索:处理后的图像特征被用来进行检索,找到与查询图像最相似的原型(prototyping)。
- 查询:查询图像通过UNI模型处理后,与检索到的原型进行比较。
Slide-level classification(切片级分类)
- 输入:多个组织切片图像。
- UNI模型:每个图像都通过UNI模型处理。
- 聚合器:处理后的图像特征被聚合。
- 分类器:聚合后的特征被送入分类器,进行切片级分类,输出包括BRCA、IDC、ILC、GBMLGG、GBM、
二、UNI在切片级别任务中的性能
2-1:器官和OncoTree代码分布
这张图是一个环形条形图,展示了不同癌症类型在OncTree分类系统中的分布情况。
图中使用了两种分类系统:OT-43和OT-108,分别代表43种癌症类型和108种OncTree代码,其中OncoTree代码是一种癌症分类系统,用于精确地描述肿瘤的类型。
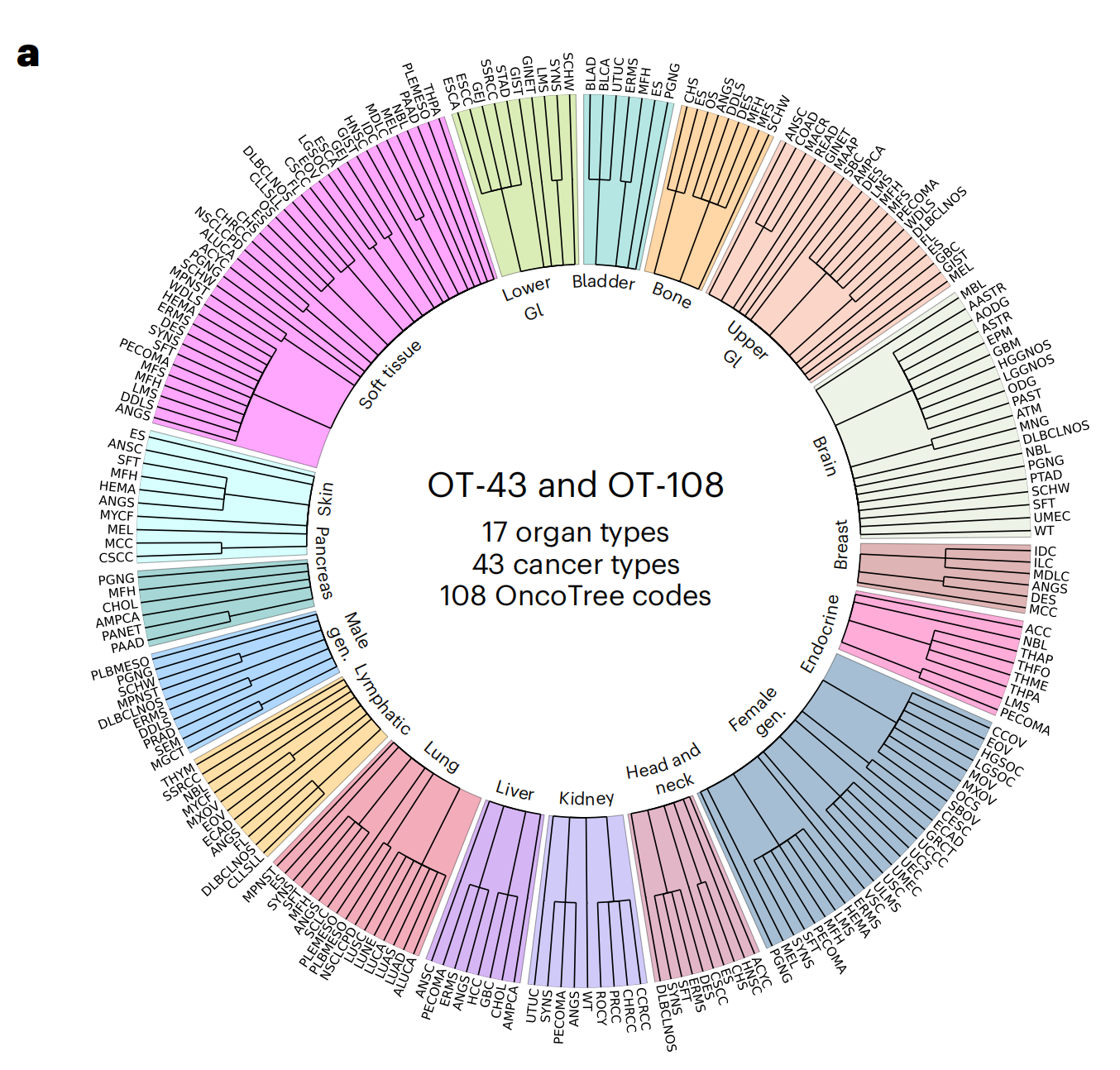
以下是对图表的详细分析:
-
癌症类型分类:
- 图中将癌症分为43种主要类型,每种类型用不同的颜色表示。
- 这些癌症类型进一步细分为108个OncTree代码,每个代码代表一种特定的癌症亚型。
-
器官类型:
- 图中还标注了17种器官类型,包括胰腺、肝脏、肾脏、头颈部、乳腺、内分泌系统等。
- 每种器官类型下展示了该器官可能发生的癌症类型。
-
癌症分布:
- 环形图的外圈显示了不同癌症类型的分布,每个条形的长度代表了该癌症类型的相对数量或比例。
- 内圈则展示了这些癌症类型在不同器官中的分布情况。
-
特定癌症类型:
- 例如,乳腺癌(Breast)在图中被详细分类,包括多种亚型,如ER+、HER2+等。
- 同样,结直肠癌(CRC)也被细分为多种亚型,如MSI、MSS等。
-
数据解读:
- 通过观察条形的长度,可以比较不同癌症类型的相对频率或重要性。
- 例如,如果某个癌症类型的条形较长,这可能表示该癌症类型在研究或临床中较为常见或重要。
-
研究应用:
- 这种分类系统有助于研究人员和临床医生更好地理解和分类癌症,从而进行更精确的诊断和治疗。
- 通过这种详细的分类,可以针对特定癌症亚型开发个性化的治疗方案。
总的来说,这张图表提供了一个全面的视觉化工具,用于展示和比较不同癌症类型及其在不同器官中的分布情况,这对于癌症研究和治疗具有重要意义。
2-2:宏平均AUROC比较
图b和d分别展示了UNI与其他预训练编码器在OT-43和OT-108任务上的宏平均AUROC(Area Under the Receiver Operating Characteristic curve)比较。

这两张图(b和d)展示了四种不同模型在某个分类任务上的性能比较,使用的是接收者操作特征曲线(Receiver Operating Characteristic curve,简称ROC曲线)。ROC曲线是一种用于评估二分类模型性能的工具,它通过绘制灵敏度(Sensitivity,也称为真正率)与1-特异性(1-Specificity,也称为假正率)之间的关系来展示模型的性能。
图b分析:
- UNI 模型的曲线最接近左上角,表明其在灵敏度和特异性之间取得了最佳平衡,其AUC(曲线下面积)为0.976,范围在0.971到0.981之间。
- REMEDIS 模型的AUC为0.954,范围在0.946到0.961之间,性能也相对较好,但略逊于UNI模型。
- CTransPath 模型的AUC为0.956,范围在0.949到0.962之间,与REMEDIS模型性能相近。
- ResNet-50 (IN) 模型的AUC为0.862,范围在0.850到0.873之间,性能明显低于其他三个模型。
图d分析:
- UNI 模型的AUC为0.972,范围在0.968到0.976之间,依然表现最佳。
- REMEDIS 模型的AUC为0.952,范围在0.946到0.956之间,性能略低于UNI模型。
- CTransPath 模型的AUC为0.959,范围在0.955到0.963之间,性能与UNI模型相近,但略低。
- ResNet-50 (IN) 模型的AUC为0.869,范围在0.860到0.877之间,性能依然最低。
总结:
- UNI 模型在两个测试中都表现出了最高的AUC值,说明其在这项任务上具有最好的分类性能。
- REMEDIS 和 CTransPath 模型的性能相近,但都略低于UNI模型。
- ResNet-50 (IN) 模型的性能明显低于其他三个模型,可能需要进一步的优化或调整。
AUC值越接近1,模型的性能越好。这些结果可以帮助研究人员选择最适合他们任务的模型。
2-3:不同预训练数据规模的Top-1准确度
针对OT-43和OT-108任务,图c和e展示了UNI在不同预训练数据规模(Mass-1K, Mass-22K, Mass-100K)下的Top-1准确度。

这两张图(c和e)展示了不同模型在不同预训练数据规模下的Top-1准确率。Top-1准确率是指模型预测的最可能类别与实际类别相匹配的比例。
图c分析:
- UNI 模型在预训练数据规模从0到100百万张图像的范围内,Top-1准确率表现出较高的水平,且随着数据规模的增加,准确率有所提升。
- REMEDIS 模型的Top-1准确率也随着数据规模的增加而提高,但整体表现略低于UNI模型。
- CTransPath 模型的准确率在小规模数据时较高,但随着数据规模的增加,准确率提升不明显。
图e分析:
- UNI 模型在图e中的表现与图c相似,Top-1准确率随着数据规模的增加而提高,且在所有模型中表现最佳。
- REMEDIS 模型在图e中的表现也与图c相似,准确率随着数据规模的增加而提高,但仍然低于UNI模型。
- CTransPath 模型在图e中的表现与图c略有不同,准确率在小规模数据时较高,但随着数据规模的增加,准确率提升有限。
这些结果表明,模型的性能与预训练数据的规模有显著关系,且不同的模型在处理不同规模的数据时表现出不同的性能特点。
2-4:弱监督切片级分类任务的监督性能
UNI与其他模型在15个弱监督切片级分类任务上的表现。
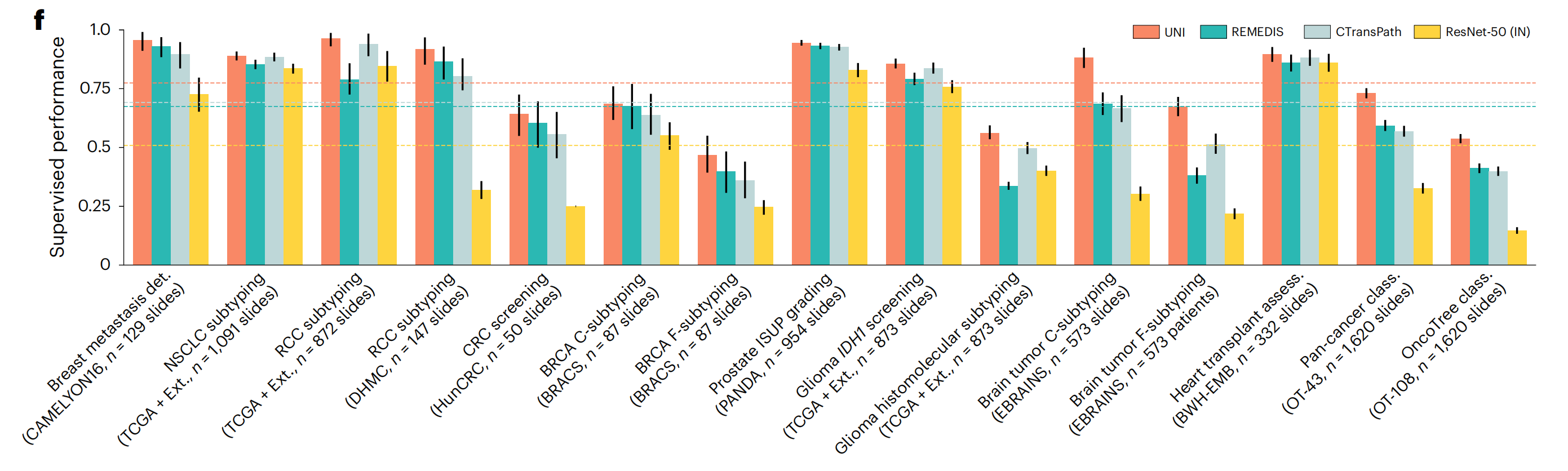
这张图是一个条形图,展示了不同模型在多个病理图像分析任务上的监督性能。每个任务的性能通过条形的高度来表示,条形上的误差线表示性能的不确定性或变异性。
模型比较
图中比较了四个模型:
- UNI(红色)
- REMEDIS(青色)
- CTransPath(灰色)
- ResNet-50 (IN)(黄色)
任务描述
| 任务名称 | 数据集缩写 | 样本数量 | 描述 |
|---|---|---|---|
| 乳腺癌转移检测 | CAMELYON16 | 129 | 乳腺癌转移检测,129张切片。 |
| 非小细胞肺癌亚型分类 | TCGA + Ext. | 1091 | 非小细胞肺癌亚型分类,1091张切片。 |
| 肾细胞癌亚型分类 | DHMC | 147 | 肾细胞癌亚型分类,147张切片。 |
| 结直肠癌筛查 | HunCRC | 50 | 结直肠癌筛查,50张切片。 |
| 乳腺癌F亚型分类 | BRACS | 87 | 乳腺癌F亚型分类,87张切片。 |
| 前列腺ISUP分级 | PANDA | 954 | 前列腺ISUP分级,954张切片。 |
| 胶质瘤IDH1筛查 | TCGA + Ext. | 873 | 胶质瘤IDH1筛查,873张切片。 |
| 胶质瘤组织分子亚型分类 | TCGA + Ext. | 873 | 胶质瘤组织分子亚型分类,873张切片。 |
| 脑肿瘤C亚型分类 | EBRAINS | 573 | 脑肿瘤C亚型分类,573张切片。 |
| 心脏移植评估 | BWH-EMB | 573 | 心脏移植评估,573名患者。 |
| 泛癌分类 | OT-43 | 1620 | 泛癌分类,1620张切片。 |
| OncTree分类 | OT-108 | 1620 | OncTree分类,1620张切片。 |
这个表格总结了不同癌症类型和相关病理分析任务的名称、数据集缩写以及样本数量。
2-5:少样本切片级性能
这组图(g, h, i, j)展示了不同模型在少样本学习(Few-shot learning)任务中的性能,随着每个类别的训练标签数量的增加,模型的性能如何变化。
这些任务包括肾细胞癌亚型分类(RCC subtyping)、乳腺癌细粒度亚型分类(BRCA fine-grained subtyping)、脑肿瘤粗粒度亚型分类(Brain tumor coarse-grained subtyping)和ISUP分级(ISUP grading)。
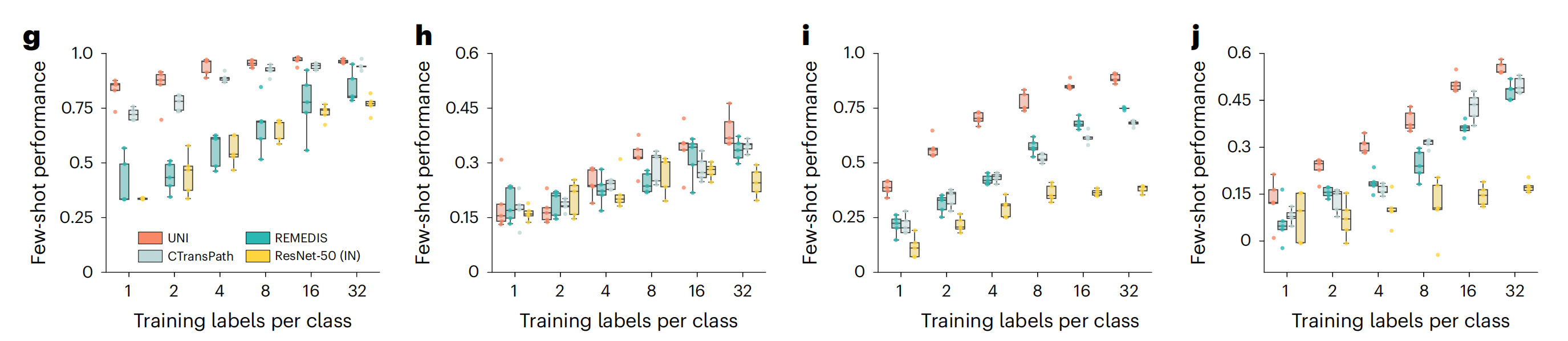
每个图中的箱线图代表了不同模型在不同训练标签数量下的性能分布。以下是对每个图的详细分析:
图g
- UNI 模型在所有训练标签数量下都表现出较高的性能,尤其是在标签数量较多时。
- REMEDIS 模型的性能次之,随着训练标签的增加,性能有所提升。
- CTransPath 和 ResNet-50 (IN) 模型的性能相对较低,且随着训练标签的增加,性能提升不明显。
图h
- UNI 模型依然表现最好,尤其是在训练标签数量增加时。
- REMEDIS 模型的性能有所提升,但仍然低于UNI模型。
- CTransPath 和 ResNet-50 (IN) 模型的性能提升有限。
图i
- 在这个图中,UNI 模型在训练标签数量较少时就已经表现出较高的性能,随着标签数量的增加,性能进一步提升。
- REMEDIS 模型的性能随着训练标签的增加而提升,但仍然低于UNI模型。
- CTransPath 和 ResNet-50 (IN) 模型的性能在训练标签数量较少时较低,随着标签数量的增加,性能有所提升,但仍然低于前两个模型。
图j
- UNI 模型在所有训练标签数量下都表现出较高的性能。
- REMEDIS 模型的性能随着训练标签的增加而提升,但仍然低于UNI模型。
- CTransPath 和 ResNet-50 (IN) 模型的性能在训练标签数量较少时较低,随着标签数量的增加,性能有所提升,但提升幅度有限。
三、UNI在ROI级别任务上的性能
3-1:ROI分类示例
这张图展示了病理图像分割任务中的"Ground truth"(真实标注)与模型"Prediction"(预测结果)的对比。图像分割任务的目标是让模型能够准确地识别和区分图像中的不同区域,例如肿瘤组织(Stroma)和正常组织(Benign)。

图像内容分析
-
Ground truth(真实标注):
- 左侧的图像显示了病理切片的真实标注情况。
- 不同颜色代表不同的组织类型或病变程度:
- Stroma(肿瘤基质):用青色表示。
- Benign(良性):用蓝色表示。
- G3, G4, G5:代表不同的肿瘤分级,用绿色、黄色和红色表示。
-
Prediction(预测结果):
- 中间的图像显示了模型对同一病理切片的预测结果。
- 预测结果与真实标注相似,但可能存在一些差异。
-
Prediction (1) 和 Prediction (2):
- 右侧的两个图像分别展示了模型在不同参数或不同训练阶段下的预测结果。
- 这些图像中,模型的预测结果被放大显示,以便更清楚地看到模型在细节上的表现。
3-2:多头自注意力(MHSA)热图可视化
这张图展示了多分辨率下的MHSA(Multi-Head Self-Attention)可视化结果,用于分析病理图像中的不同区域。图中包含两个原始的感兴趣区域(ROI)图像,以及在不同分辨率下的MHSA可视化结果。
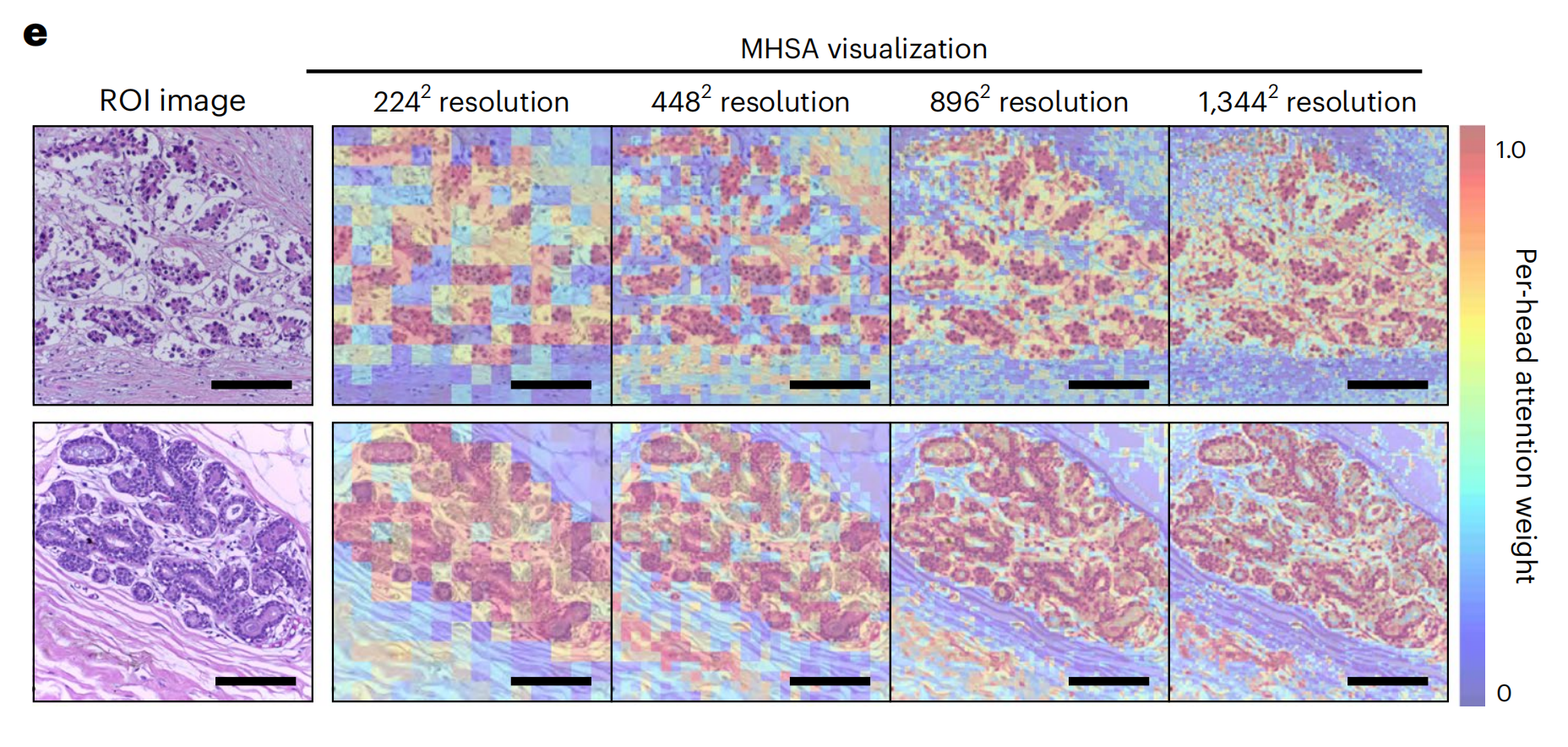
以下是对图表的详细分析:
MHSA可视化
- MHSA可视化展示了模型在不同分辨率下的注意力分布情况,分辨率从224²到1344²逐渐增加。
- 每个分辨率下的图像都显示了模型关注的区域,颜色表示注意力权重,颜色条从蓝色(低权重)到红色(高权重)。
分辨率分析
- 224² resolution:最低分辨率,显示了模型在较大区域上的注意力分布,颜色变化较为粗糙。
- 448² resolution:中等分辨率,显示了更细致的注意力分布,可以观察到模型开始关注更小的区域。
- 896² resolution:更高分辨率,模型的注意力分布更加精细,能够捕捉到更多的细节。
- 1344² resolution:最高分辨率,显示了最细致的注意力分布,模型能够关注到非常小的区域和细节。
四、UNI在少样本ROI和切片级别原型设计任务上的性能
4-1:少样本ROI分类(SimpleShot方法)
这张图展示了一个用于病理图像分析的深度学习模型的工作流程,特别是用于癌症类型的分类。图中使用了UNI模型来处理图像,并通过原型(prototype)和嵌入空间(embedding space)来进行分类预测。
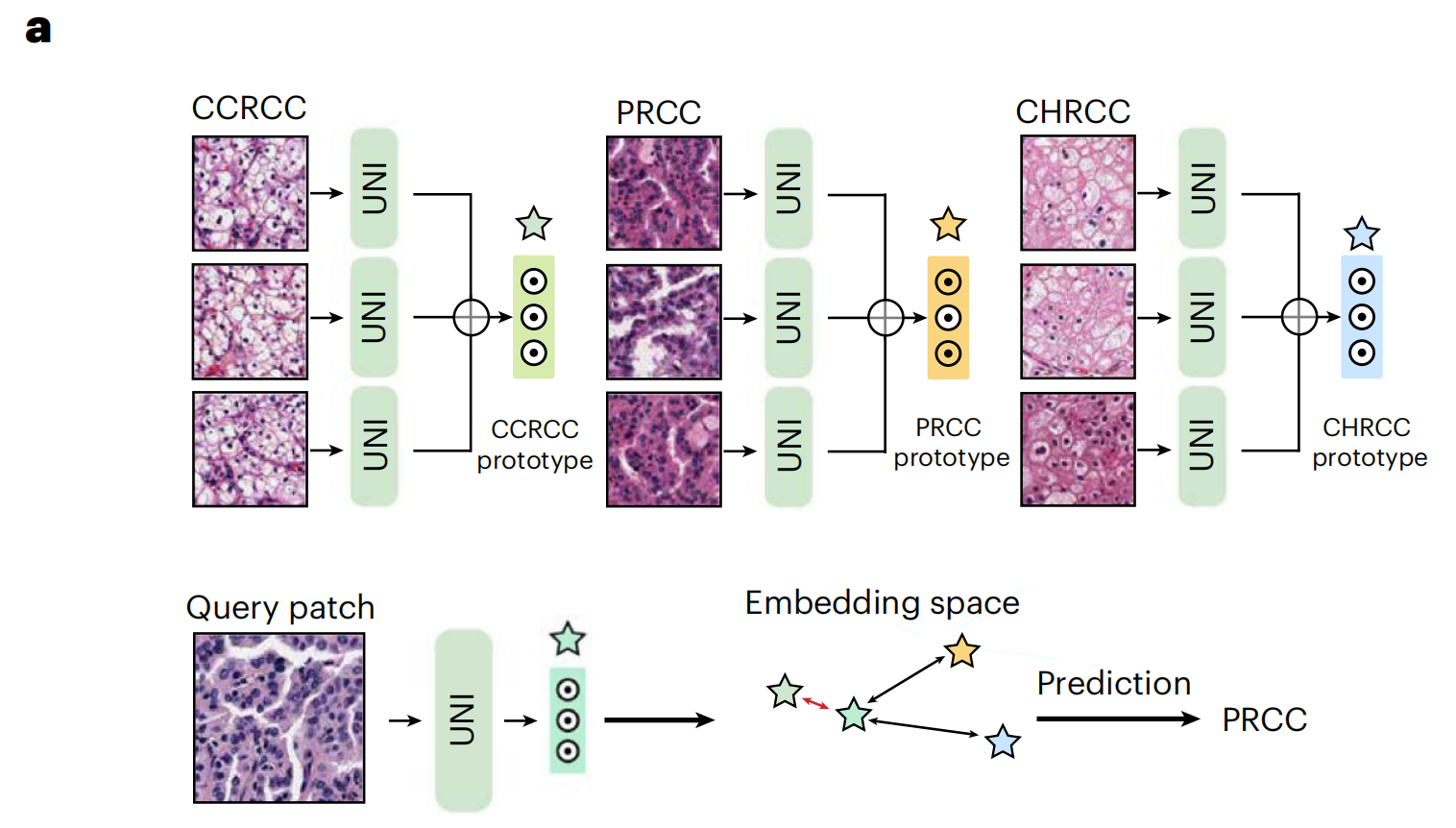
对于一个测试ROI,SimpleShot将预测标签分配给最相似的类原型(欧氏距离最小)。
图像处理流程
-
输入图像:
- 图像被分为三类:CCRCC(透明细胞肾细胞癌)、PRCC(乳头状肾细胞癌)、CHRCC(嫌色细胞肾细胞癌)。
-
UNI模型处理:
- 每种癌症类型的图像通过UNI模型进行特征提取。
- 每个图像通过UNI模型处理后,生成一个特征向量。
-
原型生成:
- 每个癌症类型的特征向量被用来生成一个原型(prototype),这个原型代表了该癌症类型的平均特征。
查询图像处理
-
查询图像:
- 查询图像(Query patch)也通过UNI模型进行特征提取。
-
嵌入空间:
- 查询图像的特征向量被放入嵌入空间(embedding space),这是一个多维空间,用于比较不同特征向量的相似性。
-
预测:
- 在嵌入空间中,查询图像的特征向量与每种癌症类型的原型进行比较。
- 根据相似度,模型预测查询图像最可能属于的癌症类型。
预测结果
- 输出 :
- 图中显示了预测结果,查询图像被预测为PRCC(乳头状肾细胞癌)。
4-2:少样本切片分类(MI-SimpleShot方法)
这张图展示了使用全切片图像(WSI)进行病理诊断的过程,特别是针对乳头状肾细胞癌(PRCC)的诊断。

相似性热图可视化了WSI中每个补丁与真实类原型之间的相似性。使用预先计算的一组ROI级别类原型(与切片具有相同的类标签),MI-SimpleShot通过查询WSI中的顶部-K个补丁与类原型的平均相似性来预测切片标签。
以下是对图表的详细分析:
WSI (PRCC)
- 左上角显示了一张全切片图像(WSI),这是一张病理切片的高分辨率图像,用于诊断。
Similarity heatmap
- 右上角的图像是一个相似性热图,它显示了WSI中不同区域与PRCC原型的相似度。颜色从蓝色(低相似度)到红色(高相似度)变化,表明哪些区域与PRCC的特征最为相似。
Top-K patches
- 下方的条形图展示了WSI中与PRCC、透明细胞肾细胞癌(CCRCC)和嫌色细胞肾细胞癌(CH RCC)原型最相似的K个区域(patches)。每个条形图代表一个区域,高度表示相似度。
- PRCC(红色):与PRCC原型最相似的区域。
- CCRCC(绿色):与CCRCC原型最相似的区域。
- CH RCC(蓝色):与CH RCC原型最相似的区域。
Average similarity
- 条形图右侧的柱状图显示了WSI中所有区域与三种癌症原型的平均相似度。这有助于整体评估WSI与每种癌症类型的相似性。
- PRCC的平均相似度最高,表明WSI最可能代表PRCC。
- CCRCC 和CH RCC的平均相似度较低。
Prediction
- 根据平均相似度,模型预测该WSI为PRCC。
4-3:少样本切片分类性能和相似性热图(MI-SimpleShot)
图f和g展示了MI-SimpleShot在非小细胞肺癌亚型分类(NSCLC,训练集TCGA,测试集CPTAC,n=1,091张切片)和肾细胞癌亚型分类(RCC,训练集TCGA,测试集CPTAC-DHMC,n=872张切片)任务上的少样本切片分类性能。
这张图展示了在少样本学习(Few-shot learning)环境下,两种不同方法(UNI的Slide prototyping和UNI的ML)在病理图像分类任务中的性能比较。
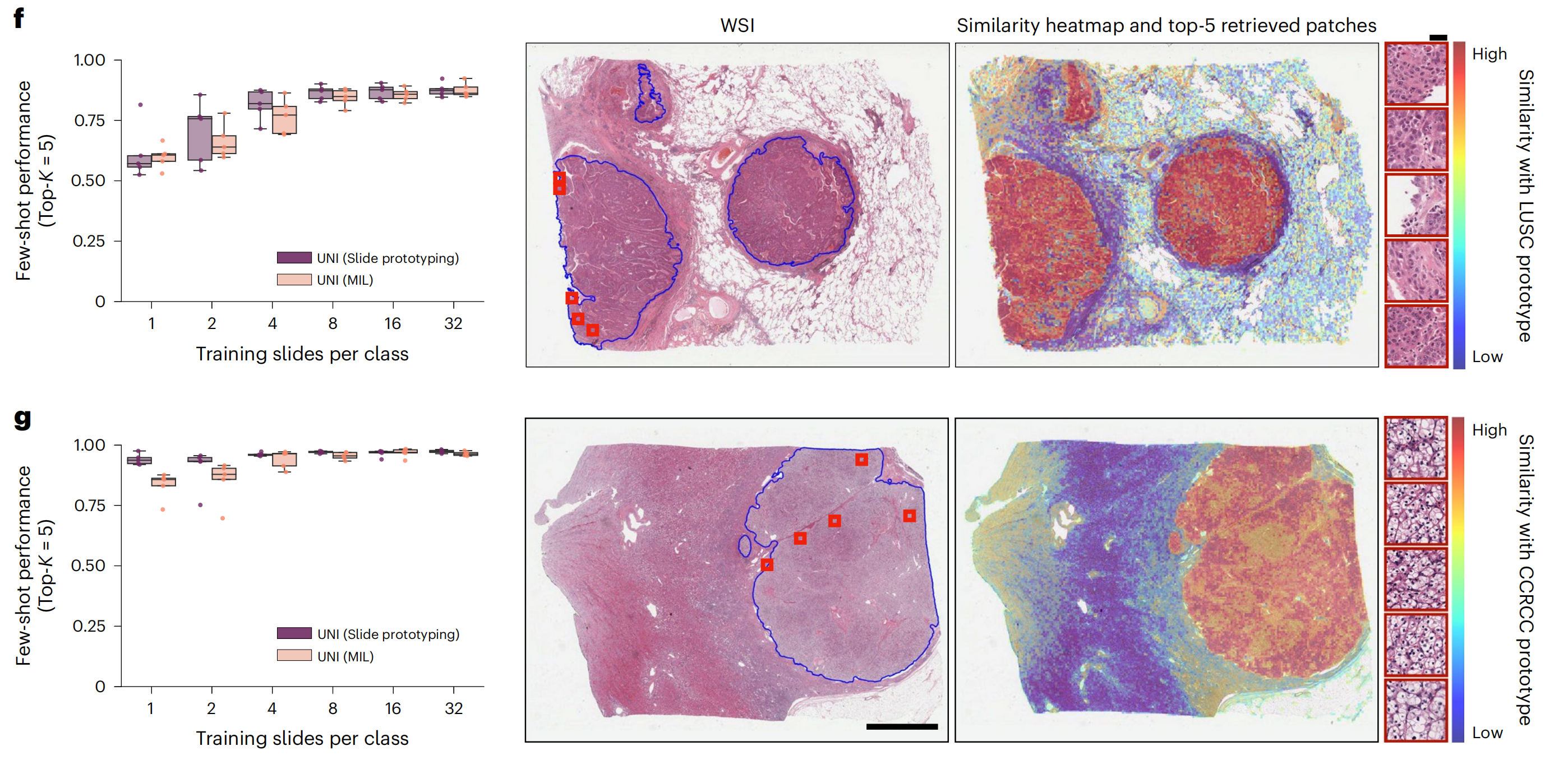
性能箱线图(f和g)
- 箱线图展示了在不同数量的训练样本(每个类别1到32张切片)下,两种方法的Top-K准确率(Top-5)。
- UNI (Slide prototyping)(紫色)在所有训练样本数量下都表现出较高的准确率,且随着训练样本的增加,性能有所提升。
- UNI (ML)(橙色)的性能相对较低,且随着训练样本的增加,性能提升不明显。
相似性热图和检索图像(f和g的右侧)
- WSI:展示了整个切片图像(Whole Slide Image)。
- Similarity heatmap:展示了模型对WSI中不同区域与查询图像的相似性评估。颜色从蓝色(低相似性)到红色(高相似性)变化。
- Top-5 retrieved patches:展示了模型检索到的与查询图像最相似的五个图像块。这些图像块被用来支持分类决策。