✨作者主页 :IT毕设梦工厂✨
个人简介:曾从事计算机专业培训教学,擅长Java、Python、PHP、.NET、Node.js、GO、微信小程序、安卓Android等项目实战。接项目定制开发、代码讲解、答辩教学、文档编写、降重等。
文章目录
一、前言
系统介绍
本系统是一个基于Hadoop+Spark大数据技术栈构建的气候驱动疾病传播可视化分析平台,采用Python/Java语言支持,后端采用Django/Spring Boot框架架构,前端使用Vue+ElementUI+Echarts技术栈实现交互式数据可视化。系统通过HDFS分布式存储管理海量气候与疾病数据,利用Spark SQL进行大规模数据处理和分析,结合Pandas、NumPy等科学计算库实现复杂的统计分析算法。平台核心功能包括地理空间分析、时间序列分析、驱动因素分析和综合风险评估四大模块,能够处理包含温度、降水量、空气质量指数、UV指数等多维气候因子与疟疾、登革热等疾病传播数据的关联分析。系统支持全球各国24年历史数据的动态可视化展示,提供热力图、趋势图、相关性矩阵、雷达图等多种图表形式,帮助研究人员识别气候变化对疾病传播的影响模式,为公共卫生决策提供数据支撑和预警预测功能。
选题背景
随着全球气候变化日益加剧,极端天气事件频发,气候因素对传染病传播模式的影响已成为公共卫生领域的重要研究课题。气温升高、降水模式改变、湿度变化等气候要素直接影响病媒生物的生存繁殖环境,进而改变疟疾、登革热等媒介传播疾病的时空分布特征。传统的疾病监测和预警系统往往缺乏对气候驱动因素的深度整合分析,难以准确预测疾病传播趋势和识别高风险区域。当前疫情防控实践中,各国卫生部门迫切需要一个能够综合分析气候数据与疾病传播数据的智能化平台,以便及时发现异常传播模式,制定针对性的防控策略。然而现有的分析工具大多功能单一,缺乏跨地域、长时间序列的综合分析能力,无法满足大规模数据处理和实时可视化的需求,这为构建基于大数据技术的气候驱动疾病传播分析系统提供了现实背景。
选题意义
本系统的构建具有重要的理论价值和实际应用意义。从技术角度来看,系统将大数据处理技术与流行病学分析相结合,为跨学科研究提供了新的技术路径,有助于推动公共卫生信息化和智能化发展。从实际应用层面考虑,系统能够为疾病预防控制部门提供科学的决策支持工具,通过可视化分析帮助相关人员更直观地理解气候与疾病传播的关联规律,提高疫情监测和预警的准确性。系统的多维度分析功能可以辅助研究人员识别疾病传播的关键影响因子,为制定更有效的防控措施提供数据依据。同时,平台的地理空间分析能力有助于优化资源配置,指导高风险地区的重点防控工作。虽然作为毕业设计项目,系统在规模和复杂度上存在一定局限性,但其设计思路和技术实现方案为后续相关系统的开发提供了有益参考,对于推动公共卫生大数据应用具有一定的借鉴意义。
二、开发环境
- 大数据框架:Hadoop+Spark(本次没用Hive,支持定制)
- 开发语言:Python+Java(两个版本都支持)
- 后端框架:Django+Spring Boot(Spring+SpringMVC+Mybatis)(两个版本都支持)
- 前端:Vue+ElementUI+Echarts+HTML+CSS+JavaScript+jQuery
- 详细技术点:Hadoop、HDFS、Spark、Spark SQL、Pandas、NumPy
- 数据库:MySQL
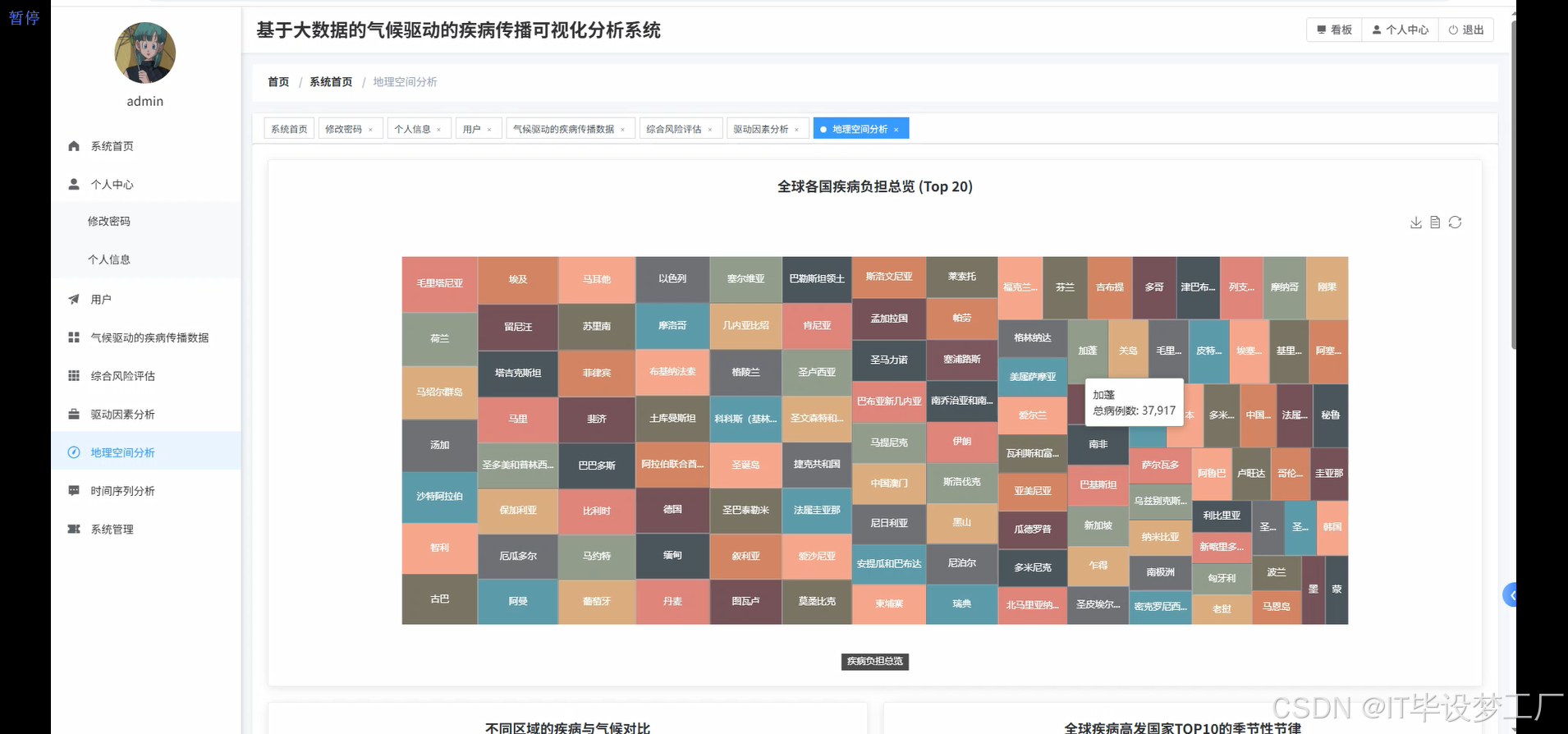
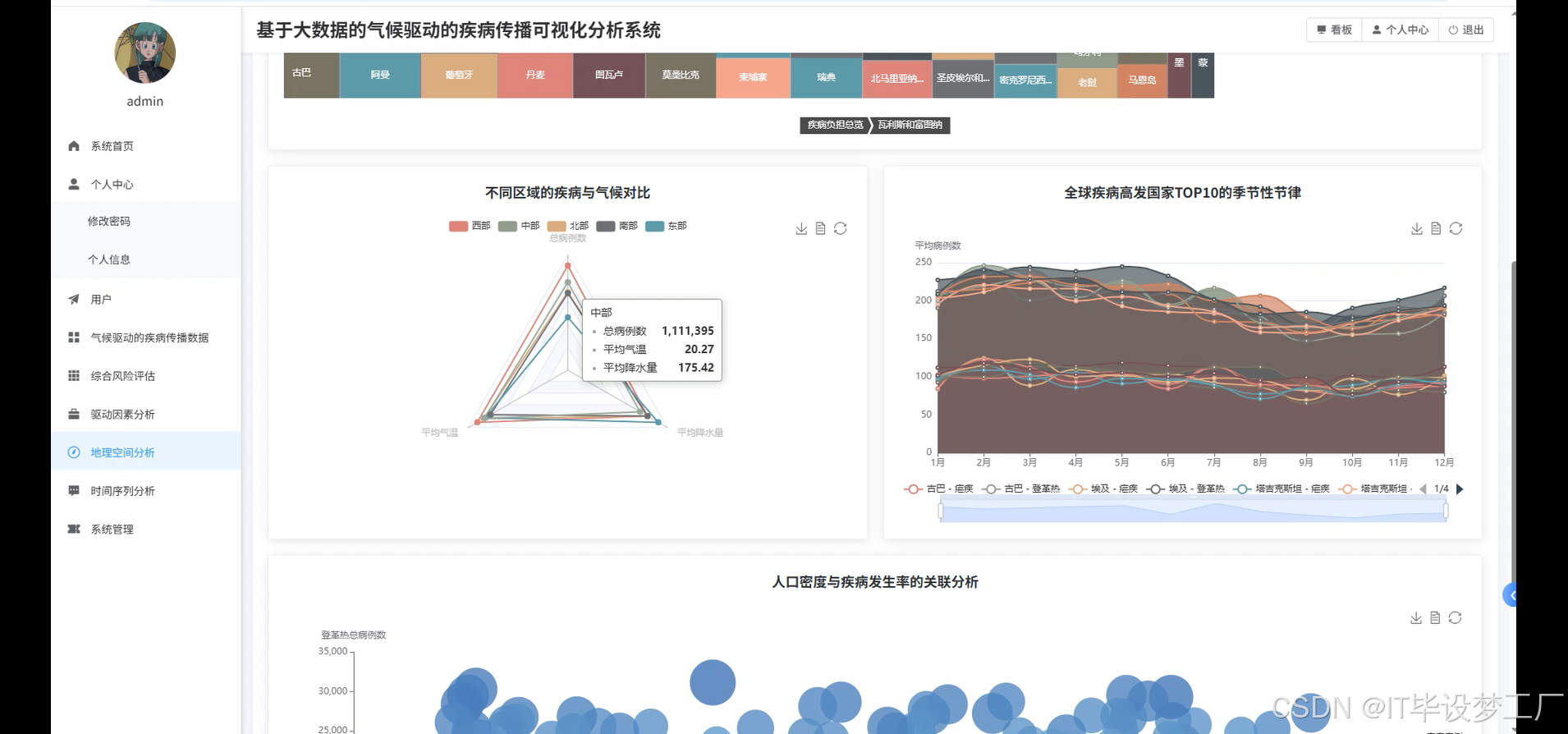
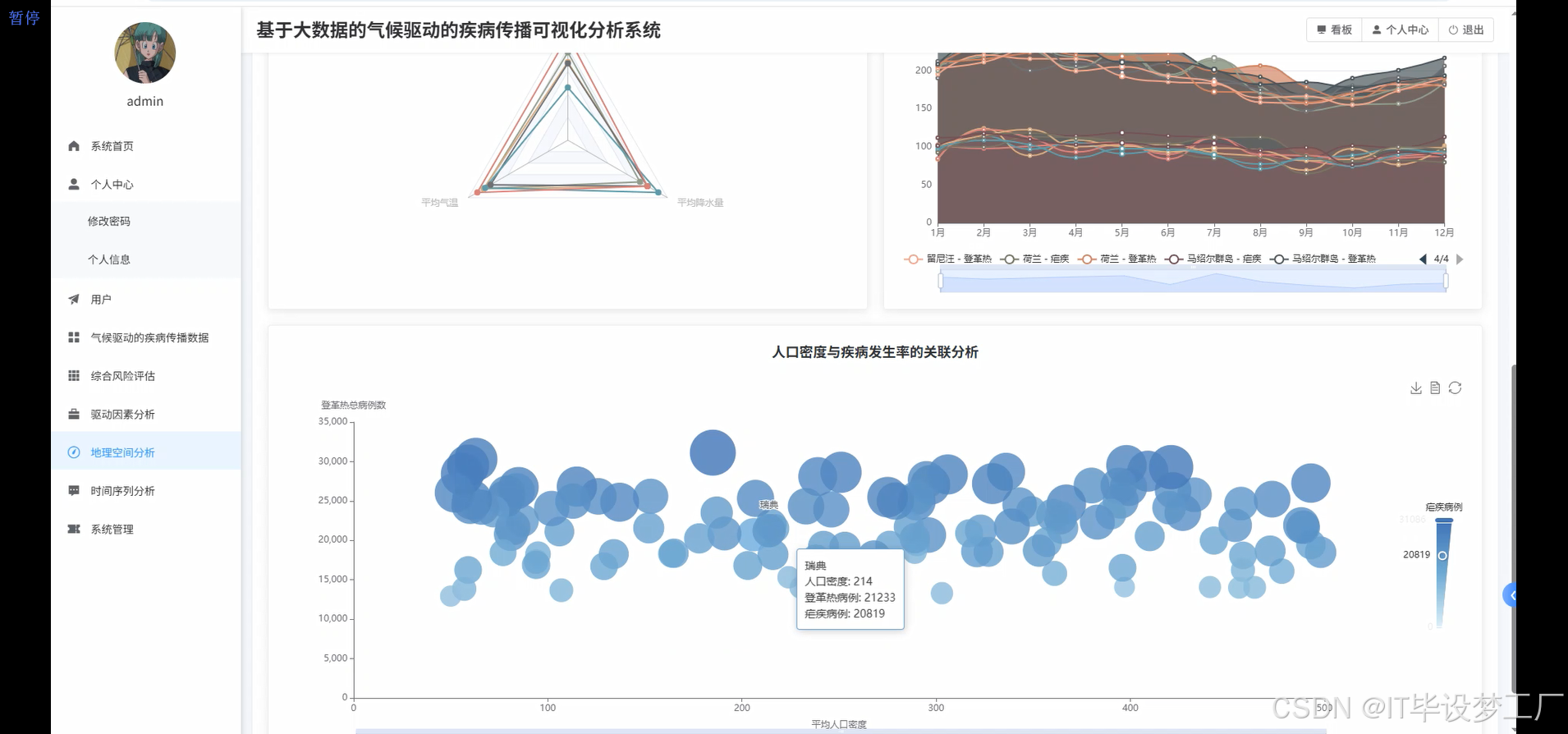
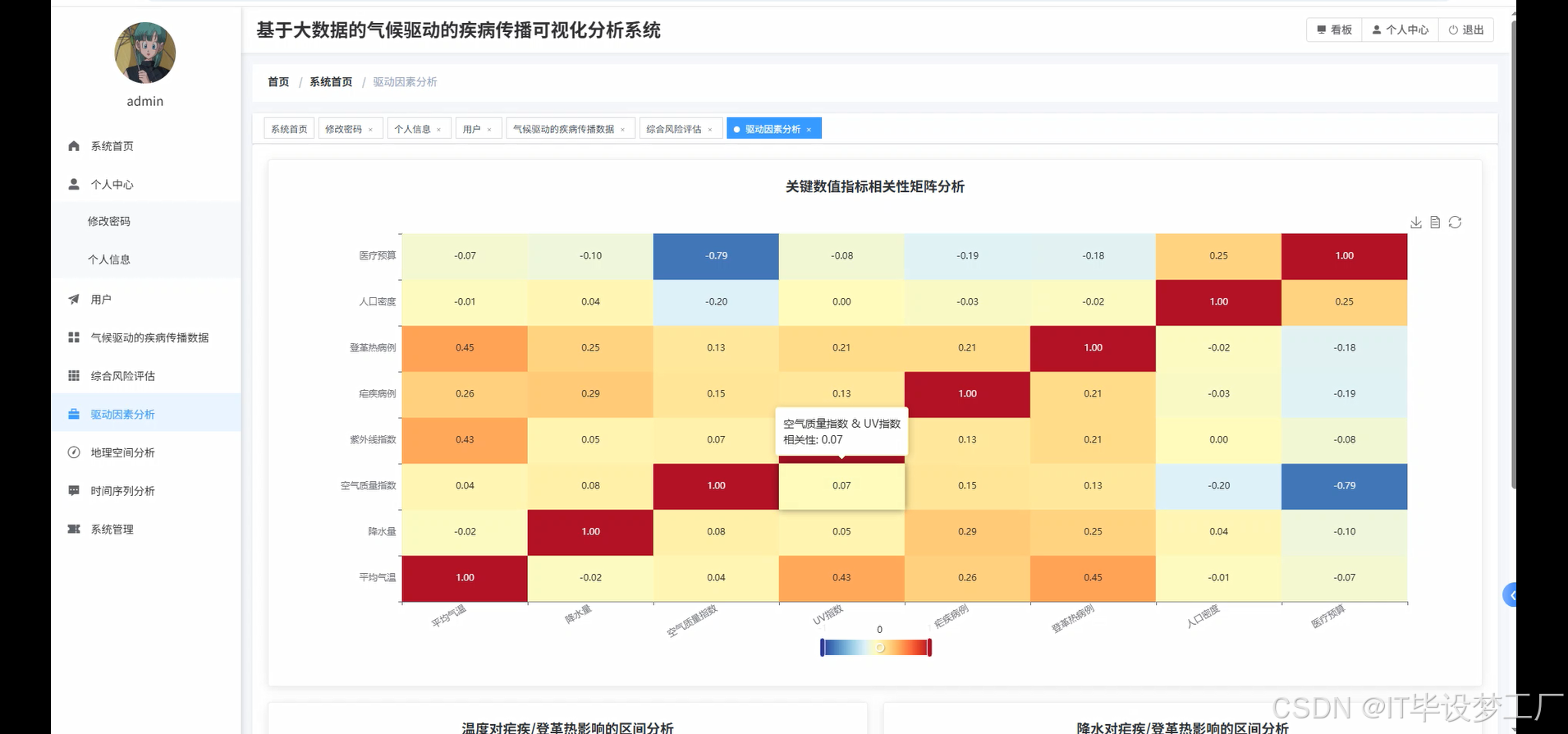
三、系统界面展示
- 基于大数据的气候驱动的疾病传播可视化分析系统界面展示:
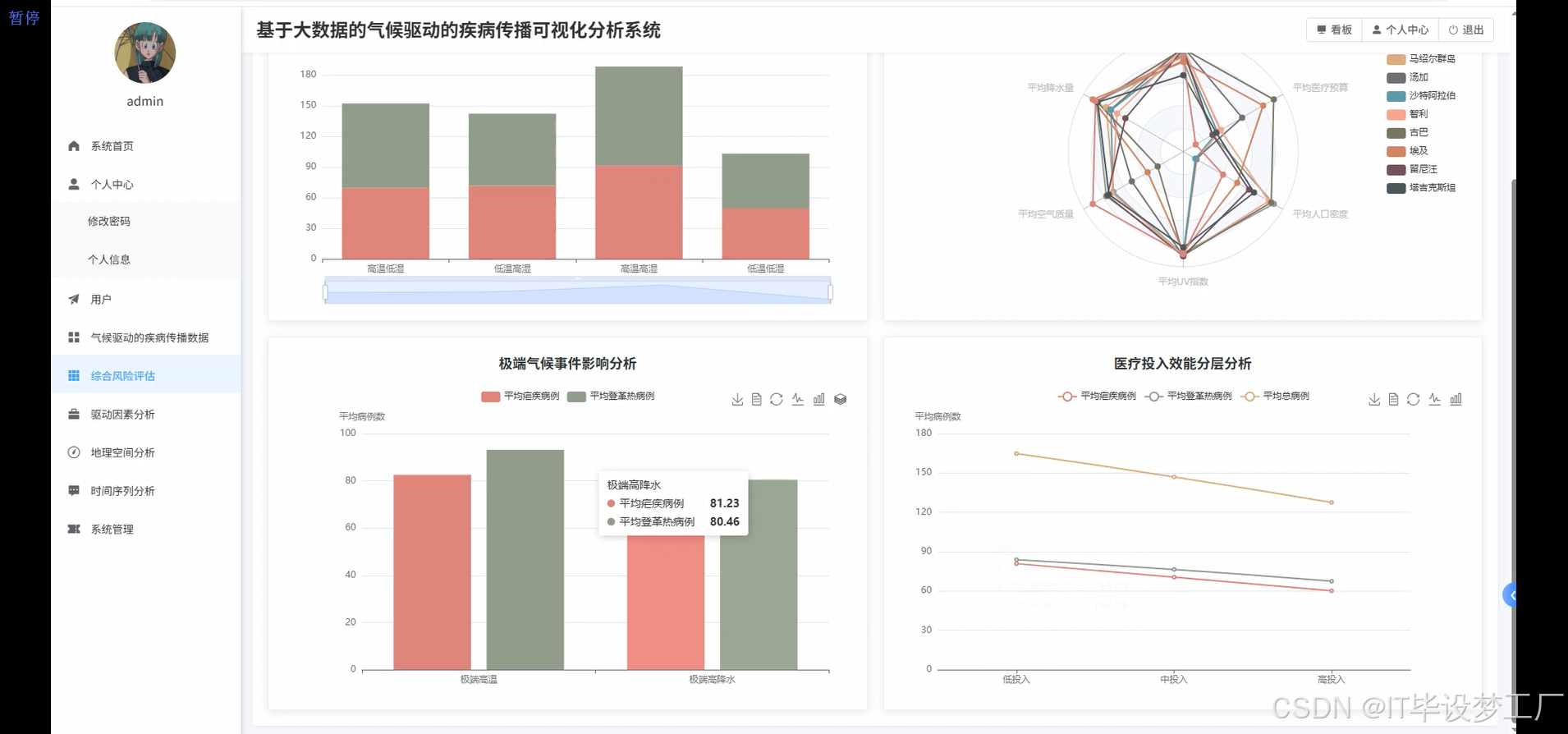
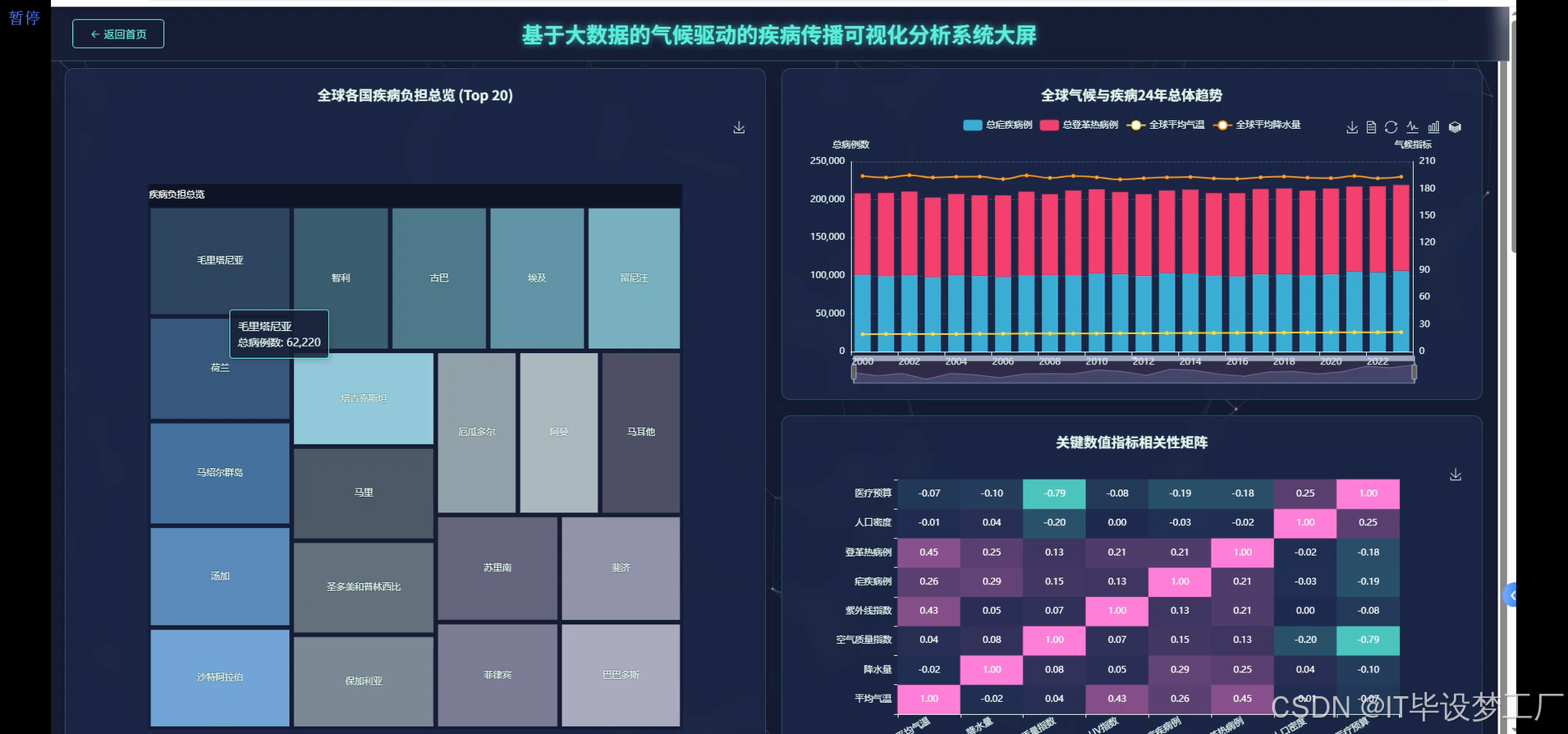
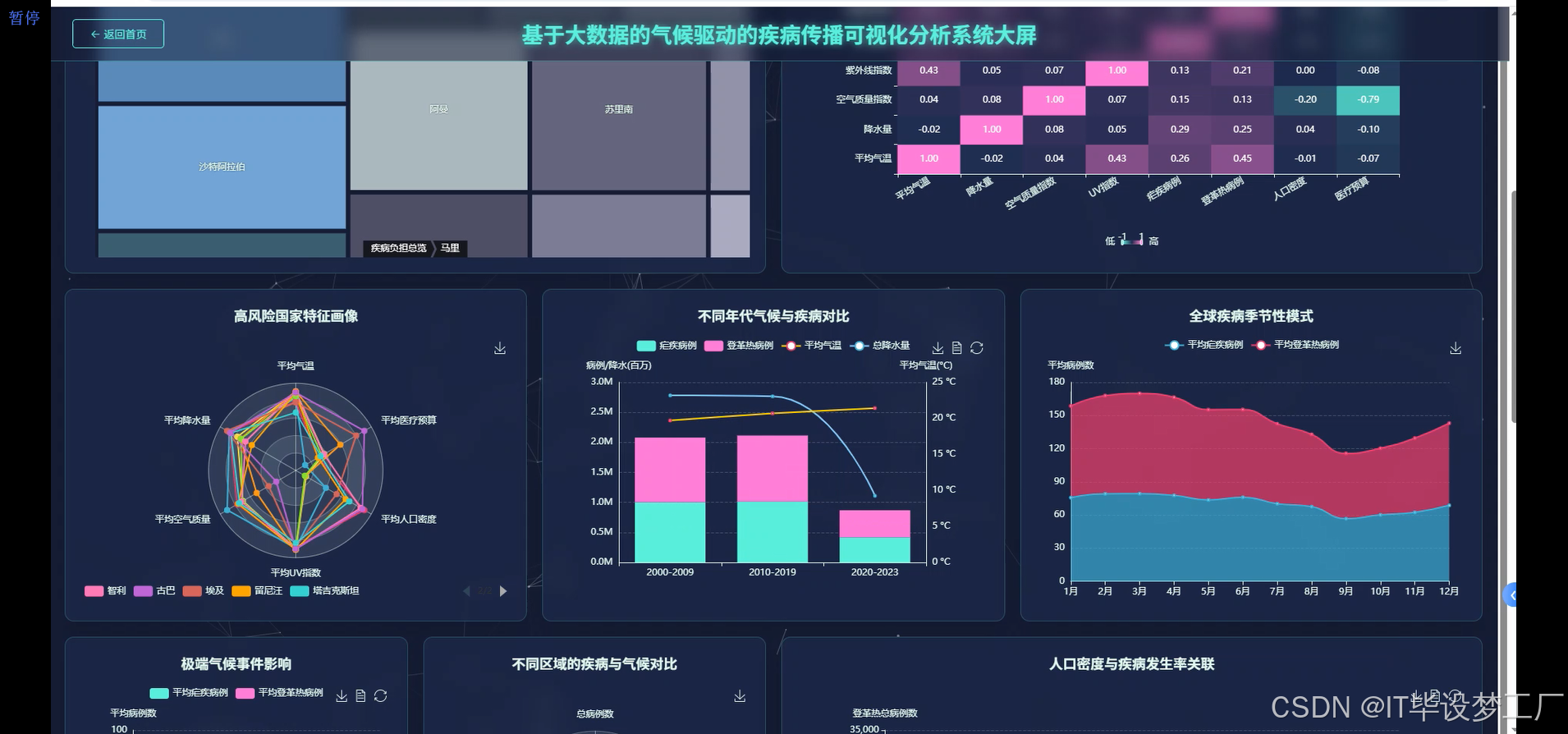
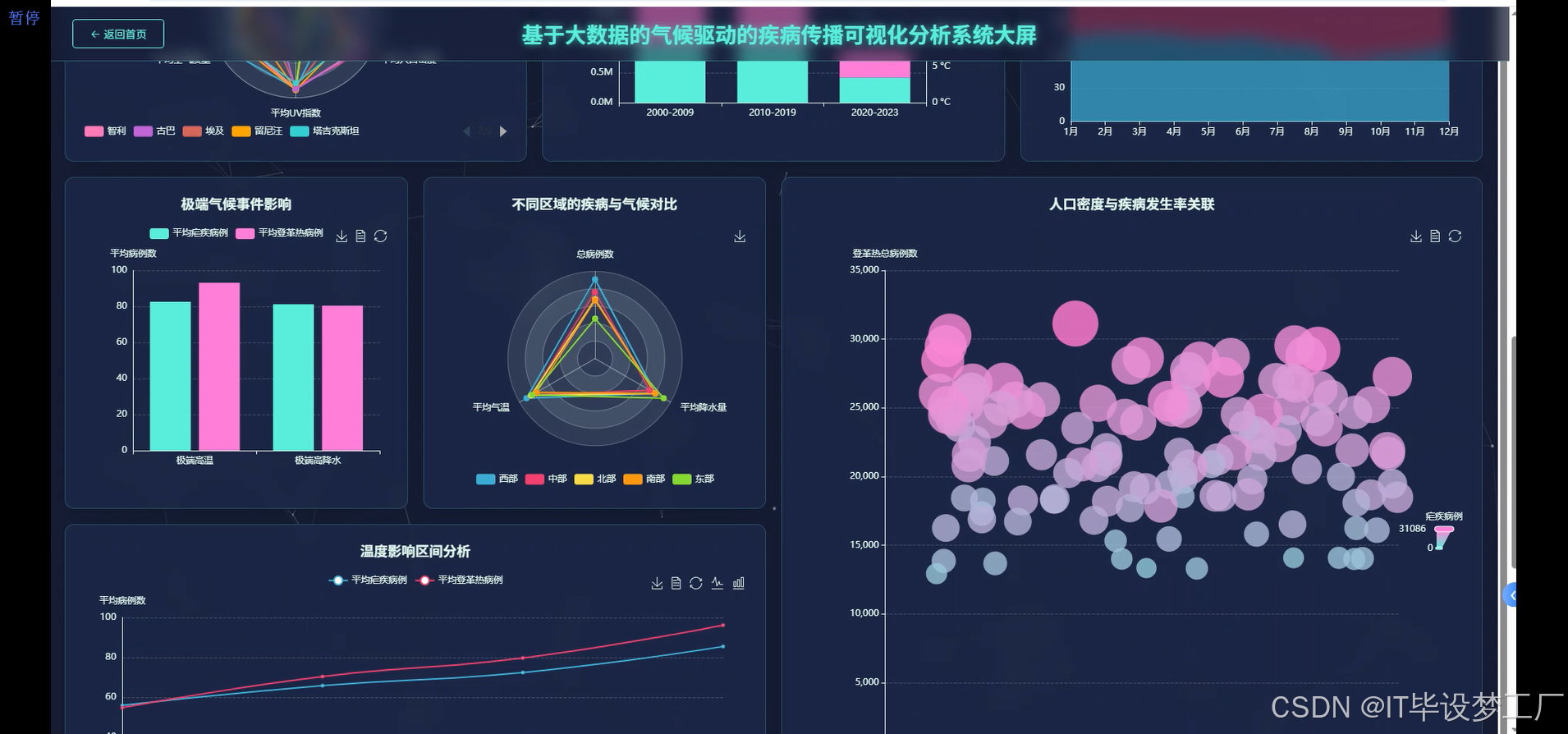
四、部分代码设计
- 项目实战-代码参考:
java(贴上部分代码)
from pyspark.sql import SparkSession
from pyspark.sql.functions import col, avg, sum, corr, when, count, stddev
import pandas as pd
import numpy as np
spark = SparkSession.builder.appName("ClimateDiseaseBigDataAnalysis").config("spark.sql.adaptive.enabled", "true").config("spark.sql.adaptive.coalescePartitions.enabled", "true").getOrCreate()
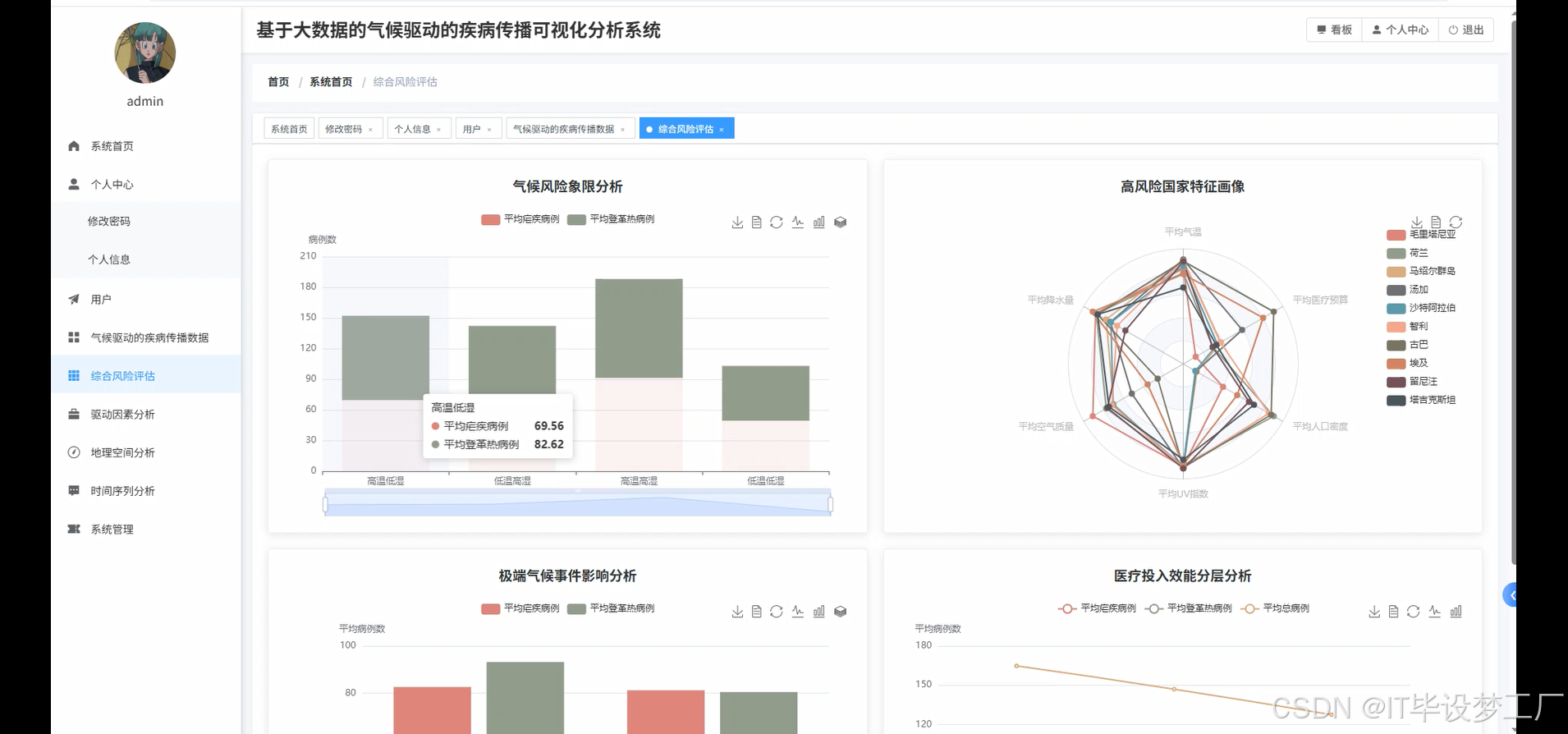
def geographic_spatial_analysis(climate_disease_df):
country_aggregated = climate_disease_df.groupBy("country", "region").agg(
avg("avg_temp_c").alias("avg_temperature"),
avg("precipitation_mm").alias("avg_precipitation"),
avg("air_quality_index").alias("avg_air_quality"),
sum("malaria_cases").alias("total_malaria"),
sum("dengue_cases").alias("total_dengue"),
avg("population_density").alias("avg_population_density"),
avg("healthcare_budget").alias("avg_healthcare_budget")
)
country_risk_scores = country_aggregated.withColumn("total_cases", col("total_malaria") + col("total_dengue"))
country_risk_scores = country_risk_scores.withColumn("climate_risk_factor",
(col("avg_temperature") / 35.0 * 0.3) +
(col("avg_precipitation") / 500.0 * 0.25) +
(col("avg_air_quality") / 150.0 * 0.2) +
(col("avg_population_density") / 500.0 * 0.25)
)
top_risk_countries = country_risk_scores.orderBy(col("total_cases").desc()).limit(20)
regional_comparison = country_aggregated.groupBy("region").agg(
avg("avg_temperature").alias("regional_avg_temp"),
avg("avg_precipitation").alias("regional_avg_precip"),
sum("total_malaria").alias("regional_malaria"),
sum("total_dengue").alias("regional_dengue")
)
population_density_correlation = climate_disease_df.select(
corr("population_density", "malaria_cases").alias("malaria_pop_corr"),
corr("population_density", "dengue_cases").alias("dengue_pop_corr")
).collect()[0]
geographic_clusters = country_risk_scores.withColumn("risk_level",
when(col("climate_risk_factor") > 0.7, "High")
.when(col("climate_risk_factor") > 0.4, "Medium")
.otherwise("Low")
)
risk_distribution = geographic_clusters.groupBy("risk_level").agg(
count("*").alias("country_count"),
avg("total_cases").alias("avg_cases_per_risk_level")
)
return {
"top_risk_countries": top_risk_countries.toPandas(),
"regional_comparison": regional_comparison.toPandas(),
"population_correlation": population_density_correlation,
"risk_distribution": risk_distribution.toPandas()
}
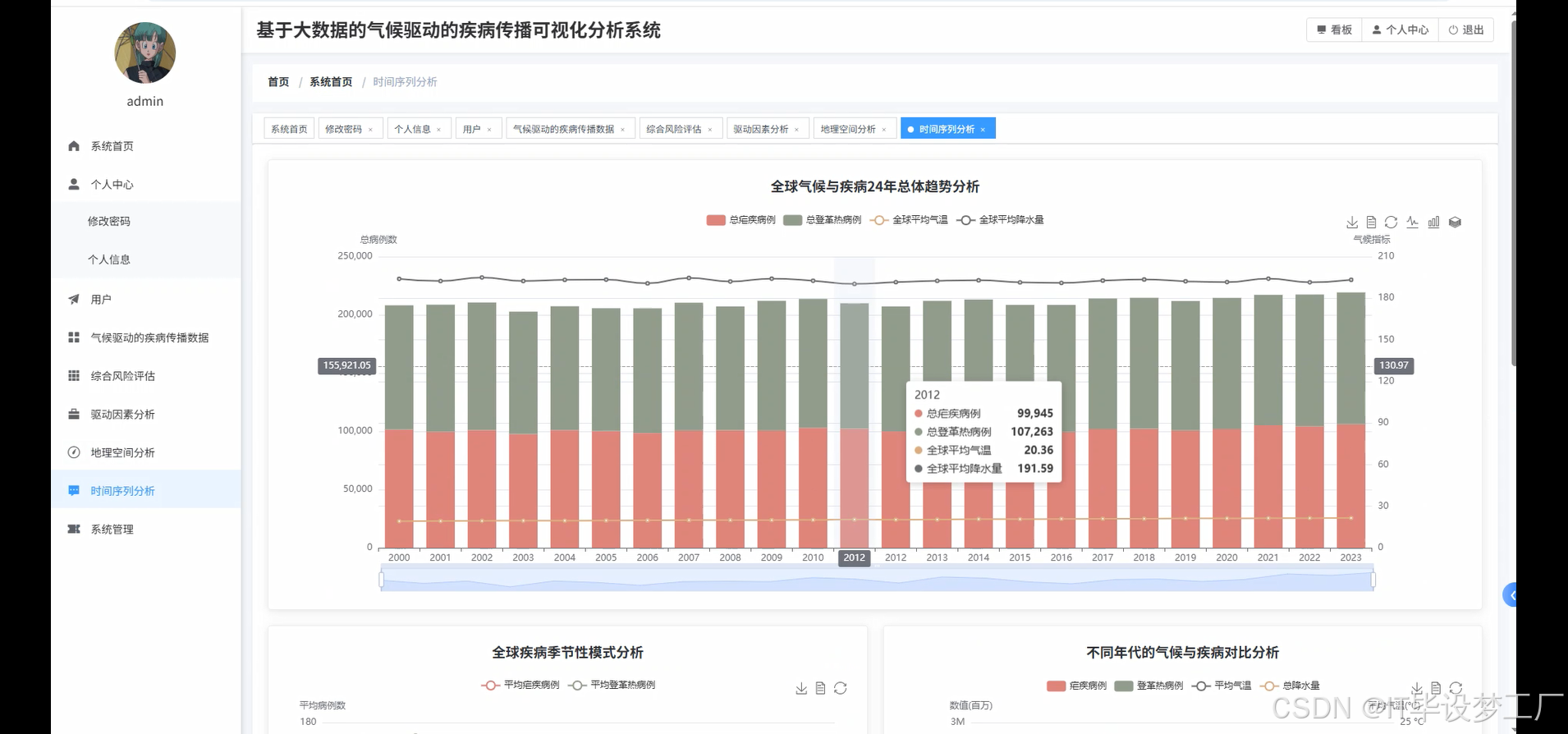
def time_series_trend_analysis(climate_disease_df):
yearly_trends = climate_disease_df.groupBy("year").agg(
avg("avg_temp_c").alias("yearly_avg_temp"),
avg("precipitation_mm").alias("yearly_avg_precip"),
sum("malaria_cases").alias("yearly_malaria_total"),
sum("dengue_cases").alias("yearly_dengue_total"),
avg("air_quality_index").alias("yearly_avg_aqi"),
avg("uv_index").alias("yearly_avg_uv")
).orderBy("year")
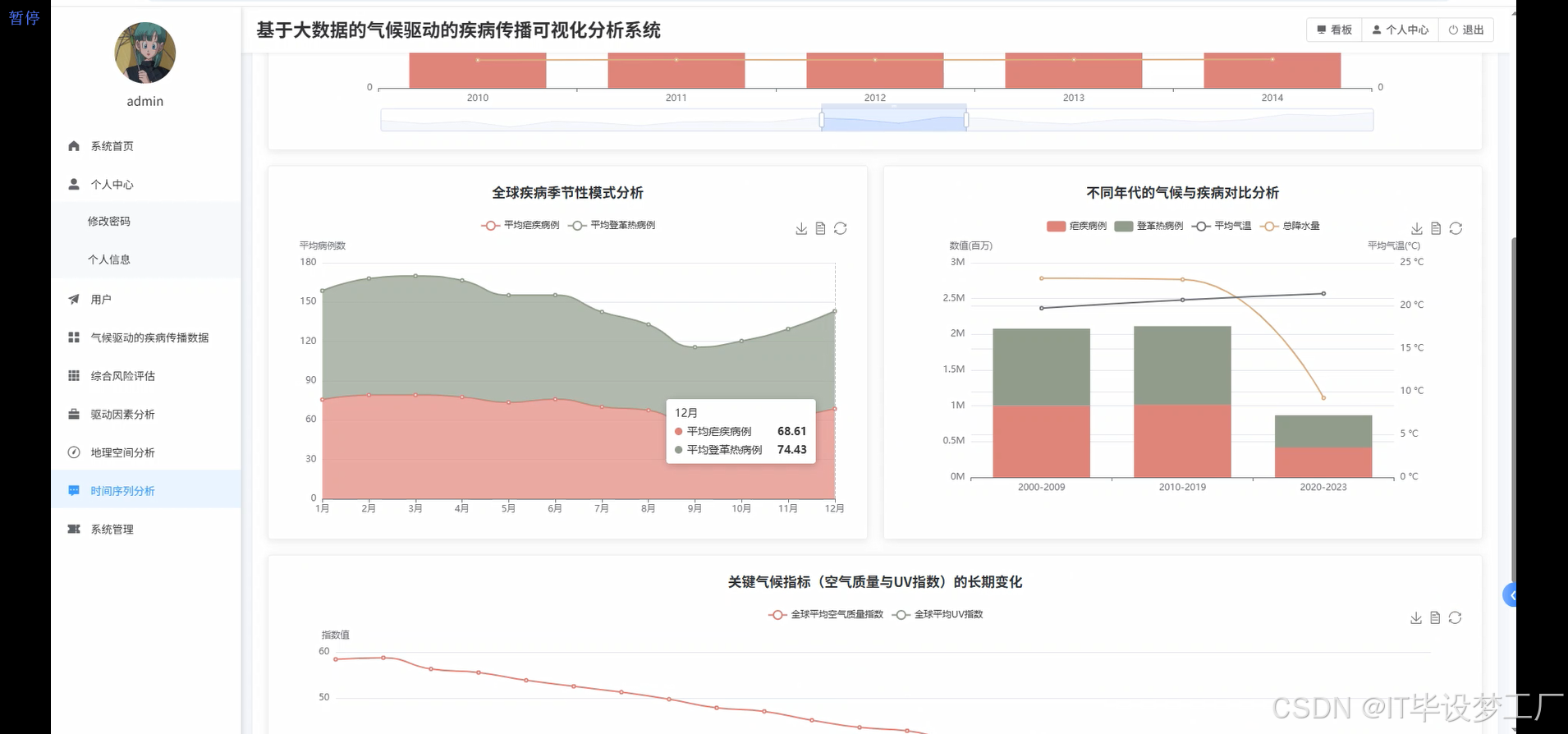
monthly_seasonal_patterns = climate_disease_df.groupBy("month").agg(
avg("malaria_cases").alias("monthly_avg_malaria"),
avg("dengue_cases").alias("monthly_avg_dengue"),
avg("avg_temp_c").alias("monthly_avg_temp"),
avg("precipitation_mm").alias("monthly_avg_precip")
).orderBy("month")
decade_comparison = climate_disease_df.withColumn("decade",
when(col("year") < 2010, "2000-2009")
.when(col("year") < 2020, "2010-2019")
.otherwise("2020-2023")
).groupBy("decade").agg(
avg("avg_temp_c").alias("decade_avg_temp"),
sum("malaria_cases").alias("decade_total_malaria"),
sum("dengue_cases").alias("decade_total_dengue"),
avg("air_quality_index").alias("decade_avg_aqi")
)
climate_disease_correlation_by_year = yearly_trends.select(
corr("yearly_avg_temp", "yearly_malaria_total").alias("temp_malaria_corr"),
corr("yearly_avg_precip", "yearly_dengue_total").alias("precip_dengue_corr"),
corr("yearly_avg_aqi", "yearly_malaria_total").alias("aqi_malaria_corr")
).collect()[0]
extreme_climate_months = climate_disease_df.filter(
(col("avg_temp_c") > 35) | (col("precipitation_mm") > 400) | (col("air_quality_index") > 120)
).groupBy("year", "month").agg(
avg("malaria_cases").alias("extreme_month_malaria"),
avg("dengue_cases").alias("extreme_month_dengue"),
count("*").alias("extreme_records_count")
)
temperature_trend_analysis = yearly_trends.select("year", "yearly_avg_temp").toPandas()
temp_slope = np.polyfit(temperature_trend_analysis['year'], temperature_trend_analysis['yearly_avg_temp'], 1)[0]
return {
"yearly_trends": yearly_trends.toPandas(),
"monthly_patterns": monthly_seasonal_patterns.toPandas(),
"decade_comparison": decade_comparison.toPandas(),
"correlation_analysis": climate_disease_correlation_by_year,
"extreme_climate_impact": extreme_climate_months.toPandas(),
"temperature_trend_slope": temp_slope
}
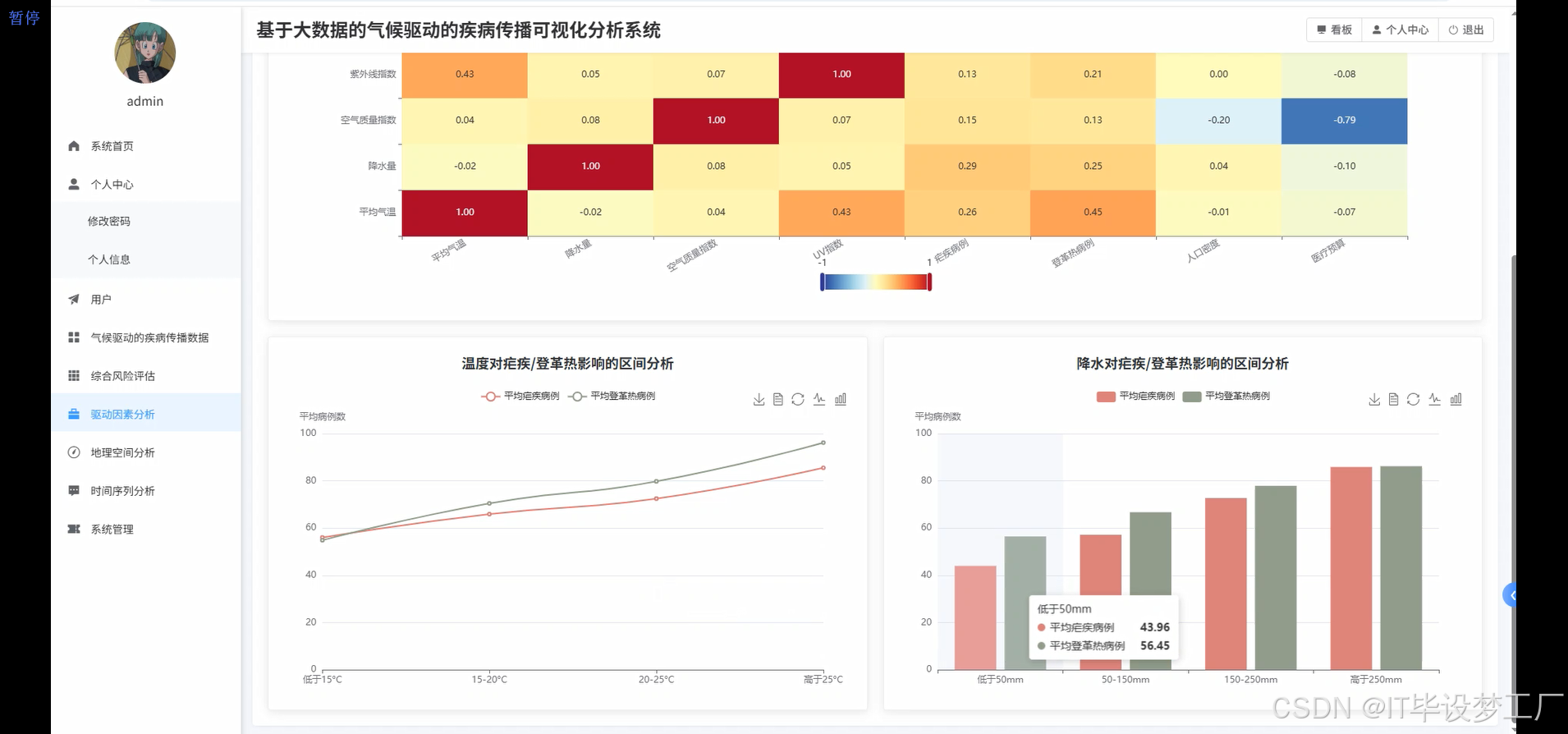
def climate_driving_factors_analysis(climate_disease_df):
correlation_matrix = climate_disease_df.select(
corr("avg_temp_c", "malaria_cases").alias("temp_malaria_corr"),
corr("avg_temp_c", "dengue_cases").alias("temp_dengue_corr"),
corr("precipitation_mm", "malaria_cases").alias("precip_malaria_corr"),
corr("precipitation_mm", "dengue_cases").alias("precip_dengue_corr"),
corr("air_quality_index", "malaria_cases").alias("aqi_malaria_corr"),
corr("air_quality_index", "dengue_cases").alias("aqi_dengue_corr"),
corr("uv_index", "malaria_cases").alias("uv_malaria_corr"),
corr("uv_index", "dengue_cases").alias("uv_dengue_corr"),
corr("population_density", "malaria_cases").alias("pop_malaria_corr"),
corr("population_density", "dengue_cases").alias("pop_dengue_corr"),
corr("healthcare_budget", "malaria_cases").alias("health_malaria_corr"),
corr("healthcare_budget", "dengue_cases").alias("health_dengue_corr")
).collect()[0]
temperature_interval_analysis = climate_disease_df.withColumn("temp_interval",
when(col("avg_temp_c") < 15, "Below_15")
.when(col("avg_temp_c") < 25, "15_to_25")
.when(col("avg_temp_c") < 35, "25_to_35")
.otherwise("Above_35")
).groupBy("temp_interval").agg(
avg("malaria_cases").alias("avg_malaria_by_temp"),
avg("dengue_cases").alias("avg_dengue_by_temp"),
count("*").alias("records_count")
)
precipitation_interval_analysis = climate_disease_df.withColumn("precip_interval",
when(col("precipitation_mm") < 50, "Very_Low")
.when(col("precipitation_mm") < 150, "Low")
.when(col("precipitation_mm") < 250, "Medium")
.otherwise("High")
).groupBy("precip_interval").agg(
avg("malaria_cases").alias("avg_malaria_by_precip"),
avg("dengue_cases").alias("avg_dengue_by_precip"),
count("*").alias("records_count")
)
multi_factor_risk_assessment = climate_disease_df.withColumn("climate_quadrant",
when((col("avg_temp_c") > 25) & (col("precipitation_mm") > 150), "Hot_Wet")
.when((col("avg_temp_c") > 25) & (col("precipitation_mm") <= 150), "Hot_Dry")
.when((col("avg_temp_c") <= 25) & (col("precipitation_mm") > 150), "Cool_Wet")
.otherwise("Cool_Dry")
).groupBy("climate_quadrant").agg(
avg("malaria_cases").alias("quadrant_avg_malaria"),
avg("dengue_cases").alias("quadrant_avg_dengue"),
avg("air_quality_index").alias("quadrant_avg_aqi")
)
healthcare_effectiveness_analysis = climate_disease_df.withColumn("healthcare_level",
when(col("healthcare_budget") > 3000, "High_Budget")
.when(col("healthcare_budget") > 1500, "Medium_Budget")
.otherwise("Low_Budget")
).groupBy("healthcare_level").agg(
avg("malaria_cases").alias("avg_malaria_by_budget"),
avg("dengue_cases").alias("avg_dengue_by_budget"),
count("*").alias("budget_level_count")
)
return {
"correlation_matrix": correlation_matrix,
"temperature_intervals": temperature_interval_analysis.toPandas(),
"precipitation_intervals": precipitation_interval_analysis.toPandas(),
"climate_quadrants": multi_factor_risk_assessment.toPandas(),
"healthcare_effectiveness": healthcare_effectiveness_analysis.toPandas()
}五、系统视频
- 基于大数据的气候驱动的疾病传播可视化分析系统-项目视频:
大数据毕业设计选题推荐-基于大数据的气候驱动的疾病传播可视化分析系统-Hadoop-Spark-数据可视化-BigData
结语
大数据毕业设计选题推荐-基于大数据的气候驱动的疾病传播可视化分析系统-Hadoop-Spark-数据可视化-BigData
想看其他类型的计算机毕业设计作品也可以和我说~谢大家!
有技术这一块问题大家可以评论区交流或者私我~
大家可以帮忙点赞、收藏、关注、评论啦~
源码获取:⬇⬇⬇