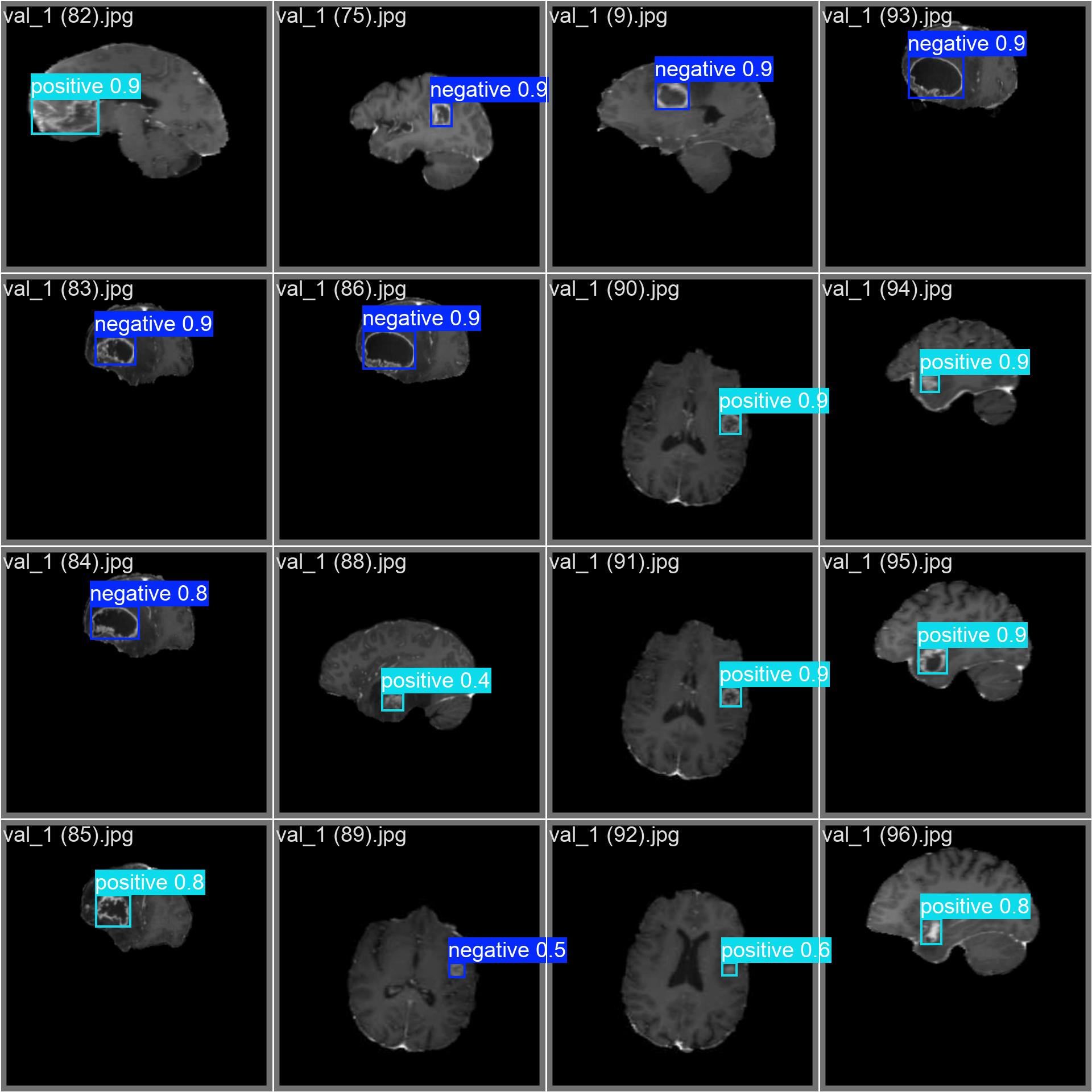
摘要 : 本文将详细介绍如何利用当前先进的深度学习目标检测算法 YOLOv12,结合强大的计算机视觉库 OpenCV,构建一个高效、准确的脑肿瘤检测系统。我们将从环境搭建、数据准备、模型训练到最终的检测应用,手把手带你完成整个流程,为医学影像辅助诊断、智能筛查等场景提供实用的解决方案。
关键词: YOLOv12, OpenCV, 脑肿瘤检测, 深度学习, 目标检测, Python
1. 引言
在神经医学影像领域,脑肿瘤(如胶质瘤、脑膜瘤等)的早期发现对患者预后至关重要。传统的阅片依赖放射科医生人工判读 MRI 图像,过程耗时且易受主观因素影响。随着人工智能技术的发展,利用计算机视觉自动检测脑部 MRI 切片中的肿瘤区域已成为研究热点和临床刚需。
YOLO(You Only Look Once)系列算法以其高速度和高精度在目标检测领域独树一帜。最新的 YOLOv12 在继承前代优点的同时,进一步优化了架构和训练策略,性能更上一层楼。OpenCV 作为最流行的开源计算机视觉库,提供了丰富的图像处理功能。本文将结合 YOLOv12 和 OpenCV,实现对脑肿瘤的精准检测,其中定义两类目标:
negative:表示图像中存在阴性肿瘤(如良性、低级别或无活性病灶)positive:表示图像中存在阳性肿瘤(如恶性、高级别或活跃增殖病灶)
⚠️ 注:此处"阴性/阳性"指肿瘤的生物学行为或临床意义,具体定义需依据标注规范。
2. 环境准备
2.1 软件依赖
首先,确保你的开发环境满足以下要求:
- Python: 推荐使用 Python 3.8 或更高版本。
- PyTorch: YOLOv12 基于 PyTorch 框架,需安装相应版本。
- YOLOv12 : 通过
ultralytics包安装。 - OpenCV: 用于图像处理和可视化。
- PyQT: 可视化UI(可选)。
安装命令:
bash
# 安装 PyTorch (根据你的CUDA版本选择)
pip install torch torchvision torchaudio
# 安装 YOLOv12
pip install ultralytics
# 安装 OpenCV
pip install opencv-python3. 数据集准备与标注
高质量的数据集是模型成功的关键。
3.1 数据收集
收集尽可能多的脑部 MRI 切片图像,涵盖:
- 不同肿瘤类型(胶质瘤、转移瘤、脑膜瘤等)
- 不同级别(低级别 vs 高级别)
- 不同模态(T1, T1ce, T2, FLAIR)
- 不同医院设备、扫描参数
- 不同病灶大小、位置、边界清晰度
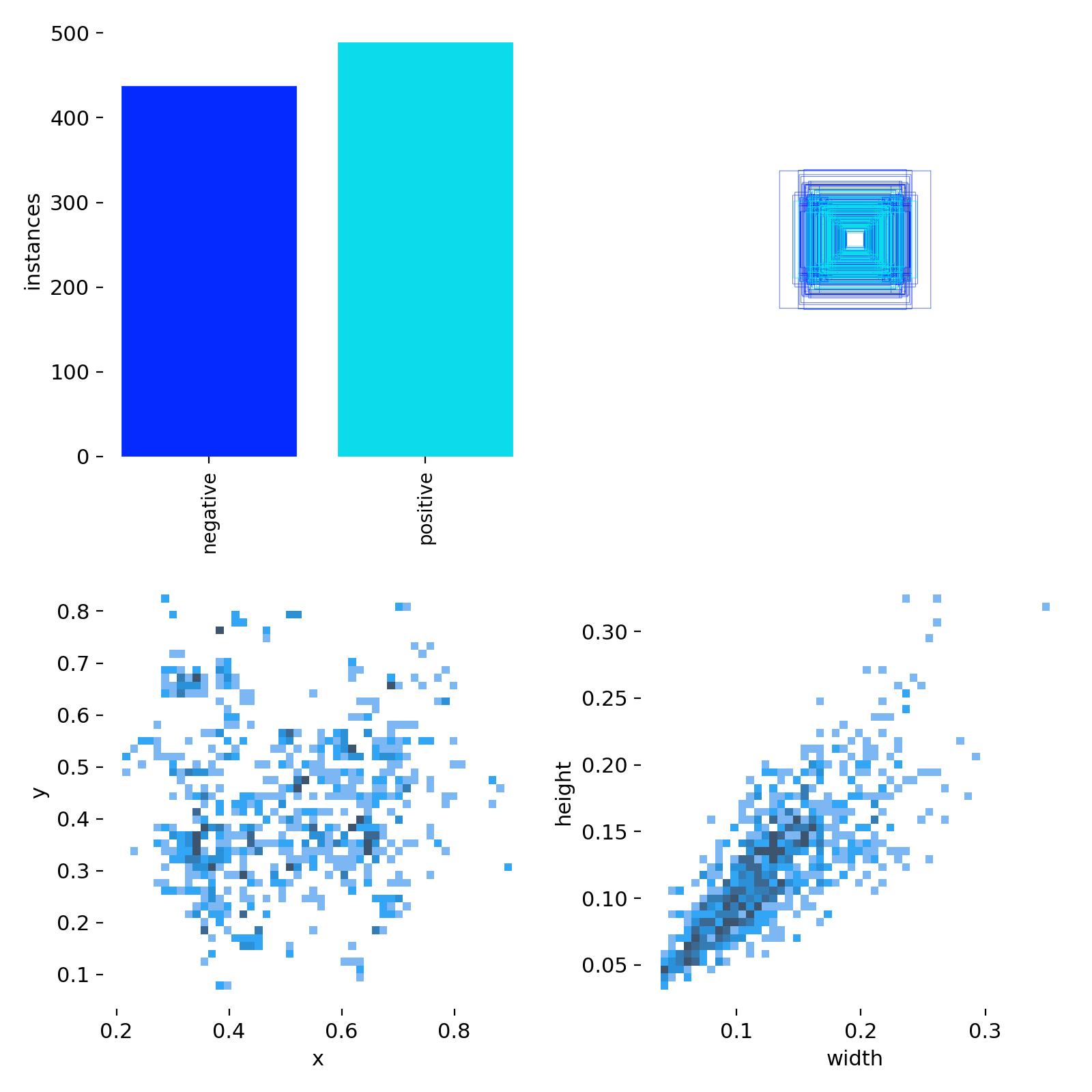

数据可来源于公开数据集(如 BraTS、Figshare Brain Tumor Dataset)或合作医疗机构(需脱敏与伦理审批)。
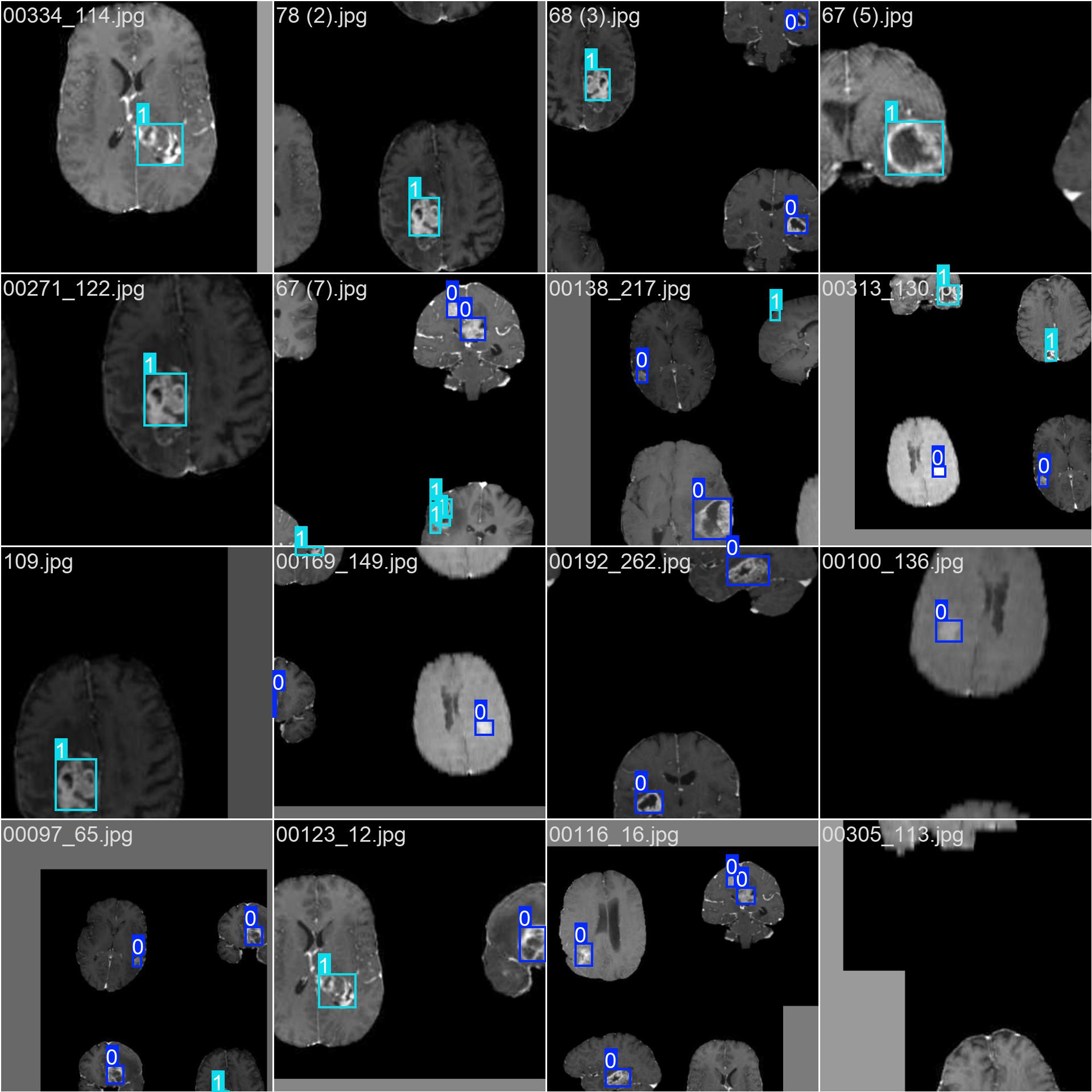
3.2 数据标注
使用标注工具(如 LabelImg, CVAT, Roboflow 等)对图像中的每一个肿瘤区域进行标注。
-
每个肿瘤需框出其边界(Bounding Box)
-
根据临床标准赋予类别标签:
negative或positive -
标注格式
:YOLO 使用
.txt文件存储标注信息,格式为:
txt<class_id> <x_center> <y_center> <width> <height>所有坐标值都是相对于图像宽高的归一化值(0-1)。
class_id = 0→negativeclass_id = 1→positive
3.3 数据集划分
将数据集划分为训练集(train)、验证集(val)和测试集(test),通常比例为 7:2:1 或 8:1:1。
3.4 数据集配置文件
创建一个 YAML 配置文件(如 brain_tumors.yaml),定义数据集路径和类别信息:
yaml
train: /path/to/dataset/images/train
val: /path/to/dataset/images/val
test: /path/to/dataset/images/test
# 类别数量
nc: 2
# 类别名称
names: ['negative', 'positive']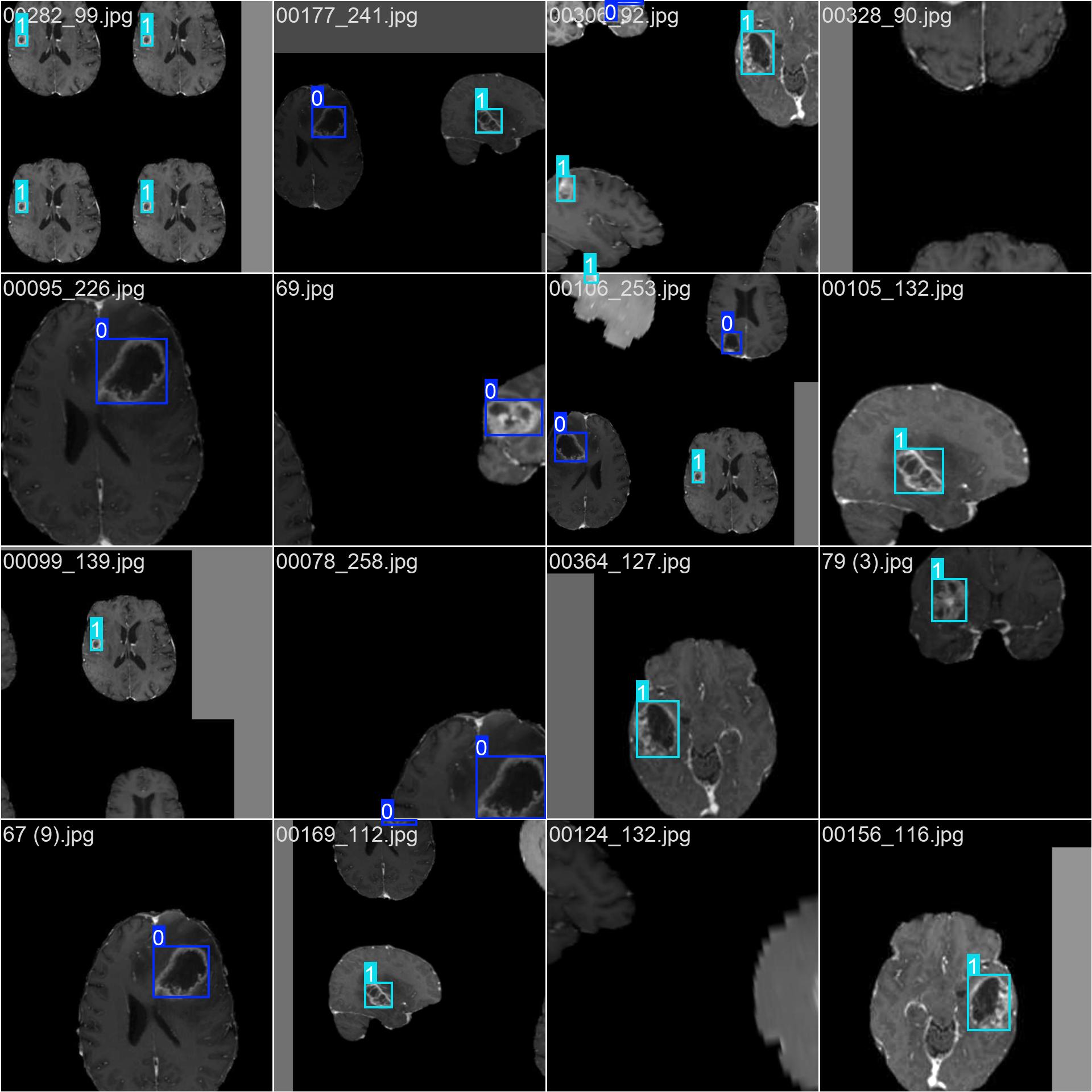
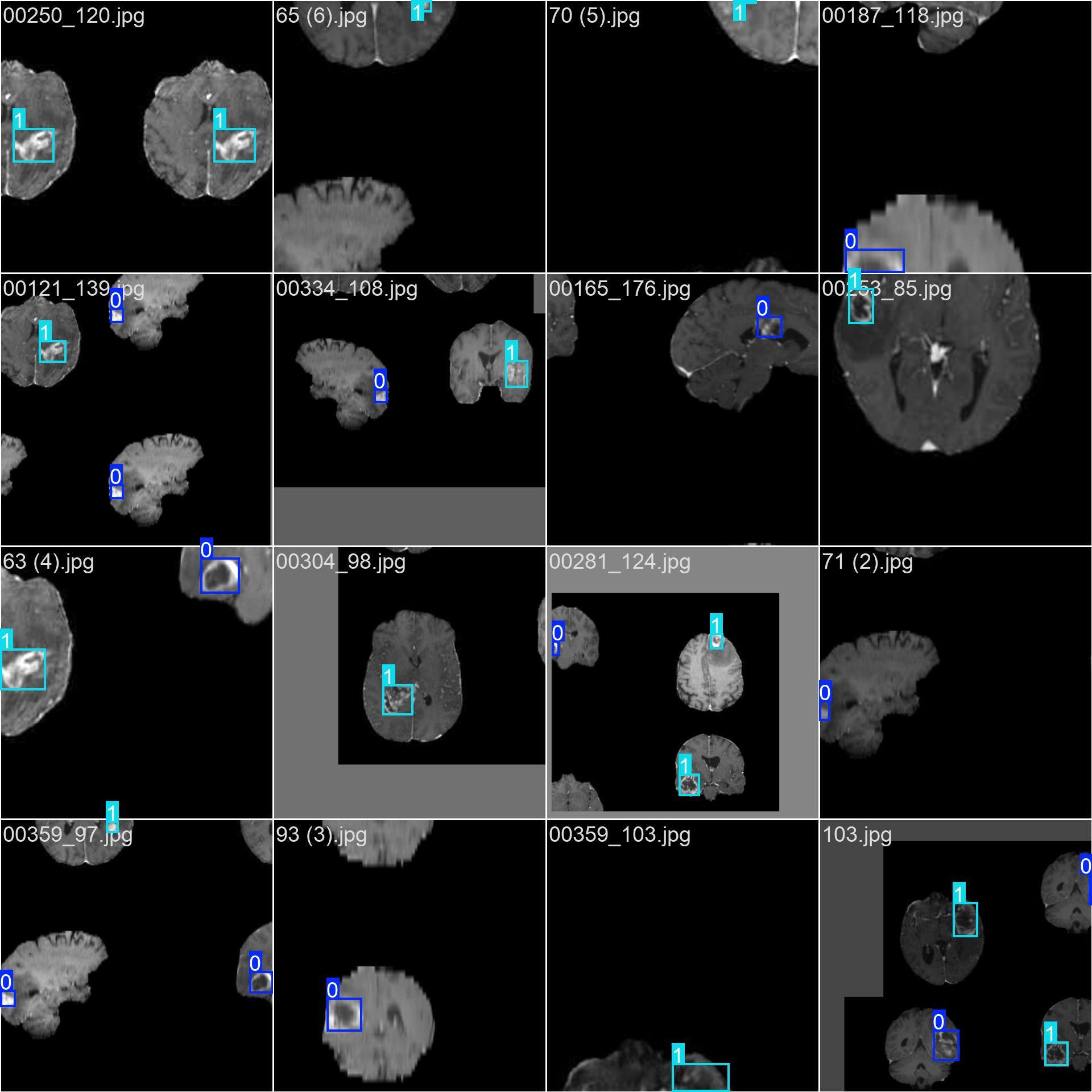
4. 模型训练
4.1 选择 YOLOv12 模型
YOLOv12 提供了多个预训练模型(yolov12n.pt, yolov12s.pt, yolov12m.pt, yolov12l.pt, yolov12x.pt),分别对应不同大小和精度。对于脑肿瘤检测(小目标、高相似度),推荐从 yolov12s.pt 或 yolov12m.pt 开始。
4.2 开始训练
使用 ultralytics 提供的命令行工具或 Python API 进行训练。
命令行方式:
bash
yolo train data=brain_tumors.yaml model=yolov12s.pt epochs=100 imgsz=512Python API 方式:
python
from ultralytics import YOLO
# 加载预训练模型
model = YOLO('yolov12s.pt') # ✅ 关键:使用 yolov12s.pt
# 训练模型
results = model.train(data='brain_tumors.yaml', epochs=100, imgsz=512)
# 评估模型
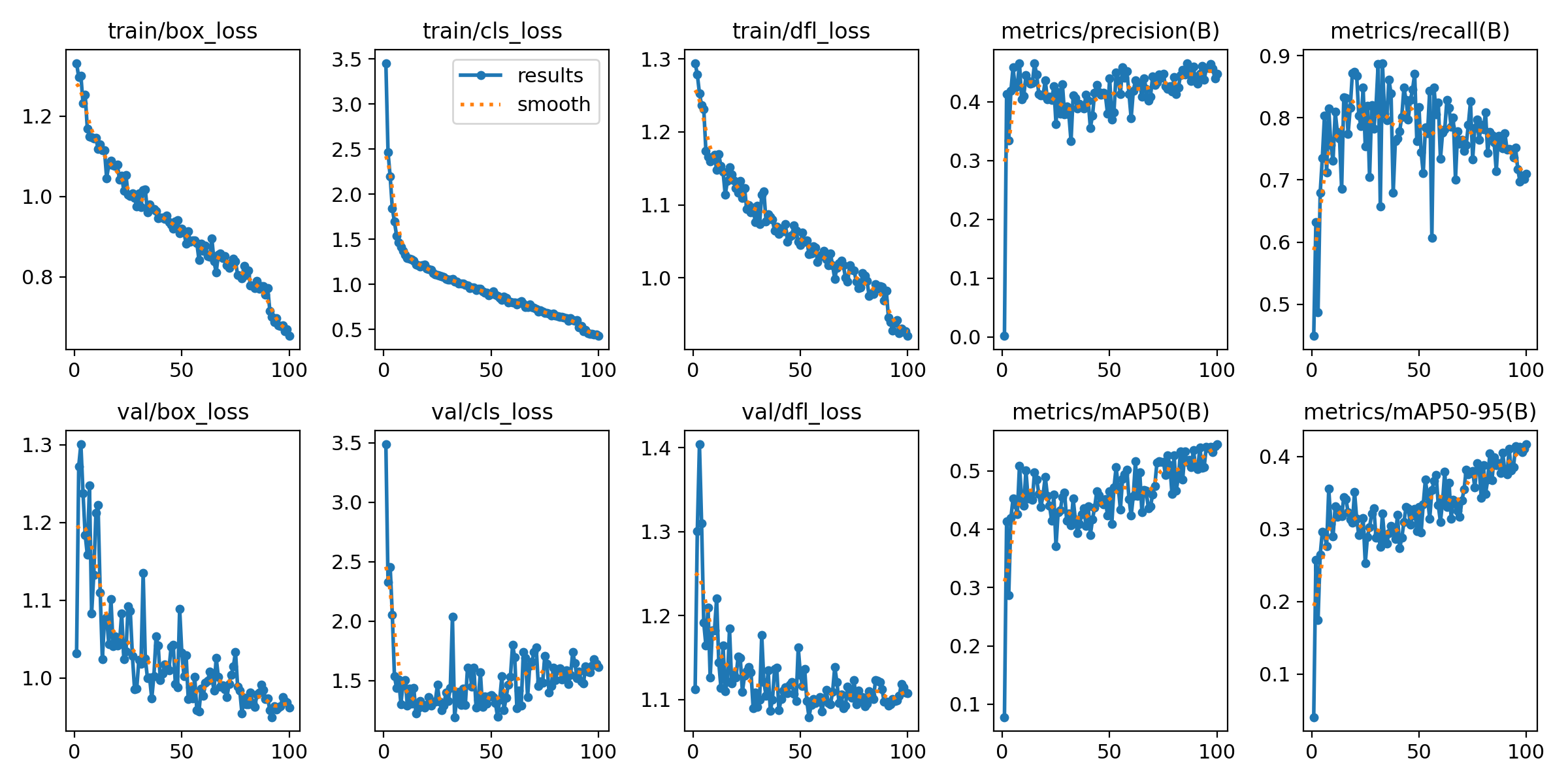
results = model.val()训练过程中,可以监控损失函数、mAP(mean Average Precision)等指标,判断模型收敛情况。
5. 脑肿瘤检测实现
训练完成后,使用训练好的模型进行检测。
5.1 加载模型
python
from ultralytics import YOLO
import cv2
# 加载训练好的模型
model = YOLO('runs/detect/train/weights/best.pt') # 替换为你的最佳权重路径5.2 图像检测
python
# 读取图像
img_path = 'mri_slice.jpg'
img = cv2.imread(img_path)
# 使用模型进行预测
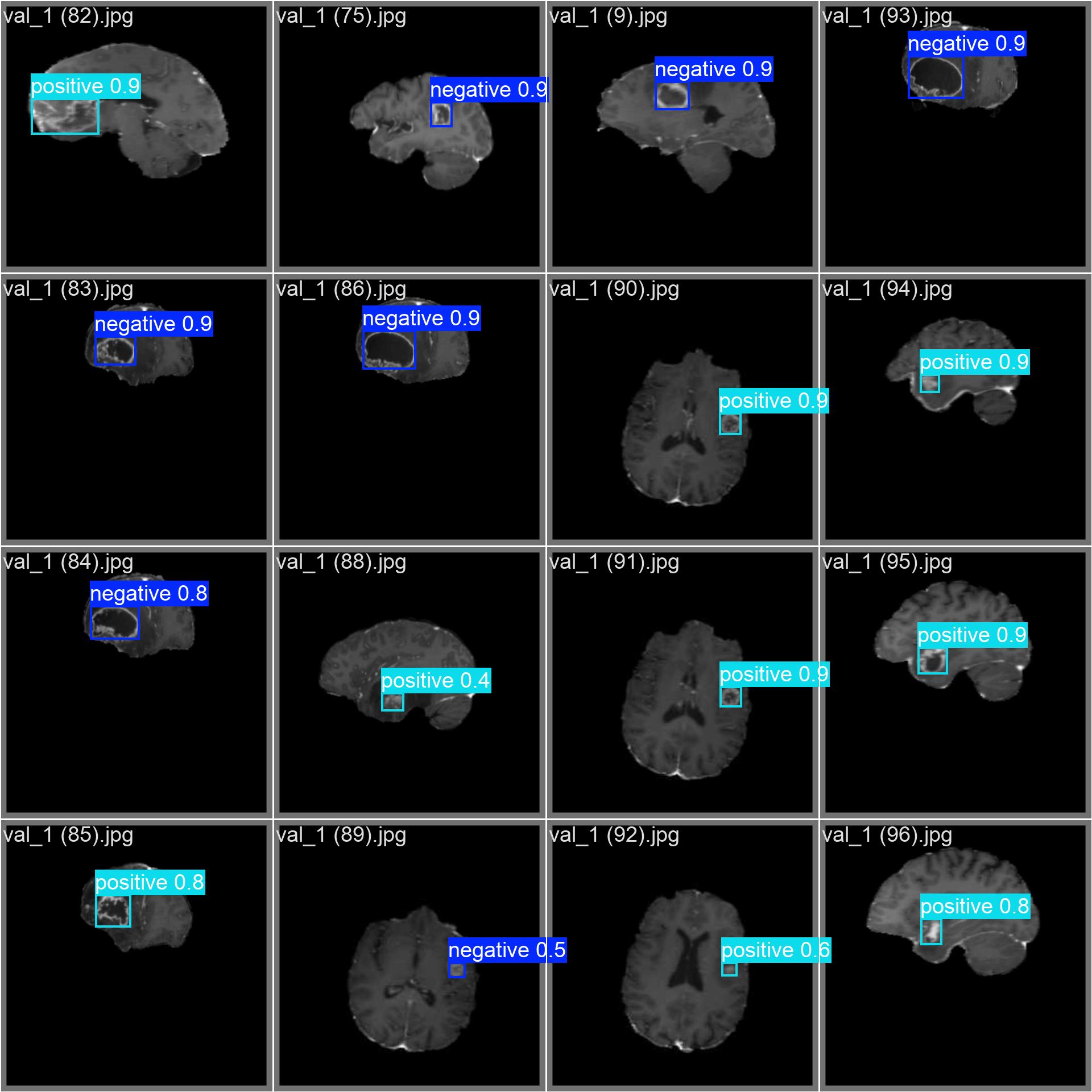
results = model(img)
# 解析结果
for result in results:
boxes = result.boxes # 获取边界框
for box in boxes:
# 提取坐标和置信度
x1, y1, x2, y2 = box.xyxy[0].cpu().numpy().astype(int)
conf = box.conf.cpu().numpy()[0]
cls = int(box.cls.cpu().numpy()[0])
label = model.names[cls] # 'negative' 或 'positive'
# 在图像上绘制边界框和标签
color = (0, 255, 0) if label == 'negative' else (0, 0, 255) # 绿色/红色区分
cv2.rectangle(img, (x1, y1), (x2, y2), color, 2)
cv2.putText(img, f'{label} {conf:.2f}', (x1, y1 - 10),
cv2.FONT_HERSHEY_SIMPLEX, 0.9, color, 2)
# 显示结果
cv2.imshow('Brain Tumor Detection', img)
cv2.waitKey(0)
cv2.destroyAllWindows()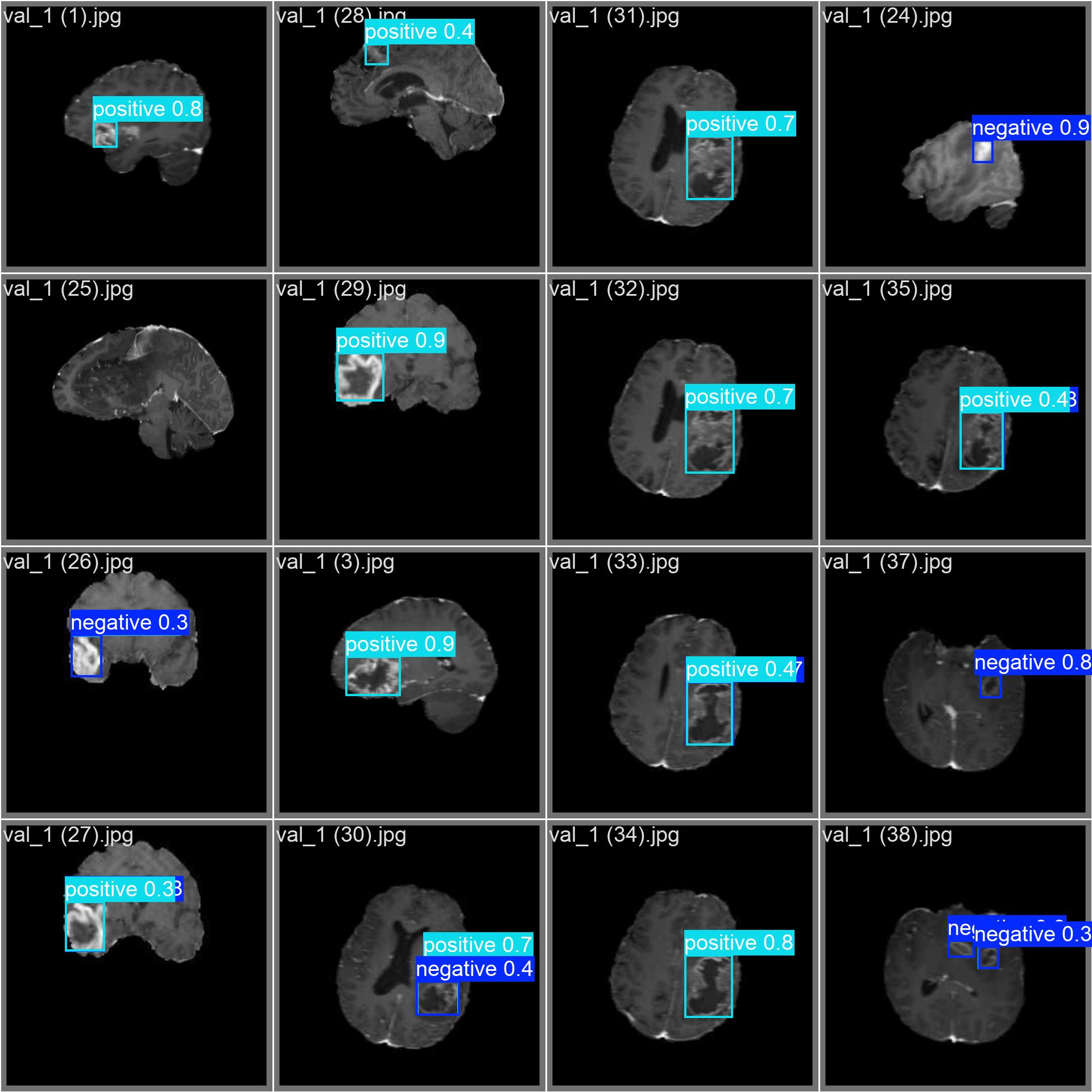

5.3 视频流/序列检测
可将上述逻辑应用于 MRI 序列的逐帧处理,实现整个扫描序列的自动筛查:
python
# 假设 mri_slices 是一个包含所有切片路径的列表
for slice_path in mri_slices:
img = cv2.imread(slice_path)
results = model(img)
# ... (同上,处理并保存结果)6. 结果与分析
- 精度 : 通过测试集评估模型的 mAP@0.5 等指标,YOLOv12 在高质量标注数据上通常能达到较高精度。
- 速度: YOLOv12 推理速度快,能满足单帧 MRI 图像的实时检测需求。
- 鲁棒性: 模型对不同 MRI 模态、噪声、部分遮挡具有一定鲁棒性。
挑战:
- 小目标检测: 早期微小肿瘤在图像中占比极小。
- 类别混淆: 阴性与阳性肿瘤在影像上可能外观高度相似。
- 边界模糊: 肿瘤浸润性生长导致边缘不清。
优化方向:
- 增加困难样本(小肿瘤、模糊边界)。
- 使用多模态 MRI 融合作为输入(如 T1ce + FLAIR)。
- 尝试更大的模型(
yolov12l.pt)或进行领域自适应微调。 - 结合分割模型(YOLOv12-Seg)获取更精确的肿瘤轮廓。
7. 总结
本文详细介绍了基于 YOLOv12 和 OpenCV 实现脑肿瘤检测的完整流程。通过定义 negative 和 positive 两类目标,我们构建了一个既能定位又能初步判别肿瘤性质的智能系统。该技术在医学影像辅助诊断、大规模筛查、手术规划等领域具有广阔的应用前景。
🔒 重要声明 :本系统旨在辅助医生提高工作效率,不能替代专业医学诊断。所有 AI 输出必须由执业医师复核确认。