文章目录
- 一.前言
- 二.核心技术&知识
-
- 1.PyQt5
- 2.YOLOv8
- 3.DeepSeek
- 4.CSV
- 5.多线程
- 6.结核杆菌
-
- 1.介绍(Introduce)
- 2.生物学特性(Characteristic)
- [3.传播与感染(Spread and Infection)](#3.传播与感染(Spread and Infection))
- [3.疾病类型(Disease type)](#3.疾病类型(Disease type))
- [4.诊断方式(Diagnostic methods)](#4.诊断方式(Diagnostic methods))
- [5.治疗与耐药性(Treatment and drug resistance)](#5.治疗与耐药性(Treatment and drug resistance))
- 三.核心功能
- 四.数据集
- 五.项目运行环境
- 六.总结
本系统功能强大!支持对结核杆菌进行目标检测,支持多种数据数据源输入并且接入了AI实现了对当前分析结果的评估,欢迎了解!

一.前言
近年来结核病在部分地区仍呈高发态势,实验室对痰涂片和显微镜图像的分析任务持续增长,显著加重了一线检测人员的工作压力。传统人工阅片在面对样本数量庞大、杆菌形态多变、图像质量参差不齐等问题时,往往容易出现漏检、误判与效率不稳定等情况,难以满足快速诊断与大规模筛查的现实需求。在此背景下,深度学习驱动的自动化检测技术逐渐成为实验室诊断升级的重要方向,其中 YOLOv8 以其高精度、强泛化和实时性优势,能够在复杂背景的涂片图像中稳定识别结核杆菌特征,大幅降低检测难度。与此同时,为了让自动检测系统更契合医学实验室的工作流,需要构建直观、可靠的本地化操作界面,因此 PyQt5 被用于搭建集图像处理、推理展示与结果管理于一体的可视化平台。随着大模型技术的发展,DeepSeek 的加入进一步增强了系统在数据特征解释、模型辅助优化和智能提示方面的能力,使平台从单纯的自动识别向智能化辅助诊断迈进。在多项技术的共同推动下,基于 PyQt5、YOLOv8 与 DeepSeek 的结核杆菌检测系统逐渐成为提升实验室诊断效率与准确性的可行方案。
二.核心技术&知识
在这章我将要介绍本系统的核心技术。
1.PyQt5
PyQt5 是一套用于创建跨平台桌面应用程序的 Python GUI 工具包,它是 Qt 应用框架的 Python 绑定。通过 PyQt5,开发者可以使用 Python 编写具有现代图形界面的应用程序,支持丰富的控件、信号与槽机制、窗口管理、事件处理等功能。它兼容主流操作系统(如 Windows、macOS 和 Linux),适用于开发各种规模的桌面软件,常与 Qt Designer 配合使用以加快开发效率。
2.YOLOv8
YOLOv8(You Only Look Once version 8)是由 Ultralytics 推出的最新一代实时目标检测模型,属于 YOLO 系列的改进版本。相比前代模型,YOLOv8 在精度、速度和灵活性上都有显著提升,支持目标检测、图像分割、姿态估计等多任务处理。它采用了更加高效的网络结构和训练策略,并提供开箱即用的 Python 接口和命令行工具,适用于边缘设备和云端部署,广泛应用于安防监控、自动驾驶、工业检测等场景。
3.DeepSeek
DeepSeek是由深度求索公司开发的AI大模型助手,作为纯文本模型,我擅长自然语言处理、文档分析和智能对话。当与YOLO(You Only Look Once)实时目标检测系统结合时,可以形成强大的多模态应用架构------YOLO系统负责实时视觉识别和目标检测,快速准确地识别图像或视频流中的物体;而我则对YOLO检测到的结果进行深度语义分析和上下文理解,提供物体属性的详细解读、场景描述、行为分析以及决策建议。这种结合使得计算机视觉的"看到"与AI的"理解"完美融合,可广泛应用于智能监控、自动驾驶、工业质检等领域,实现从视觉感知到智能决策的完整闭环。
4.CSV
本系统使用CSV进行数据的存储与数据导出。
CSV(Comma-Separated Values)是一种简单通用的文本文件格式,以纯文本形式存储表格数据。它用逗号分隔不同的字段,每行代表一个数据记录,类似于电子表格或数据库中的行。由于其格式简单、易于读写且兼容性强,CSV被广泛应用于数据交换、导出和存储,支持几乎所有常见的办公软件和编程语言处理,是数据分析和系统间传输结构化信息时最常用的轻量级格式之一。
5.多线程
QThread 是 PyQt5 提供的线程类,用于在界面程序中安全地执行耗时任务,从而避免界面阻塞和卡顿。它允许将数据处理、模型推理、文件读写等操作放到独立线程中运行,并通过信号与槽机制与主线程进行通信,确保界面更新的稳定性与线程之间的安全交互。使用 QThread 可以显著提升应用程序的响应速度,使界面在后台任务执行期间依旧保持流畅,同时具备良好的扩展性与可维护性,在复杂的桌面端应用开发中尤为常用。
6.结核杆菌
1.介绍(Introduce)
结核杆菌是一种由科赫发现的致病菌,属于分枝杆菌属,具有生长缓慢、耐酸染色和细胞壁富含脂质的特点。它主要通过呼吸道传播,在人体肺部定植后可能引发咳嗽、发热、盗汗、消瘦等典型症状,并在免疫力低下时进一步扩散至全身。由于其隐匿性强、潜伏期长和耐药性逐渐增强,结核杆菌的诊断与防控一直是全球公共卫生的重要挑战。及时发现并准确识别该菌对于阻断传播链和提高治疗成功率具有关键意义。
2.生物学特性(Characteristic)
它属于抗酸杆菌,因为细胞壁中含有大量分枝菌酸,导致普通染色不易进入,常用抗酸染色来观察。它的生长速度极慢,分裂一次大约需要 18 至 24 小时。细胞壁厚而富含蜡质,使其对干燥和部分化学物质有较强抵抗力。
3.传播与感染(Spread and Infection)
主要通过空气中的飞沫核传播。吸入后进入肺部,可被巨噬细胞吞噬,但不易被杀灭,因为它能阻断吞噬体与溶酶体融合,从而在细胞内长期存活。机体免疫系统会形成肉芽肿来限制其扩散。
3.疾病类型(Disease type)
最常见的是肺结核,表现为久咳、低热、盗汗、乏力和体重下降等。它还可以引起淋巴结结核、骨与关节结核、肾结核、结核性脑膜炎,以及严重的粟粒性结核。
4.诊断方式(Diagnostic methods)
常用方法包括痰涂片抗酸染色、培养(虽然耗时但非常准确)、核酸检测(如 GeneXpert)、皮肤试验(TST)和干扰素释放试验(IGRA)等。
5.治疗与耐药性(Treatment and drug resistance)
标准治疗一般需要多种药物联合使用,常见方案包括异烟肼、利福平、吡嗪酰胺和乙胺丁醇。疗程多为 6 个月或更长。结核杆菌容易产生耐药性,例如多重耐药(MDR-TB)和更严重的广泛耐药(XDR-TB),使治疗难度显著增加。
三.核心功能
1.登录注册
1.登录
软件启动后首先进入登录页面,用户需要输入正确的用户名和密码才能使用本系统的正式功能,登录页面整体采用了垂直布局,局部采用了水平布局,登录界面简约不简单,登录功能后端采用CSV本地文件存储用户信息,每次登录都是通过查库进行验证的,实现了流程标准化。
我们设计了简约的登录注册界面展示与用户交互的所有组件,登录注册界面的标题展示了系统的名称。
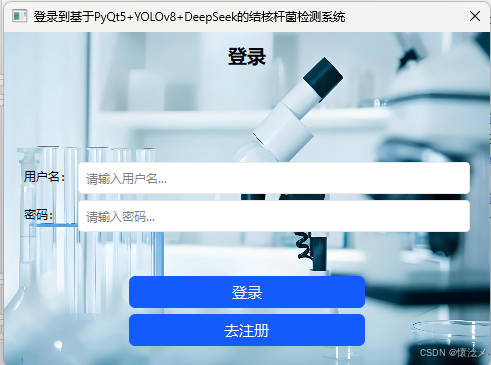
2.注册
没有账号的用户需要进行注册,注册操作流程十分简单,用户点击登录界面的注册按钮就跳转到了注册窗口,用户需要输入自定义的用户名和两次匹配的密码才能完成登录,值得一提的是,成功注册的用户软件会自动填写用户名和密码到登录界面,实现了登录流程路径的简化。
2.主界面
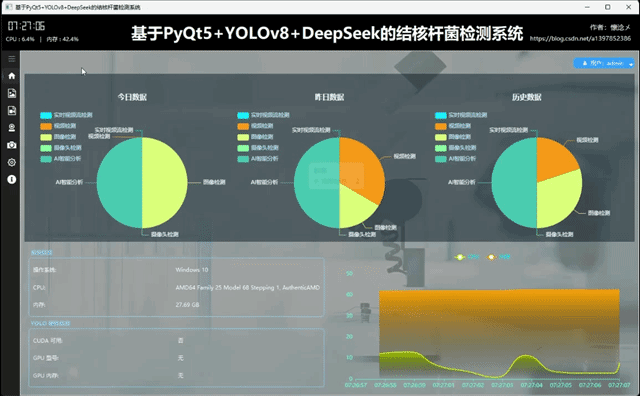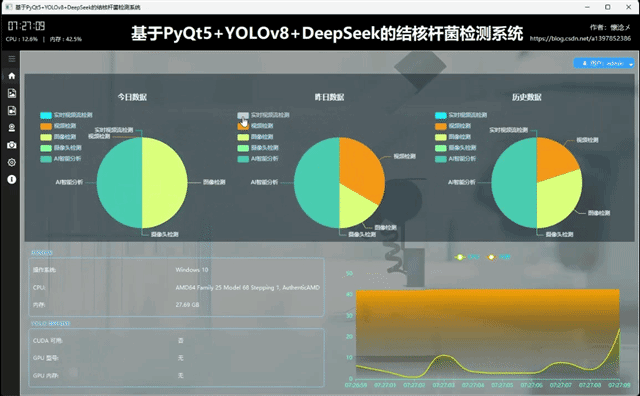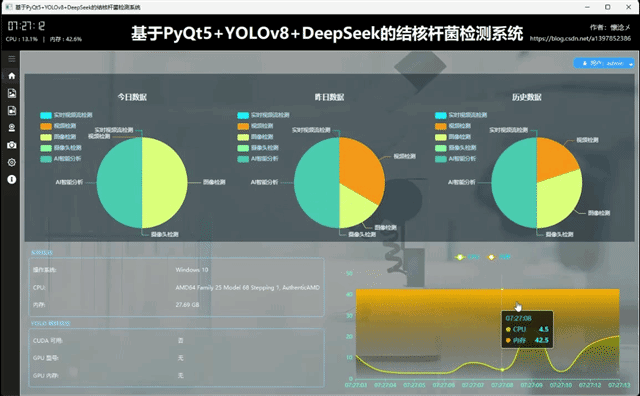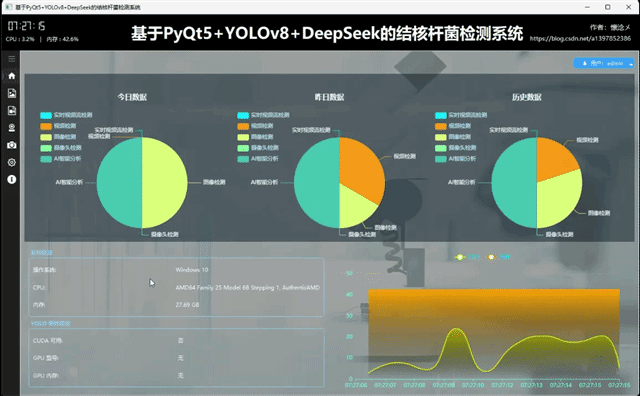
用户通过输入自己的用户名和密码登录到本系统后进入主界面,主界面内容十分丰富,我来一一介绍:首先软件整体是垂直布局,顶部是系统的标题,从左到右依次展示了系统的作者信息、系统名称、当前时间以及CPU内存占用情况,下方为水平布局,左侧是系统的导航区域,我们设计了windows风格支持展开与收缩的内容导航区域,右侧是内容核心区域,通过点击导航按钮切换展示内容,主界面主要展示了以日期为维度统计的数据、用户信息操作按钮、系统信息、系统环境信息以及实时CPU、内存可视化折线图
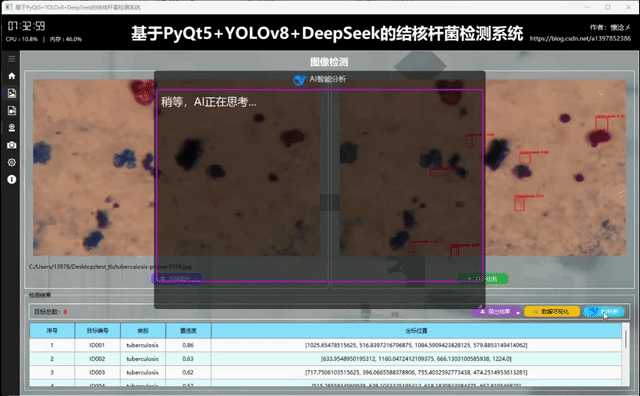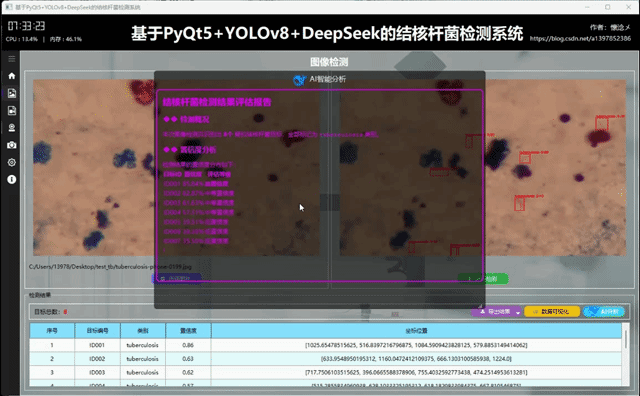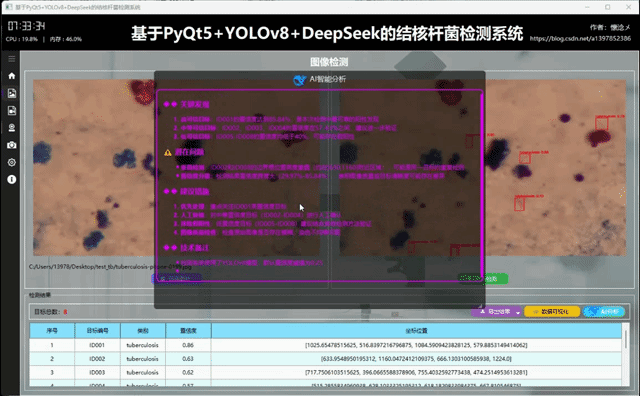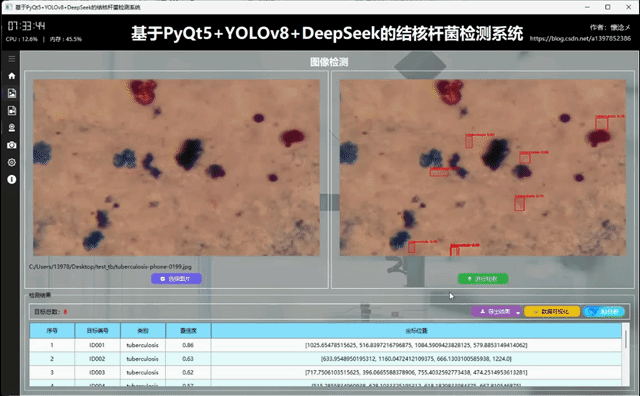
3.图像检测界面
1.检测结果展示
用户通过点击左侧导航栏按钮切换到图像检测界面,在此界面支持选择图像进行输入,用户选择完之后被选择的图像会展示在左侧并且展示图像绝对路径信息,用户可以通过点击右侧的"进行检测"按钮对输入的图像数据进行检测,系统会自动调用YOLOv8相关算法根据指定的参数对输入图像内容进行检测,最后将检测结果展示到右侧,这样用户可以通过比对左右图像的区别得到直观的检测结果,系统自动使用红色边框框选出目标区域并且使用红色文字展示出目标类别以及它的置信度,这些参数和展示效果都可以在设置页面进行详细设置。
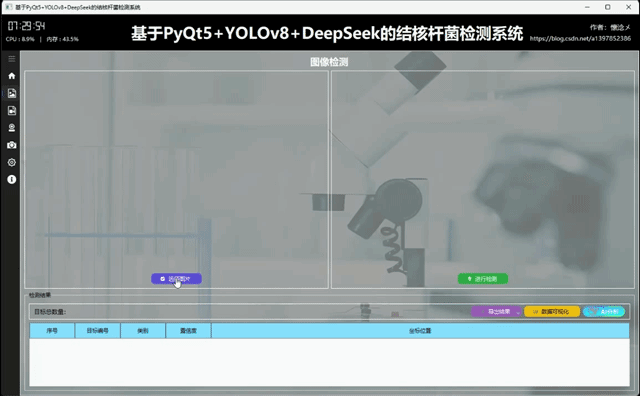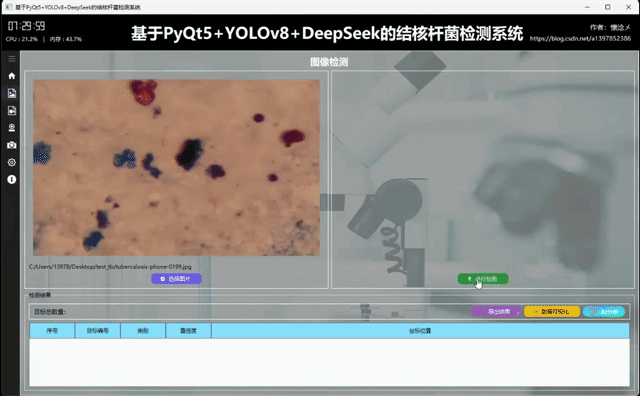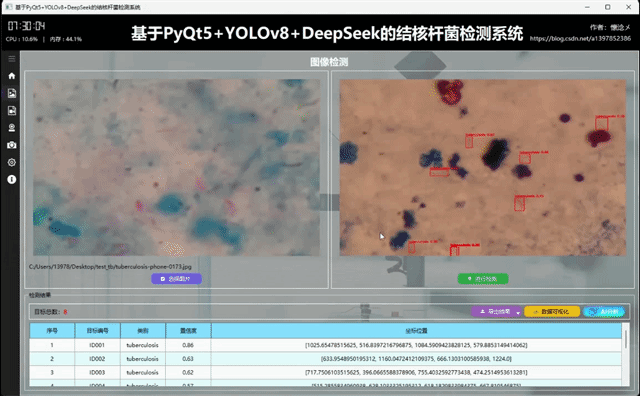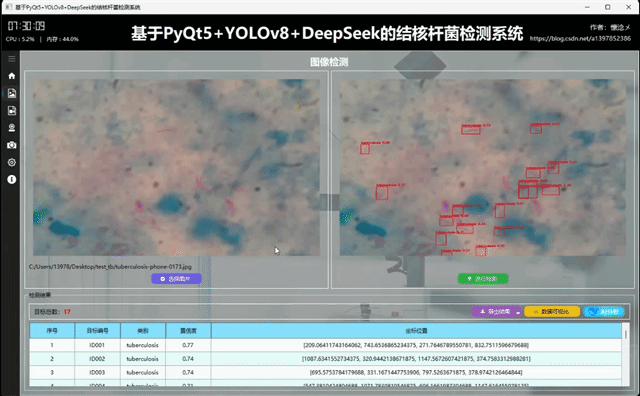
2.导出检测结果
我们设置了检测结果区域,包括检测目标数量的展示以及详细检测结果表格,用户可以更加直观地看到检测结果数据,另外当用户检测结束之后右侧的三个按钮自动设置为可以点击,这三个按钮的作用分别是:
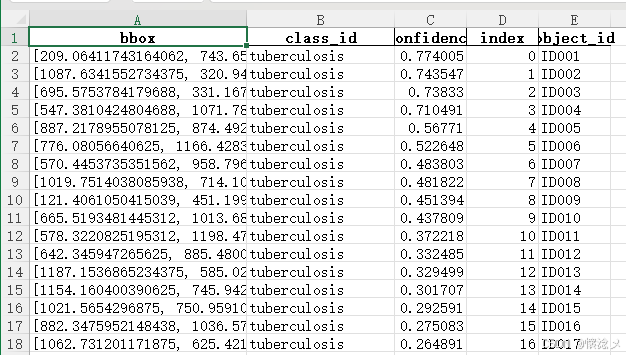
导出结果:用户可以将检测结果进行导出,导出的文件格式可以是Excel、CSV、TXT,可以根据情况自行选择导出格式,我截图给大家看下导出的CSV文件内容格式。
3.可视化展示
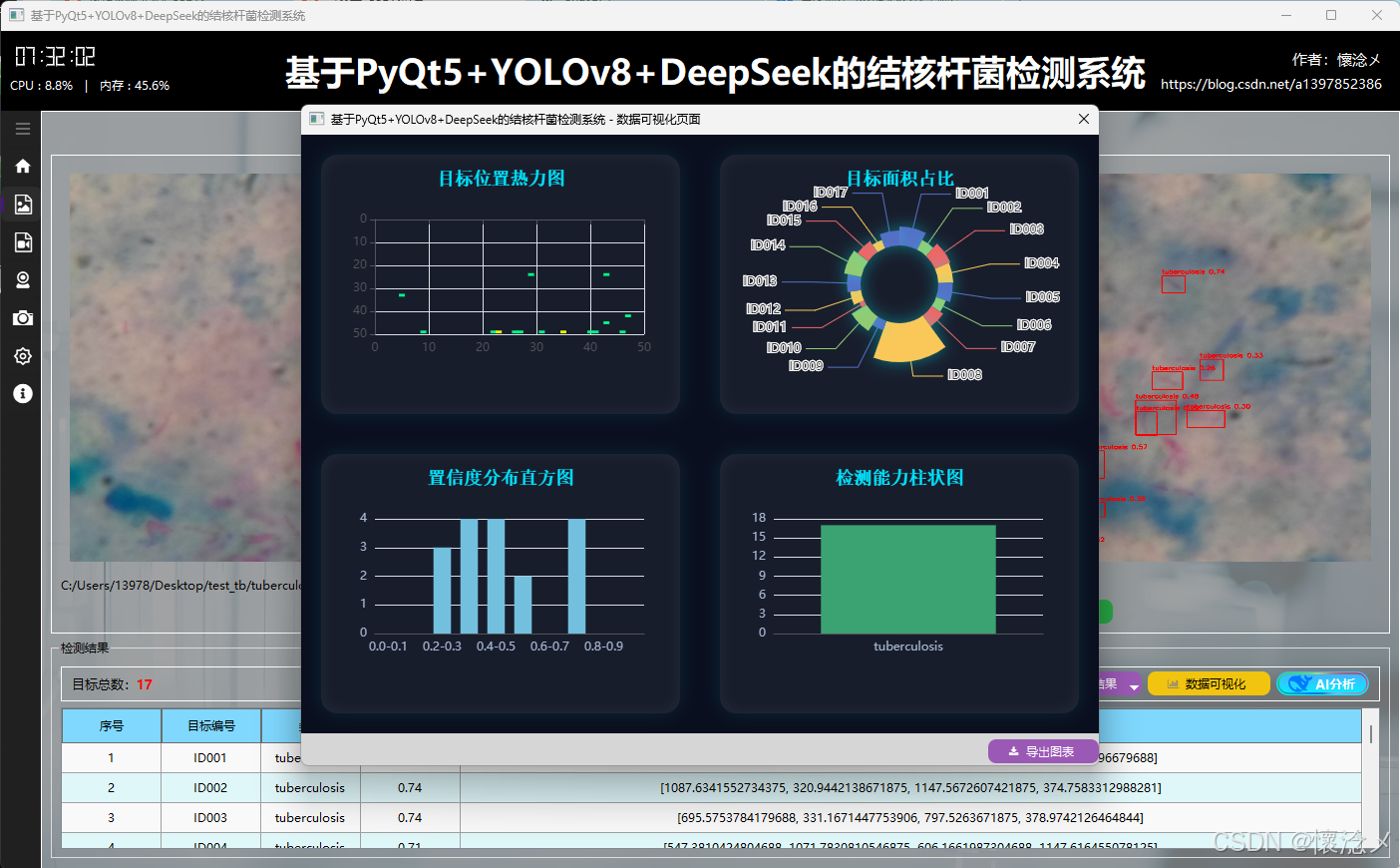
然后就是可视化展示,用户可以点击进行可视化按钮,查看对于本次检测的可视化效果,系统内置了四种可视化效果:分别是:目标位置热力图、目标面积占比、置信度分布直方图、检测能力柱状图,这些图标通过不同维度对当前数据进行了可视化展示,更便于用户理解,这里指的一体的是,支持可视化图表进行导出操作,用户可以点击紫色的导出按钮,对当前的可视化效果图表进行导出,生成一张本地的PNG图像文件。
4.AI(DeepSeek)智能分析
将YOLOv8球体检测系统与DeepSeek等AI大模型深度融合,能够实现从"单纯检测"到"智能认知"的跨越式升级。该系统在YOLOv8高效精准的球体定位能力基础上,融入了DeepSeek强大的语义理解和推理决策能力,不仅可以实时识别球体位置,还能深度分析场景语义、生成战术策略并提供智能决策支持,从而在体育分析、工业质检和机器人视觉等领域构建出更加智能化、自适应的一体化解决方案。
这里是软件的另外一个核心:AI智能分析,我们的目标检测系统接入了DeepSeek大模型,支持对当前检测结果数据进行AI分析,AI会通过不同维度对当前检测结果进行多角度分析,最后生成检测结果分析报告,用户可以根据这个结果对系统进行调整,不断完善系统功能和目标检测准确度!
在AI分析结束后下方会展示一些按钮,用户可以方便地复制结果、导出文本内容、生成PDF报告、重新生成以及关闭,多重的操作方式给于用户了多种选择!
4.视频检测界面
1.视频文件检测
我们的系统支持视频内容中的球体检测,支持输入的视频类型包括:视频文件、视频流以及摄像头,通过识别视频画面的内容对内容中的目标球体进行检测,试试标注与展示,通过相关帧率控制保证了视频的流畅性,用户可以通过比对左右两侧的画面使用肉眼评估当前检测结果,我们的视频检测界面拥有图像检测界面相同的操作功能,这里不多赘述。
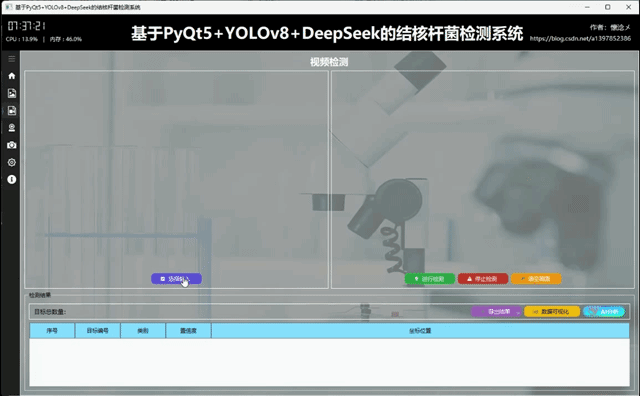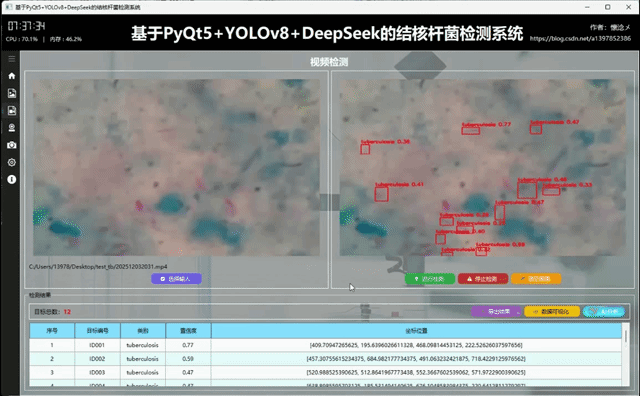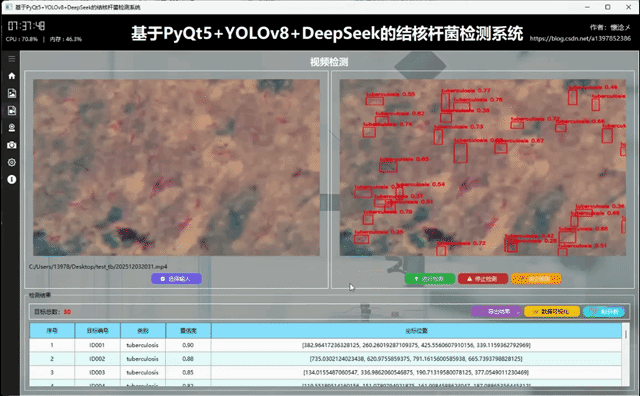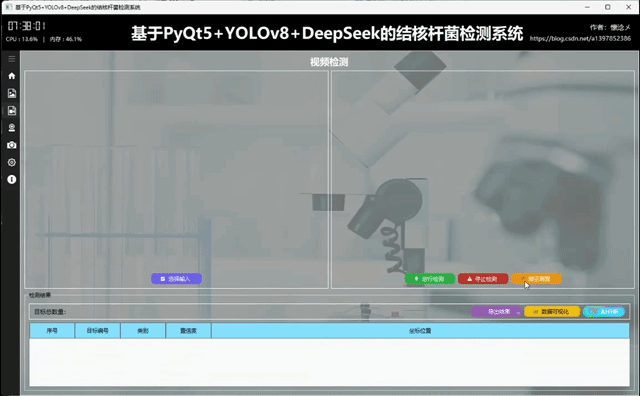
2.摄像头内容检测
用户点击"进行检测"按钮之后系统会自动调用摄像头,打开摄像头展示摄像头画面,实时检测目标画面中的球体,通过左右画面比对让用户看到检测结果,这里本人就不露脸啦~
5.系统设置界面
我们的系统是支持简单的参数设置的,具体可以设置目标检测模型、置信度阈值(Conf)、交并比阈值(IOU)、还有一些检测结果控制参数,比如检测框展示、目标类别展示、目标置信度展示,用户可以点击绿色的刷新按钮刷新可用模型,亦可通过点击退出按钮退出系统或者退出登录,本设置页面实现了目标检测参数的灵活配置!
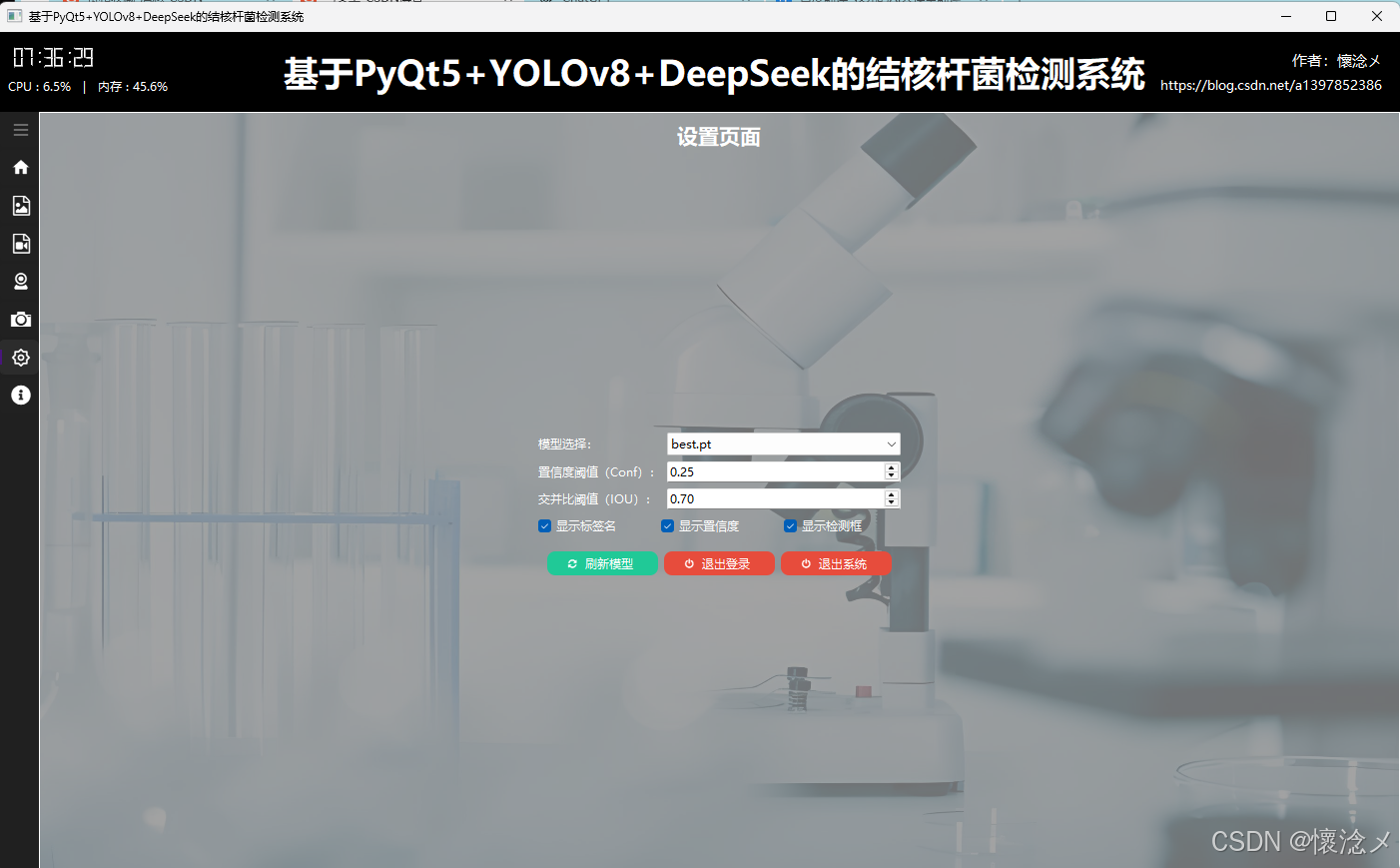
6.关于软件界面
我们使用富文本html的形式展示了软件相关的信息,包括系统用到的相关技术,对于二维的数据使用二维表进行了展示,最底部放置了四个按钮,分别是:关于YOLO、关于软件、关于作者、关于QT,点击之后都会弹出对应的信息提示框,这个页面的作用是让用户更多的了解软件和创作者信息,跨过技术的鸿沟!

四.数据集
基于 PyQt5 + YOLOv8 + DeepSeek 的结核杆菌检测系统"旨在解决传统结核杆菌识别流程中效率低、依赖经验强与误判率较高等问题。结核病长期以来是全球范围内的重大公共卫生挑战,其临床检测多依赖人工显微镜阅片,流程繁琐、主观性强,在基层医疗机构尤为突出。随着深度学习与医学图像分析技术的发展,利用 YOLOv8 进行快速目标检测能够在显微镜图像中高效定位并识别结核杆菌,实现秒级推理与高召回率,从而显著提升检测效率与稳定性。同时引入 DeepSeek 等大模型能力,可在数据预处理、特征表达、模型优化等环节提供更强的语义理解与智能分析,使系统在小样本、复杂背景和噪声干扰条件下仍具备鲁棒性。PyQt5 作为桌面端图形界面框架,为临床人员提供直观、易操作的可视化界面,使算法能力真正转化为可用的应用工具。三者结合不仅提高了检测过程的自动化程度,也使 AI 辅助诊断在基层环境中具有可部署、可扩展、易使用的优势。该系统的研究对推动结核病筛查智能化、缩短诊断周期、降低医务人员负担、提升资源受限地区的公共卫生能力具有重要意义,同时为显微图像智能识别领域提供了可复用的技术路径与系统方案。
1.数据准备
本系统附带1265张结核杆菌检测测试集、验证集,大家可以根据自己的情况自行训练数据自己的数据集!
我们为模型训练准备了几百张结核杆菌图像,其中大多数为网络图片,然后使用VOC的格式存储数据标注文件,单数据标注文件内容如下:
bash
<annotation>
<source>
<database>Makerere Automated Lab Diagnostics Database</database>
<annotation>Makerere University</annotation>
<image>Mulago National Referral Hospital</image>
</source>
<segmented>0</segmented>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>1448</xmin>
<ymin>1124</ymin>
<xmax>1507</xmax>
<ymax>1217</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>1111</xmin>
<ymin>794</ymin>
<xmax>1159</xmax>
<ymax>849</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>135</xmin>
<ymin>1078</ymin>
<xmax>230</xmax>
<ymax>1140</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>228</xmin>
<ymin>1074</ymin>
<xmax>323</xmax>
<ymax>1145</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>69</xmin>
<ymin>575</ymin>
<xmax>172</xmax>
<ymax>643</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>79</xmin>
<ymin>686</ymin>
<xmax>180</xmax>
<ymax>732</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>87</xmin>
<ymin>385</ymin>
<xmax>178</xmax>
<ymax>482</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>106</xmin>
<ymin>354</ymin>
<xmax>261</xmax>
<ymax>393</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>789</xmin>
<ymin>11</ymin>
<xmax>901</xmax>
<ymax>61</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>1328</xmin>
<ymin>404</ymin>
<xmax>1429</xmax>
<ymax>447</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>1311</xmin>
<ymin>600</ymin>
<xmax>1392</xmax>
<ymax>651</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>1002</xmin>
<ymin>1128</ymin>
<xmax>1119</xmax>
<ymax>1173</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>28</xmin>
<ymin>750</ymin>
<xmax>73</xmax>
<ymax>853</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>775</xmin>
<ymin>451</ymin>
<xmax>826</xmax>
<ymax>503</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>651</xmin>
<ymin>598</ymin>
<xmax>760</xmax>
<ymax>645</ymax>
</bndbox>
</object>
<object>
<label>TBbacillus</label>
<pose>Unspecified</pose>
<truncated>0</truncated>
<occluded>0</occluded>
<bndbox>
<xmin>771</xmin>
<ymin>536</ymin>
<xmax>839</xmax>
<ymax>653</ymax>
</bndbox>
</object>
</annotation>2.数据集处理
1.数据集拆分
我们的原始数据集文件包括图像文件和标注文件,两者混合在同一个目录下,这就需要我们手动拆分数据集,目的是将图像和标注文件分开,便于后面的数据划分。
这段脚本用于清理与归类数据集目录中的文件。它会扫描指定根目录下的所有项目,将常见图片格式(JPG、JPEG、PNG)自动移动到新建的 images 文件夹,将 XML 格式的标注文件移动到 annotations 文件夹,其余非文件项会被忽略。整个流程确保目录结构更加规范、清晰,方便后续训练或数据处理。
大家首先执行step1_dataset_split.py
python
import os
import shutil
# 原始数据集目录
root = r"E:\thunder_download\useful\tuberculosis-phonecamera"
# 输出目录
img_dir = os.path.join(root, "images")
ann_dir = os.path.join(root, "annotations")
os.makedirs(img_dir, exist_ok=True)
os.makedirs(ann_dir, exist_ok=True)
# 图片扩展名
image_exts = {".jpg", ".jpeg", ".png"}
for filename in os.listdir(root):
src = os.path.join(root, filename)
if not os.path.isfile(src):
continue
ext = os.path.splitext(filename)[1].lower()
# 移动图片
if ext in image_exts:
shutil.move(src, os.path.join(img_dir, filename))
print("Moved image:", filename)
# 移动标注
elif ext == ".xml":
shutil.move(src, os.path.join(ann_dir, filename))
print("Moved annotation:", filename)
print("\n整理完成!")下图为拆分后的数据集图像文件
下图为数据集标注文件,格式均为xml
2.数据集标注文件类型转换
直接使用VOC格式的数据标注文件进行训练是不行的,需要我们将xml转成txt文件,
这段脚本实现了从 VOC 风格的 XML 标注批量生成 YOLO 所需的 txt 标签文件。程序逐一读取 XML 文件,匹配对应的图像以获取宽高,再提取每个目标的坐标信息,并按 YOLO 格式将框中心点和宽高归一化。所有类别根据预设映射转换为 class id,最终在指定目录下生成同名的 txt 标签,为后续训练 YOLO 模型做好数据准备。
执行下面脚本:step2_yolo_to_txt.py
bash
import os
import xml.etree.ElementTree as ET
from PIL import Image
# 原始路径
xml_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\annotations"
img_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\images"
# 新建一个目录用于保存YOLO格式txt
yolo_txt_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\labels"
os.makedirs(yolo_txt_dir, exist_ok=True)
# 类别映射
class_map = {"TBbacillus": 0}
# 遍历XML文件
for xml_file in os.listdir(xml_dir):
if not xml_file.endswith(".xml"):
continue
xml_path = os.path.join(xml_dir, xml_file)
tree = ET.parse(xml_path)
root = tree.getroot()
# 获取图像大小
img_filename = root.find("filename")
if img_filename is not None:
img_path = os.path.join(img_dir, img_filename.text)
else:
# 如果 XML 中没有 filename,使用同名jpg
img_path = os.path.join(img_dir, xml_file.replace(".xml", ".jpg"))
if not os.path.exists(img_path):
print(f"Warning: {img_path} 不存在,跳过")
continue
with Image.open(img_path) as img:
w, h = img.size
txt_lines = []
for obj in root.findall("object"):
label = obj.find("label").text
if label not in class_map:
continue
class_id = class_map[label]
bndbox = obj.find("bndbox")
xmin = float(bndbox.find("xmin").text)
ymin = float(bndbox.find("ymin").text)
xmax = float(bndbox.find("xmax").text)
ymax = float(bndbox.find("ymax").text)
# 转换成YOLO格式(归一化)
x_center = ((xmin + xmax) / 2) / w
y_center = ((ymin + ymax) / 2) / h
box_width = (xmax - xmin) / w
box_height = (ymax - ymin) / h
txt_lines.append(f"{class_id} {x_center:.6f} {y_center:.6f} {box_width:.6f} {box_height:.6f}")
txt_file_path = os.path.join(yolo_txt_dir, xml_file.replace(".xml", ".txt"))
with open(txt_file_path, "w") as f:
f.write("\n".join(txt_lines))
print(f"转换完成,YOLO TXT 文件保存在: {yolo_txt_dir}")拆分好之后,朋友们就能拿到可以给YOLO进行训练的YOLO格式数据集标注文件了,格式为TXT。
3.数据集拆分
YOLO 推荐训练集和测试集按 8:2 划分,主要是因为目标检测对样本量非常依赖,需要尽可能多的训练数据来学习特征,同时又必须保留足够的独立测试数据来评估模型的真实泛化能力。8:2 被证明在"训练数据够多"与"测试评估足够稳定"之间取得了较好平衡,因此成为默认且通用的实践比例。
从原始图像与标签目录中随机挑选 200 张图片,并按 8:2 的比例划分为训练集和验证集,然后将对应的图片和标签文件复制到新的数据集结构中。如果某张图片缺少同名的标签 .txt 文件,就为它创建一个空的标签文件,保证数据集结构完整。最终会在目标目录下生成:

执行脚本step3_auto_part.py
bash
import os
import random
import shutil
# 原始数据路径
img_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\images"
label_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\labels"
# 新数据集路径
dataset_dir = r"E:\thunder_download\useful\tuberculosis-phonecamera\dataset"
train_img_dir = os.path.join(dataset_dir, "train", "images")
train_label_dir = os.path.join(dataset_dir, "train", "labels")
val_img_dir = os.path.join(dataset_dir, "val", "images")
val_label_dir = os.path.join(dataset_dir, "val", "labels")
# 创建目录
for dir_path in [train_img_dir, train_label_dir, val_img_dir, val_label_dir]:
os.makedirs(dir_path, exist_ok=True)
# 获取所有图片文件
all_images = [f for f in os.listdir(img_dir) if f.lower().endswith((".jpg", ".png", ".jpeg"))]
# 随机抽取200张
if len(all_images) < 200:
raise ValueError(f"图片数量不足200张,当前数量: {len(all_images)}")
selected_images = random.sample(all_images, 200)
# 分割训练集和验证集
random.shuffle(selected_images)
val_images = selected_images[:40]
train_images = selected_images[40:]
def copy_files(image_list, target_img_dir, target_label_dir):
for img_file in image_list:
# 复制图片
src_img_path = os.path.join(img_dir, img_file)
dst_img_path = os.path.join(target_img_dir, img_file)
shutil.copy(src_img_path, dst_img_path)
# 对应的txt
label_file = os.path.splitext(img_file)[0] + ".txt"
src_label_path = os.path.join(label_dir, label_file)
if os.path.exists(src_label_path):
dst_label_path = os.path.join(target_label_dir, label_file)
shutil.copy(src_label_path, dst_label_path)
else:
# 如果没有对应txt文件,创建一个空文件
open(os.path.join(target_label_dir, label_file), "w").close()
# 复制训练集
copy_files(train_images, train_img_dir, train_label_dir)
# 复制验证集
copy_files(val_images, val_img_dir, val_label_dir)
print(f"随机抽取完成!训练集: {len(train_images)} 张,验证集: {len(val_images)} 张")
print(f"数据集路径: {dataset_dir}")3.模型训练
数据集准备好之后就可以开始模型训练了,我们首先准备一个训练的配置文件,比如说是data.yaml
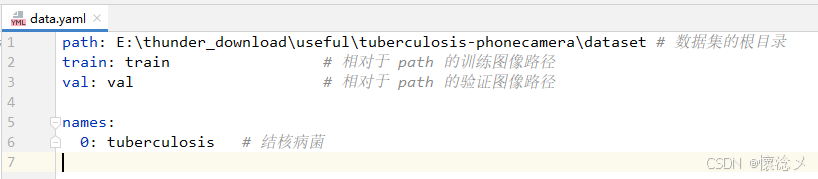
然后就可以开始模型训练了,直接执行我们准备好的train.bat文件,内容就是下面的内容
bash
yolo task=detect mode=train model=../data/model/yolov8n.pt data=./data.yaml epochs=50 imgsz=640 batch=16 lr0=0.01然后模型就开始训练了
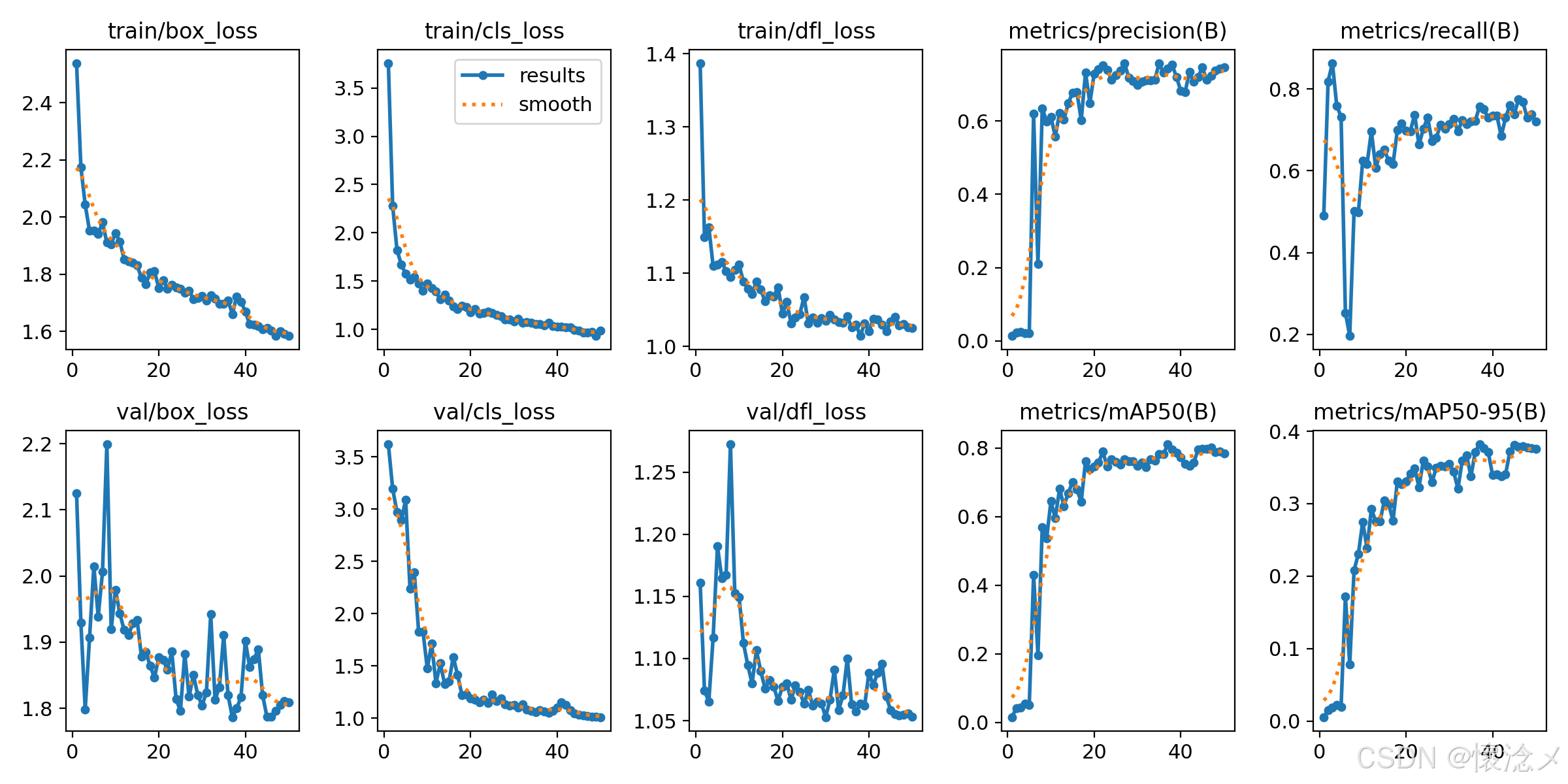
这里我贴一些训练验证结果截图


最后的results.png见下图,训练效果还是可以的!
我们的检测系统可以用在的应用场景:
1、在基层医疗机构中,用于辅助肺结核筛查,帮助医生快速判断痰涂片或培养图像中是否存在结核杆菌,提高诊断效率。
2、在疾控中心或防疫单位中,用于大规模样本检测,通过批量图像识别减少人工显微镜观察的工作量,降低漏检率。
3、在医院检验科,用作实验人员的智能辅助工具,对初筛结果进行自动预判,提升检测一致性,减少人为差异。
4、在偏远地区或医疗资源不足的场景,为移动检测车、便携式显微镜或远程医疗平台提供自动化图像分析能力。
5、在教学和科研机构中,用于结核杆菌图像数据的处理、可视化和模型研究,帮助学生和研究人员观察样本特征。
6、在实验室质量控制流程中,用于对比人工读片结果,提升检测质量管理水平,作为第二读片者降低误判风险。
7、在智慧医疗系统中,可与医院信息系统、远程会诊平台整合,实现病例图像上传后自动识别并反馈给医生,提高整体诊疗流程效率。
五.项目运行环境
本项目名称为yolov8-tb-detection
1.项目依赖
博主是在Windows电脑上使用Python3.8开发的本系统,建议大家使用的Python版本别太高。
其中项目依赖为:
bash
PyQt5==5.15.11
QtAwesome==1.3.1
torch==2.4.1
torchvision==0.19.1
Pillow==9.3.0
pyqtgraph===0.13.3
PyQtWebEngine==5.15.5
opencv-python==4.10.0.82
ultralytics==8.3.234
Requests==2.32.5
pandas==2.0.3
numpy==1.24.4
Markdown==3.4.4我已经整理到了requirements.txt,大家直接使用命令
pip install -r requirements.txt
即可一键安装项目依赖,其中的torch和torchvision只要匹配即可,不一定非要和博主开发环境的版本一致。
2.项目结构
很多小伙伴担心拿到代码后项目看不懂,这个大家不必担心,我们采用文件+类名对相关功能进行了模块化定义,大家见名知意。
下图博主采用tree命令生成了文件、目录树
bash
tree "D:\projects\gitee\2025\yolov8-tb-detection" /f /a
bash
D:\PROJECTS\GITEE\2025\yolov8-solar-defect
| .gitignore
| main.py(程序入口)
| record.txt(开发记录)
| requirements.txt(项目依赖)
|
|
+---script
| create_qrc.py
|
+---src(源代码核心目录)
| +---conf(配置内容)
| | | icon_conf.py
| | | style_conf.py
| | | system_conf.py
| | | test_data.py
| | | __init__.py
| |
| +---engine(核心引擎)
| | engines.py
| | __init__.py
| |
| +---resource(资源目录)
| | | resource.qrc
| | | resource_rc.py
| | | __init__.py
| | |
| | \---imgs
| | ai.svg
| | bg.jpg
| |
| +---threads(线程、信号总线)
| | main_threads.py
| | signal_bus.py
| | __init__.py
| |
| +---utils(工具方法、工具类)
| | custom_utils.py
| | user_manager.py
| | __init__.py
| |
| \---widgets(组件目录)
| base_widgets.py
| custom_pages.py
| custom_widgets.py
| main_page.py
| unique_widgets.py
| __init__.py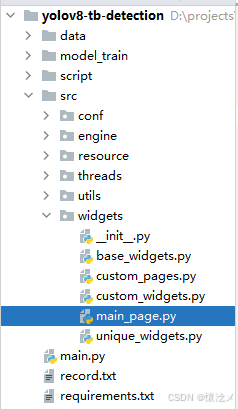
六.总结
本次给大家介绍了我使用PyQt5+YOLOv8+DeepSeek开发的结核杆菌检测系统,本系统功能强大,支持多种数据源输入,包含多种用户交互按钮以及模式,内置数据可视化方案、大模型AI加持,是您学习、工作使用的不错选择!
需要代码的朋友可以点击箭头下方的二维码加我好友,欢迎您了解!