Co-Seg: Mutual Prompt-Guided Collaborative Learning for Tissue and Nuclei Segmentation
2025年9月发表在arXiv上
Co-Seg:互提示引导的组织与细胞核分割协同学习
一个通过 "两个任务互相提供引导信息" 实现 "组织分割" 与 "细胞核分割" 双向增强的协同优化框架
协同分割(Collaborative Segmentation):不孤立处理两个分割任务,而是让它们 "组队协作"
- 传统方法要么用两个独立网络分别做组织分割和细胞核分割,要么共用编码器但解码器分离,本质还是 "各做各的"
- 两个完全独立的网络:训练时:网络 A 只拿 "组织分割标注数据" 训练,只优化 "组织分割的损失";网络 B 只拿 "细胞核分割标注数据" 训练,只优化 "细胞核分割的损失"。两者的参数、训练过程、学习到的知识完全独立,没有任何交集。预测时:输入一张病理图,网络 A 输出组织分割结果,网络 B 输出细胞核分割结果,两者的输出互不影响
- 共用编码器,但解码器分离:编码器:把原始病理图转换成通用的视觉特征;解码器:把编码器提取的通用特征,转换成具体任务的分割结果。训练时:两个任务共用同一个编码器提取通用特征,但后续的解码器是分开的;组织分割解码器只拿通用特征 + 组织标注数据训练,优化自己的组织分割损失;细胞核分割解码器只拿通用特征 + 细胞核标注数据训练,优化自己的细胞核分割损失。预测时:编码器提取的通用特征分别传给两个解码器,各自独立生成结果
- Co-Seg 的 "协同" 是深度交互:两个任务共享特征、互相提供引导、联合优化,最终实现 "1+1>2" 的效果,这是框架的核心创新
互提示引导:任务间双向提供 "引导信息"
提示"(Prompt) :不是用户手动输入的交互指令(如点击、框选),而是模型从自身任务结果中自动提取的 "引导信息",比如组织分割生成的 "组织区域特征"、细胞核分割生成的 "细胞核分布特征",这些特征能为另一个任务提供 "先验约束"。比如 "这片区域是肿瘤组织,细胞核大概率更密集"
双向引导,而非单向。组织分割的结果(如组织边界、区域类型)会作为 "提示",帮助细胞核分割更精准定位;细胞核分割的结果(如细胞核密度、分布)会作为 "提示",帮助组织分割更清晰区分组织亚型
这个 "互提示" 通过两个模块实现:
- RP-Encoder(区域感知提示编码器):从两个任务的掩码特征中提取高质量 "提示信息";
- MP-Decoder(相互提示掩码解码器):通过交叉注意力机制,让两个任务的 "提示" 互相作用,优化最终分割结果。
框架要同时完成两个具体任务:
- 组织语义分割(Tissue Semantic Segmentation):把病理图像中的 "组织区域" 按类型划分(比如肿瘤组织、正常组织、间质组织等),核心是 "划分区域、识别组织类型"
- 细胞核实例分割(Nuclei Instance Segmentation):把每个细胞核单独分离出来(区分 "哪个细胞核是哪个"),核心是 "分离个体、定位单个细胞核"
核心是:把两个任务的优化绑定在一起,让它们在训练中互相促进。
摘要
组织病理学图像分析至关重要,但该领域面临一项关键挑战:为实现肿瘤微环境与细胞形态学分析,需同时完成组织区域分割与细胞核实例分割。现有研究多单独聚焦于组织语义分割或细胞核实例分割,却忽略了这两项任务间的内在关联,导致对组织病理学图像的理解不够充分。为解决这一问题,本文提出一种名为 Co-Seg 的协同组织与细胞核分割框架。具体而言,我们引入一种新颖的协同分割范式,使组织分割与细胞核分割任务能够相互增强。为此,我们首先设计了区域感知提示编码器(RP-Encoder),以生成高质量的语义区域提示和实例区域提示,将其作为先验约束;此外,我们还构建了相互提示掩码解码器(MP-Decoder),该解码器利用交叉引导增强两项任务的上下文一致性,协同计算语义分割掩码与实例分割掩码。在 PUMA 数据集上开展的大量实验表明,所提出的 Co-Seg 框架在肿瘤组织与细胞核实例的语义分割、实例分割及全景分割任务中,均超越了现有最优方法。源代码已开源,地址为https://github.com/xq141839/Co-Seg。
理解1
组织病理学图像分析至关重要,但该领域面临一项关键挑战:为实现肿瘤微环境与细胞形态学分析,需同时完成组织区域分割与细胞核实例分割。
组织病理学图像(简称 "病理图")是医生通过显微镜拍摄的生物组织切片照片,像素分辨率通常很高(1024x1024),能清晰看到组织的细微结构
组织病理学图像分析是用计算机技术解读这些图像,提取关键信息。比如 "这片组织是不是肿瘤""肿瘤细胞的形态是否异常""肿瘤周围的环境有没有变化",最终为疾病诊断、肿瘤分级、治疗方案制定提供依据
病理分析的核心需求:肿瘤微环境与细胞形态学分析
- 肿瘤微环境分析:肿瘤不是孤立的 "癌细胞团",而是有自己的 "生态系统",这个 "生态系统" 的状态直接决定了肿瘤的生长速度、转移风险和治疗效果。所以分析 "肿瘤微环境" 就是搞清楚这个 "生态系统" 的结构和状态。
- 细胞形态学分析:重点看单个细胞的 "长相",比如细胞核的大小、形状、染色深浅、是否出现异常分裂。正常细胞和癌细胞的形态差异很大,通过分析细胞形态,能判断细胞是否癌变、癌变的程度
两个前提分割任务:"组织区域分割" 和 "细胞核实例分割"
- 组织区域分割:把图像中不同类型的组织区域明确划分开。比如分析肿瘤微环境时,首先要知道 "哪里是肿瘤组织,哪里是正常组织",才能进一步看两者的边界是否清晰、正常组织是否被侵犯;如果不分割,就没法区分 "肿瘤区域的血管" 和 "正常区域的血管",分析就会失真。
- 细胞核实例分割:把每个细胞核单独识别出来。如果不把每个细胞核分开,就没法测量单个核的大小、形状,也没法统计异常细胞核的比例;如果细胞核都连在一起,根本无法做精准的形态分析。
需同时完成:不是只做其中一个分割就行,而是两个都要做,且要做好,还要保证两者的一致性
比如:先通过 "组织区域分割" 确定 "这片是肿瘤组织区",再在这个区域内通过 "细胞核实例分割" 提取所有细胞核,分析它们的形态 ;如果组织区域分割错了(把正常组织当成了肿瘤组织),后续的细胞核形态分析就全错了;反之,如果细胞核分割漏检了很多异常细胞核,也会导致对肿瘤微环境的判断失误。
理解2
为此,我们首先设计了区域感知提示编码器(RP-Encoder),以生成高质量的语义区域提示和实例区域提示,将其作为先验约束;
区域感知提示编码器(RP-Encoder) :从两个任务的目标区域中,提取出精准、有效的 "引导信息"(即 "语义区域提示" 和 "实例区域提示"),并把这些信息作为 "提前定好的参考标准 / 约束条件",指导后续分割结果的计算,避免分割跑偏。
RP-Encoder的运作逻辑:
- 输入:模型第一阶段生成的 "初步分割掩码日志
- 处理过程:通过卷积、自注意力、与共享图像特征的交叉注意力,过滤掉草稿中的噪声,强化关键信息
- 输出:两个 "高质量提示"------g₁(语义区域提示,对应组织分割)和g₂(实例区域提示,对应细胞核分割)
提示:区域感知:只从任务的 "目标区域" 提取信息,不涉及无关背景:
- 语义区域提示(对应组织分割):比如 "这片区域是肿瘤组织(语义类型),边界在这(空间范围),和正常组织的交界在哪(结构特征)"。
- 实例区域提示(对应细胞核分割): 比如 "这些像素是单个细胞核(实例身份),细胞核的密度分布(比如肿瘤区域细胞核更密集)、形态特征(比如癌细胞核更大)"。
- "漏检"或 "误检"。
先验约束:模型分割时必须遵守的 "参考规则"
RP-Encoder从两个任务的初步分割结果中,提炼出针对 "组织区域属性" 和 "细胞核个体属性" 的精准引导信息(两个提示),并将这些信息作为 "不可违背的参考标准",确保后续分割过程中,两个任务能 "对齐核心特征、互相校准",而不是各自乱判。
理解3
此外,我们还构建了相互提示掩码解码器(MP-Decoder),该解码器利用交叉引导增强两项任务的上下文一致性,协同计算语义分割掩码与实例分割掩码。
为了让组织分割与细胞核分割真正实现 "双向协作",论文进一步设计了 "相互提示掩码解码器(MP-Decoder)",它的核心作用是让两个任务 "互相参考对方的关键信息"(交叉引导),确保两者的分割结果不矛盾、逻辑一致(上下文一致性),最终一起算出精准的组织语义分割掩码和细胞核实例分割掩码(协同计算)。
传统方法的解码器是 "各管各的"(比如组织解码器只处理组织特征,细胞核解码器只处理细胞核特征),而 MP-Decoder 的本质是:一个能让两个任务 "实时互动" 的统一解码框架。
基础结构:
- 包含两个 "任务头":组织分割头(负责输出组织掩码)和细胞核分割头(负责输出细胞核掩码);
- 存储两个任务的 "解码信息":用两组查询嵌入(q₁ 对应组织,q₂ 对应细胞核)记录各自的分割进度和关键特征;
- 核心能力:能让两个任务头 "互相读取对方的提示信息",并基于对方的信息修正自己的分割结果。
交叉引导:两个任务 "互相参考对方的关键信息"
本质是 "任务 A 用任务 B 的提示优化自己,任务 B 用任务 A 的提示优化自己",具体流程:
- 两个任务头先对自己的查询嵌入(q₁/q₂)做 "自注意力"
- 再互相交换参考:组织分割头用细胞核的提示(g₂,来自 RP-Encoder)做 "交叉注意力";同理,细胞核分割头用组织的提示(g₁)做 "交叉注意力"
- 通过 "元素相加" 和 "MLP(多层感知机)",把对方的参考信息融入自己的解码过程,让自身的特征更精准。
上下文一致性:保证两个任务的结果 "不矛盾、逻辑自洽"
两个任务的分割结果必须符合病理逻辑,不能出现 "矛盾":
- 比如:组织分割判定某区域是 "正常组织"(上下文),那么细胞核分割在该区域的结果就必须是 "细胞核形态规则、密度适中"(一致性),不能出现 "正常组织里全是异常形态的细胞核";
- 再比如:细胞核分割检测到某区域 "细胞核密集且形态异常"(上下文),组织分割就必须把该区域判定为 "肿瘤组织"(一致性),不能仍标为 "正常组织"。
协同计算:两个任务同时利用对方的信息,同步优化,最终一起输出结果:
- 交叉引导后,MP-Decoder 会通过 "反向交叉注意力",生成两个任务的 "专属特征"(h₁ 对应组织,h₂ 对应细胞核)
- 再通过 "像素解码器"(参考 SAM 的设计)对特征进行上采样(把低分辨率特征恢复到原图大小);
- 最后,通过 "特征与查询的点积",同时输出组织语义分割掩码(y₁)和细胞核实例分割掩码(y₂)。
理解4
在 PUMA 数据集上开展的大量实验表明,所提出的 Co-Seg 框架在肿瘤组织与细胞核实例的语义分割、实例分割及全景分割任务中,均超越了现有最优方法。
PUMA 数据集:黑色素瘤专用病理数据集,含 206 张 1024×1024 分辨率的组织病理学图像;训练 / 验证 / 测试集比例为 7:1:2;专门用于 "组织语义分割" 与 "细胞核实例分割" 任务
关键词
协同学习(Collaborative Learning)、组织分割(Tissue Segmentation)、细胞核分割(Nuclei Segmentation)、互提示(Mutual Prompt)
引言
医学图像分割在临床应用中发挥着至关重要的作用,已成为研究领域的热点方向 [7, 29, 10, 5]。具体而言,组织病理学图像分析面临着两项核心挑战:一是需勾勒出组织区域的轮廓,二是要进一步分离区域内的单个细胞核,以此实现组织亚型评估与肿瘤分级 [36]。这两项需求催生了该领域的两大关键分割任务,即组织语义分割与细胞核实例分割。
在医学语义分割领域,现有方法 [26, 35, 24, 34] 多依赖分层编码与解码结构,通过增强多尺度感知能力以生成精准的分割掩码。其中,基于 ViT [1] 和 Mamba [17] 的架构能够建模长距离依赖关系,从而获取目标区域的全局上下文信息。在实例分割方面,一系列研究 [6, 30, 2, 10] 采用了不同的距离代理图,以提升对实例边界的理解。近年来,基于 SAM(Segment Anything Model)的方法 [3, 21, 29, 5, 29, 4, 33, 16, 19] 通过手动提供对应任务提示,在医学语义分割与实例分割任务中展现出了令人瞩目的性能。
尽管相关研究已取得显著进展,但现有方法 [1, 31, 18, 32, 8] 通常仅聚焦于组织语义分割或细胞核实例分割的单独优化,仅能依赖孤立的知识进行监督训练。事实上,这两项任务高度相关,其核心目标均是实现对组织病理学图像的充分感知与深度理解。例如,精准识别细胞核能够为解析潜在的组织结构提供宝贵线索,而组织分割则可辅助细胞核的定位。这种强烈的相互依赖性促使我们开发一种协同方法,将组织分割与细胞核分割相结合,以推动组织病理学图像分析达到新的水平。
为突破这一瓶颈,本文提出了 Co-Seg 框架,一种用于组织与细胞核协同分割的方法,能够实现语义分割与实例分割任务的相互增强。如图 1 (c) 所示,Co-Seg 基于协同分割范式,通过捕捉两项任务间的上下文依赖关系,提升分割掩码的质量。具体而言,我们首先设计了区域感知提示编码器(RP-Encoder),从两项任务的目标区域中提取高质量的语义提示与实例提示,为分割解码过程提供引导;进一步引入相互提示掩码解码器(MP-Decoder),该解码器利用交叉引导机制,联合计算语义分割图与实例分割图。这种协同学习方法能够实现上下文一致性,减少两项任务中的预测误差。在黑色素瘤组织与细胞核分割任务上的实验结果表明,本文提出的 Co-Seg 框架性能显著优于现有的最优分割方法。
理解1
一是需勾勒出组织区域的轮廓,二是要进一步分离区域内的单个细胞核,以此实现组织亚型评估与肿瘤分级 [36]。
组织亚型评估:判断肿瘤的 "具体种类 / 亚型";肿瘤分级:判断肿瘤的 "恶性程度"
理解2
在医学语义分割领域,现有方法 [26, 35, 24, 34] 多依赖分层编码与解码结构,通过增强多尺度感知能力以生成精准的分割掩码。其中,基于 ViT [1] 和 Mamba [17] 的架构能够建模长距离依赖关系,从而获取目标区域的全局上下文信息。
医学语义分割的现有主流方法,靠 "分层提取特征 + 分层还原结果" 的结构,兼顾图像不同大小的细节(多尺度感知)来精准分割;而基于 ViT 和 Mamba 的模型,还能捕捉图像中远距离区域的关联(长距离依赖),拿到全局信息,让分割更全面、不局限于局部。
分层编码与解码结构:
- 编码(Encoder):把原始病理图像(比如 1024×1024 的像素图)层层压缩、提炼。从最表层的像素点,到中层的纹理、边缘,再到深层的 "这是肿瘤组织""这是正常组织" 的语义特征,一步步过滤冗余,留下有用的关键信息。
- 解码(Decoder):把编码器提炼的深层特征,层层放大、还原回和原图一样大的尺寸,同时补充细节,最终生成 "哪些像素属于某类组织" 的分割掩码
编码时从粗到细抓特征,解码时从细到粗补细节
多尺度感知能力:同时看清 "大区域" 和 "小细节"
ViT/Mamba 的长距离依赖建模:捕捉 "远距离区域的关联",拿到全局上下文
- 长距离依赖:指病理图像中 "离得远,但有逻辑关联" 的区域。比如某张图里,左上角和右下角都有分散的肿瘤细胞,这两个区域看似无关,但本质属于同一类组织,这种 "远距离的关联" 就是长距离依赖。
- 全局上下文信息:就是把整个图像的所有区域都纳入考虑,而不是只看某个局部。比如判断 "某块小区域是不是肿瘤" 时,不仅看它自身的特征,还看它和远处肿瘤组织的关联、和正常组织的分布关系
- ViT 和 Mamba 的作用:传统分层模型在捕捉这种远距离关联时能力较弱(比如只能顾及相邻区域),而 ViT(视觉 Transformer)靠 "自注意力机制"、Mamba 靠 "长序列建模能力",能高效跨越像素距离,把整个图像的关联信息整合起来
理解3
在实例分割方面,一系列研究 [6, 30, 2, 10] 采用了不同的距离代理图,以提升对实例边界的理解。近年来,基于 SAM(Segment Anything Model)的方法 [3, 21, 29, 5, 29, 4, 33, 16, 19] 通过手动提供对应任务提示,在医学语义分割与实例分割任务中展现出了令人瞩目的性能。
实例分割(比如细胞核单独分离)的现有研究,要么靠 "距离代理图" 帮模型看清目标边界,要么靠 "手动给提示" 的 SAM-based 方法聚焦目标
距离代理图:帮模型 "看清目标边界" 的辅助工具
- 实例分割的关键难点是 "区分相邻的同类目标"(比如病理图中挤在一起的细胞核,容易被模型当成一个整体)
- 距离代理图 不是直接输出分割结果,而是生成一张 "辅助图像",给每个像素标注 "关键距离信息",比如:
- 像素到最近细胞核中心的距离;
- 像素到相邻细胞核边界的距离;
- 像素属于哪个细胞核的 "归属概率距离"。
- 让模型通过这些距离信息,明确 "两个相邻目标的分界在哪里"。比如两个挨在一起的细胞核,距离代理图会显示 "中间的像素到两个中心的距离相等",模型就能判断这里是边界,从而把它们分开,避免合并成一个实例。
SAM-based 方法:靠 "手动提示" 聚焦目标的高效思路
通过手动提示,让 SAM 精准聚焦医学任务的目标(比如只分割组织内的细胞核、只识别特定类型的组织),避免无关区域干扰,从而在医学语义分割(组织分类)和实例分割(细胞核分离)中都取得了好效果。
理解4
组织病理学分析中,"组织分割" 和 "细胞核分割" 都是核心任务,领域内确实有研究尝试同时处理这两类任务,但有两个局限:
- 仅 "并行处理" 两个任务,未建立任务间的协同关联
- "同时做两个任务",但没有利用两者的内在关联(细胞核是组织的组成单元,组织是细胞核的存在背景)实现 "1+1>2" 的增强,本质还是 "各做各的"。
- "提示传递"(如细胞核的形态特征无法指导组织亚型的判断)。
- 聚焦单一任务的延伸,未将两者作为平等的协同对象
- "主从关系",而非 "对等协同关系",无法实现论文强调的 "双向互促"(组织分割帮细胞核定位,细胞核分割帮组织辨型)。
- HoverNet、CellPose)会在分割细胞核的同时,输出简单的 "组织区域掩码"(如区分 "有细胞区域" 和 "无细胞区域"),但这种组织掩码是 "粗糙的背景过滤工具",并非精准的 "组织语义分割"(无法区分肿瘤 / 正常 / 间质组织),也无法反过来从组织类型中获取细胞核分割的约束。
图一

图 1 本文 Co-Seg 与现有方法在组织与细胞核分割任务中的对比
(a) 用于组织与细胞核分割的两个独立网络;(b) 共享图像编码器但任务解码器分离的组织与细胞核分割网络;(c) 本文提出的 Co-Seg 框架,利用互提示实现组织与细胞核的协同分割。
术语:
- Sem.(Semantic):对应 "组织语义分割"
- Ins.(Instance):对应 "细胞核实例分割"
- Enc.(Encoder) ;Dec.(Decoder)
- Map:最终的分割结果图;
- Prompts:任务间的 "互提示信息"。
对比:
- 独立分割(Independent Segmentation):完全 "各做各的"
- 组织分割(上半部分):单独用 "Sem. Enc.(语义编码器)+ Sem. Dec.(语义解码器)",输出组织分割结果(Sem. Map);
- 细胞核分割(下半部分):单独用 "Ins. Enc.(实例编码器)+ Ins. Dec.(实例解码器)",输出细胞核分割结果(Ins. Map)。
- 两个任务无任何共享 / 交互------ 既不共享图像特征,也不传递任务信息,相当于 "两个完全无关的模型分别干活",完全忽略了组织和细胞核的内在关联。
- 共享编码器分割(Encoder-shared Segmentation):"共享基础特征,但任务独立"
- 通用编码器提取图像的基础特征,同时传给 "Sem. Dec.(语义解码器)" 和 "Ins. Dec.(实例解码器)";
- 两个解码器各自独立生成组织分割结果(Sem. Map)和细胞核分割结果(Ins. Map)。
- 仅共享了 "基础图像特征",但任务解码器之间无任何交互------ 组织分割的结果不会影响细胞核分割,反之亦然,本质还是 "两个任务并行执行",没利用任务间的关联。
- Co-Seg(本文方法):"互提示引导的协同分割"
- Forward 1 阶段:共享编码器提取特征后,先通过 Sem. Dec. 和 Ins. Dec. 生成 "初步分割结果";
- 互提示生成阶段:从初步结果中提取 "组织语义提示" 和 "细胞核实例提示"(中间的 Prompts);
- Forward 2 阶段:把这些互提示传递给两个解码器,让它们 "参考对方的任务信息",最终生成更精准的 Sem. Map 和 Ins. Map。
(c)图
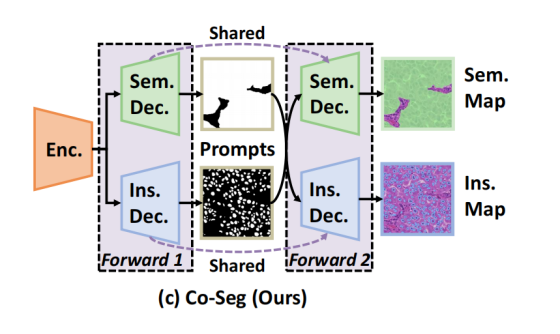
流程:
- 先通过Forward 1得到初步的组织 / 细胞核分割结果,从中提取 "提示(Prompts)" 信息。
- Forward 1阶段,Sem. Dec.(组织分割解码器)和Ins. Dec.(细胞核分割解码器)会先输出各自的初步分割结果,这些结果被提炼成 "提示(Prompts)"(比如组织的大致区域、细胞核的初步分布),暂存在中间的Prompts区域。
- 这些 "提示" 会跨任务传递(比如细胞核的分布提示给组织分割,组织的边界提示给细胞核分割)。
- Sem. Dec.的黑色箭头:把Ins. Dec.在 Forward 1 生成的初步细胞核分布提示 ,传给组织分割解码器,帮助它更精准地识别组织边界;指向Ins. Dec.的黑色箭头:把Sem. Dec.在 Forward 1 生成的初步组织边界提示,传给细胞核分割解码器,帮助它区分密集细胞核的个体边界
- 再通过Forward 2,两个解码器结合对方的 "提示",最终输出更精准的分割图。
Forward 1 和 Forward 2 阶段用到的Sem. Dec.(组织分割解码器)是同一个模块(参数完全复用),Ins. Dec.(细胞核分割解码器)也是同一个模块(参数完全复用),不是单独训练两个不同的解码器,这样能减少模型参数、同时保证前后两次前向过程的一致性。
方法
如图2所述,我们设计了基于协同分割范式的Co-Seg框架,实现了医学影像中组织分割与细胞核分割的相互优化 。为实现这一目标,我们设计了RP-Encoder ,为两个任务提供引导性的先验约束;同时设计了MP-Decoder ,通过双向信息交互协同生成语义图与实例图。这种一体化方法使得两个任务能够相互增强,借助交互式提示提升分割精度。
理解1
RP-Encoder ( Region-aware Prompt Encoder - 区域感知提示编码器 )
MP-Decoder ( Mutual-Prompt Mask Decoder - 互提示掩码解码器 )
图二
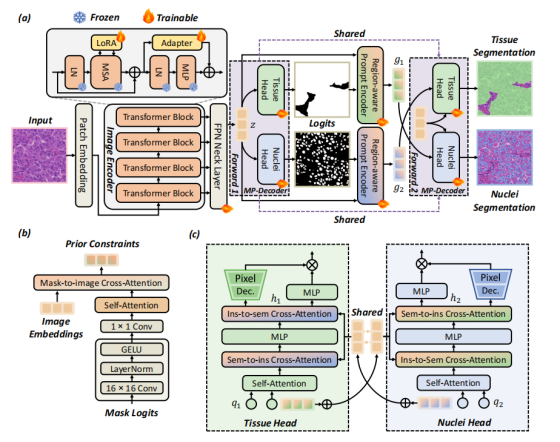
图2. (a)所提出的Co-Seg框架概览,用于协同进行组织与细胞核分割,该框架包括(b)RP-Encoder与(c)MP-Decoder。Co-Seg通过利用交互式提示,充分挖掘了互补信息。
协同分割范式
Co-Segmentation Paradigm
现有的组织分割方法 [9,13,22] 和细胞核分割方法 [23,20,3] 均将语义分割与实例分割的参数空间解耦,破坏了两者间的内在依赖关系,导致性能下降。为解决这一问题,本文提出协同分割范式:通过闭环双向交互实现双任务协同优化。具体而言,本文的目标是训练一个模型

,其中
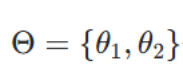
分别代表语义分割和实例分割的参数,y1和y2则为对应任务的分割掩码。
为建模双任务间的依赖关系,本文将y1和y2的联合预测定义为:
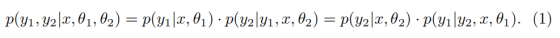
需注意,式(1)通过相互交织的条件概率,揭示了语义分割与实例分割间的双向依赖关系。该式表明:在给定图像和模型参数的前提下,获取两种分割结果的联合概率
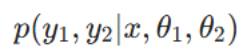
可通过两种对称方式分解:

若已知语义分割结果y1,则其会直接影响实例分割结果y2的预测;
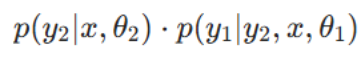
若已知实例分割结果y2,则其会影响语义分割结果y1的输出。
上述两种表达式在数学上等价,二者从不同视角描述同一联合概率,凸显了任一分割任务均可提供关键上下文信息,从而提升另一任务的精度。这种双向依赖关系表明,我们需要一种协同方法:将两个分割任务结合优化,而非孤立训练,充分利用各任务的上下文信息以提升整体分割效果。
将该依赖关系转化为两个参数的梯度计算,可得:
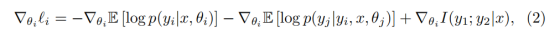

基于上述梯度,可得参数更新规则如下:
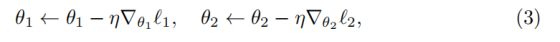

通过这种方式,协同分割范式打破了多任务学习中 "各任务梯度流孤立" 的壁垒,使 Co-Seg 框架能够实现组织分割与细胞核分割的双向优化。
理解1
现有的组织分割方法 [9,13,22] 和细胞核分割方法 [23,20,3] 均将语义分割与实例分割的参数空间解耦,破坏了两者间的内在依赖关系,导致性能下降。
在医学图像分析(比如分析组织切片)中,通常需要做两件事:
- 语义分割 :把图像中的每个像素分类为不同的组织类型(例如,癌变区域、正常区域、背景)。这回答的是"这是什么"。
- 实例分割 :把图像中每一个独立的细胞核轮廓都精确地勾勒出来,并区分开。这回答的是"这个东西在哪里,有多少个"。
过去的做法 :大多数模型把这两个任务完全分开处理,用两套独立的参数(模型大脑)来学习。这忽略了一个关键点:这两个任务的信息是相互依赖、相互促进的。
- 知道"这是什么"(组织类型),能帮你更好地"数出有多少个"(细胞核)。例如,在肿瘤区域,细胞核可能更密集、形态更不规则。
- 知道"有多少个、在哪里"(细胞核分布),也能帮你更准确地判断"这是什么"(组织类型)。例如,细胞核大量聚集的区域很可能是病变区。
理解2
为解决这一问题,本文提出协同分割范式:通过闭环双向交互实现双任务协同优化。具体而言,本文的目标是训练一个模型
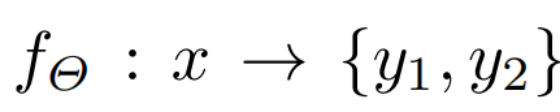
,其中
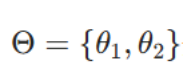
分别代表语义分割和实例分割的参数,y1和y2则为对应任务的分割掩码。
本文提出的新方法,就是让这两个任务协同工作、互相学习,形成一个闭环。
核心思想 :训练一个 模型,但同时输出两个结果(组织分割图 y1 和细胞核分割图 y2)。关键在于,模型内部的两部分参数(θ1负责语义分割,θ2负责实例分割)不是孤立的,它们通过信息流紧密连接。
理解3
为建模双任务间的依赖关系,本文将y1和y2的联合预测定义为:
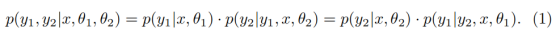
需注意,式(1)通过相互交织的条件概率,揭示了语义分割与实例分割间的双向依赖关系。该式表明:在给定图像和模型参数的前提下,获取两种分割结果的联合概率
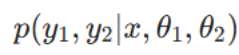
可通过两种对称方式分解:
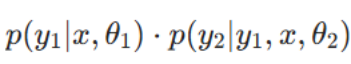
若已知语义分割结果y1,则其会直接影响实例分割结果y2的预测;
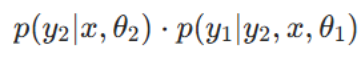
若已知实例分割结果y2,则其会影响语义分割结果y1的输出。
上述两种表达式在数学上等价,二者从不同视角描述同一联合概率,凸显了任一分割任务均可提供关键上下文信息,从而提升另一任务的精度。这种双向依赖关系表明,我们需要一种协同方法:将两个分割任务结合优化,而非孤立训练,充分利用各任务的上下文信息以提升整体分割效果。
如何实现协同?
p(y1, y2 | ...) = p(y1|...) · p(y2|y1, ...) = p(y2|...) · p(y1|y2, ...)
是这个思想在数学上的表达。它用"概率"来描述这种依赖关系,意思是:
- 第一种分解方式 (p(y1|...) · p(y2|y1, ...)): 要得到最终的两个结果,可以先预测组织分割 (y1),然后在已知组织分割结果的前提下,再去预测细胞核分割 (y2)。这样,组织信息就指导了细胞核的识别。
- 第二种分解方式 (p(y2|...) · p(y1|y2, ...)): 也可以先预测细胞核分割 (y2),然后在已知细胞核分布的前提下,再去优化组织分割 (y1)。这样,细胞核的细节信息又反过来帮助了组织分类。
这完美体现了任务间的"双向依赖":任何一方的输出,都可以作为另一方输入的宝贵"上下文信息"。
理解4
将该依赖关系转化为两个参数的梯度计算,可得:


如何训练?
∇θᵢ ℓᵢ
- ∇:梯度算子
- θᵢ:任务i的模型参数(θ₁为语义分割参数,θ₂为实例分割参数)
- ℓᵢ:任务i的总体损失函数(待优化的目标)
- ∇θᵢ ℓᵢ :损失函数ℓᵢ关于参数θᵢ的梯度。这就是反向传播中用来更新参数的梯度向量。
-∇θᵢ ��[log p(yᵢ|x, θᵢ)]
- - :负号,表示最小化(因为默认是最大化似然,加负号转为最小化损失)
- ∇θᵢ:对θᵢ求梯度
- ��[...] :期望(Expectation),在实际训练中用批量样本的平均值近似
- log:自然对数
- p(yᵢ|x, θᵢ) :条件概率,表示在给定输入x和参数θᵢ时,输出yᵢ的概率
- 整体含义 :任务i自身的基本损失梯度。这就是传统单任务训练时用的梯度。
-∇θᵢ ��[log p(yⱼ|yᵢ, x, θⱼ)]
- 结构类似第一项,但条件概率不同
- p(yⱼ|yᵢ, x, θⱼ) :条件概率,表示在给定x、yᵢ和θⱼ时,输出yⱼ的概率
- 关键区别 :这里的条件包含了yᵢ(任务i的输出)
- 整体含义 :任务i的参数θᵢ对任务j的性能影响梯度。它回答:"如果我调整θᵢ改变了yᵢ,这对任务j的预测yⱼ有什么影响?"
+∇θᵢ I(y₁; y₂|x)
- + :正号,表示最大化
- I(y₁; y₂|x):条件互信息(Conditional Mutual Information)
- 定义:I(y₁; y₂|x) = ��[log(p(y₁, y₂|x) / (p(y₁|x)·p(y₂|x)))]
- 物理意义 :衡量在已知x的情况下,y₁和y₂之间共享的信息量
- *∇θᵢ I(...)****:****最大化互信息关于θᵢ的梯度**
当更新任务i的参数θᵢ时,梯度来自三个部分:
- 第一部分(主任务梯度):让任务i自身更准确,例如:让组织分割的边界更清晰
- 第二部分(条件任务梯度) :让任务i的输出能更好地帮助任务j,例如:调整组织分割的结果,使其能更好地指导细胞核分割。让两个任务互相成为对方的训练信号
- 第三部分(互信息梯度) :让两个任务的输出共享更多信息,例如:促使组织分割结果和细胞核分割结果在特征层面更一致
∇θ₁ ℓ₁ = -∇θ₁ ��[log p(y₁|x, θ₁)] // 让组织分割更准确
- ∇θ₁ ��[log p(y₂|y₁, x, θ₂)] // 让组织分割结果能更好地帮助细胞核分割
- ∇θ₁ I(y₁; y₂|x) // 让组织分割和细胞核分割的结果更相关
对公式(1)取负对数后,得到:
-log p(y₁, y₂|x, θ₁, θ₂) = -log p(y₁|x,θ₁) - log p(y₂|y₁,x,θ₂)
如果我们希望最大化联合概率p(y₁, y₂|x)(即让两个任务的预测都准确且一致),那么我们就需要同时最小化这两个负对数项。
最大似然估计 :找到一组参数θ,使得模型预测出真实标签y的可能性最大。
- 我们希望 p(y|x,θ) → 越大越好
- 等价于 log p(y|x,θ) → 越大越好
- 等价于 -log p(y|x,θ) → 越小越好
-∇θᵢ ��[log p(yᵢ|x, θᵢ)]
- p(yᵢ|x, θᵢ):任务i的似然(我们希望它大)
- log p(...):对数似然(我们希望它大)
- -log p(...) :负对数似然(我们希望它小)← 这就是损失项
- ∇θᵢ ��[-log p(...)]:损失对参数的梯度(用于减小损失)
- 公式中前面的负号:因为 ∇[-log p(...)] = -∇[log p(...)],所以写成 -∇��[log p(...)]
为了让模型在训练时就能学会这种"互相帮助",设计了特殊的优化(学习)规则。
目标:更新参数 θ1 和 θ2,让模型的预测结果越来越好。
- 传统方法:θ1 只根据组织分割的误差来更新,θ2只根据细胞核分割的误差来更新。两者没有直接交流。
- 本文方法 :在计算更新 θ1 的梯度时,不仅考虑组织分割本身的误差(公式(2)第一项),还额外加入一项 ,这项反映了"当前的组织预测结果,对细胞核分割任务有多大帮助"(公式(2)第二项)。反之,更新 θ2 时也同样考虑对组织分割任务的帮助。
- 互信息项 (I(y1; y2|x)):这是为了确保两个任务学到的特征不是随机的,而是高度相关的、共享的 。它促使模型去挖掘那些既能解释组织类别、又能解释细胞核形态的底层共同特征。它不直接指导"该怎么做",而是设定一个终极优化目标 :让组织分割结果 y1 和细胞核分割结果 y2 之间的内在相关性最大化。
在训练时,最大化互信息 I(y1; y2|x) 会产生一种强大的"拉力":
- 它会惩罚"各学各的"行为:如果 θ1 学到的特征只对预测组织类型有效,但与 θ2 预测细胞核的特征毫无关系,那么 y1 和 y2 的互信息就会很低,模型会收到一个很大的"惩罚信号"(高损失)。
- 它会奖励"共享特征" :模型会发现,只有去寻找那些同时 影响组织类别 和 细胞核形态的底层图像属性(如纹理、密度、染色强度、边界形态等),才能让 y1 和 y2 的输出自然变得高度相关,从而降低惩罚,达成优化目标。
神经网络中:
- 期望�� → 用小批量(mini-batch)的平均计算
- 概率p(...) → 通过网络的输出层(通常是softmax/sigmoid)获得
- 梯度∇ → 通过****自动微分(PyTorch/TensorFlow的反向传播)****计算
- 互信息项 → 需要估计联合概率和边缘概率
理解5
基于上述梯度,可得参数更新规则如下:
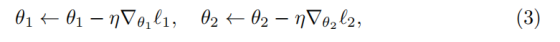

通过这种方式,协同分割范式打破了多任务学习中 "各任务梯度流孤立" 的壁垒,使 Co-Seg 框架能够实现组织分割与细胞核分割的双向优化。
- ←:赋值操作(更新参数)
- η:学习率(learning rate)
- ∇θᵢ ℓᵢ:就是公式(2)计算出的协同梯度
- 整体 :标准的随机梯度下降 更新规则,但关键是梯度本身包含了跨任务协同信息
区域感知提示编码器
Region-aware Prompt Encoder, RP-Encoder
所提出的协同分割范式需要在组织分割与细胞核分割任务之间建立隐式依赖关系。为实现这一目标,我们引入了区域感知提示编码器(RP-Encoder):该编码器利用掩码提示捕捉任务特定的目标区域,并将其作为先验约束,如图 2 (b) 所示。
具体而言,我们首先采用多个卷积块,从掩码日志mui中提取特征mi。之后,RP-Encoder 先执行自注意力操作,再与共享图像嵌入z进行交叉注意力操作,生成一组密集提示gi,具体过程如下:

其中,Conv1表示用于下采样的 16×16 卷积,Conv2表示用于通道扩展的 1×1 卷积,(LN(·))代表层归一化(LayerNorm)。这些操作会对掩码日志的结构上下文进行编码。在此基础上,我们将 RP-Encoder 应用于语义掩码日志与实例掩码日志,有效得到区域感知提示g1(组织分割提示)和g2(细胞核分割提示),并将其作为 Co-Seg 框架中组织分割与细胞核分割任务的先验约束。
理解1
区域感知提示编码器
- 区域感知 :它能"理解"输入掩码所描绘的特定区域(比如一片肿瘤组织,或一群细胞核)的上下文信息。
- 提示 :它的输出不是最终答案,而是一种指导性信号,用于影响或"提示"另一个任务的模型。
- 编码器:它是一个神经网络模块,负责将输入的信息(这里是粗糙掩码)编码成更高级、更紧凑的特征表示。
如何工作?
步骤1:输入------粗糙的"掩码日志" (μᵢ)
- 它不是完美的最终分割图,而是模型在当前步骤预测出的、比较粗糙的初步分割结果。
- μ₁ :来自组织分割分支的语义分割草图(标注了不同组织类型的区域)。
- μ₂ :来自细胞核分割分支的实例分割草图(标注了各个细胞核的轮廓)。
步骤2:特征提取与精炼 (公式4)
目标:把粗糙的草图转换成更结构化、更富含信息的特征图 mᵢ
过程:
- Conv₁:用于下采样,把 μᵢ的特征图尺寸缩小(比如从 1024×1024 缩到 64×64),既减少计算量,又能保留 "目标区域的宏观结构"
- LN, GELU:进行层归一化和激活,稳定特征的数值分布(比如让特征值集中在 - 1~1 之间);给特征引入 "非线性",让模型能捕捉复杂的区域模式,避免特征只能表达简单的线性关系。稳定训练并引入非线性
- Conv₂:用于通道扩展,把特征的 "信息维度" 增加,让 mᵢ能承载更多细节,为后续注意力操作提供足够的 "信息容量"
- 结果:得到了精炼后的区域特征 mᵢ,它比原始草图 μᵢ 更能代表该区域的本质信息。
步骤3:生成"智能提示" (公式5)
目标:给提纯的特征 mᵢ 加'全局视野',让提示既懂 "自身结构",又懂 "在整个图像中的位置和语境"
过程:
- 自注意力 ( SelfAttention(mᵢ) )
- 作用:捕捉掩码内部的结构依赖
- 目的:强化区域内各部分之间的关系。例如,在组织分割提示中,让属于同一肿瘤区域的不同部分相互确认;在细胞核提示中,让相邻的细胞核相互关联。
- 结果 :得到了自洽、上下文关联更强的区域特征。
- 交叉注意力 ( CrossAttention(..., z, z) )
- 输入 :第一步自注意力后的区域特征,以及从原始图像提取的共享图像嵌入 z(z 是图像最本质的特征表示)。
- 作用:让 mᵢ"对接全局图像信息",交叉注意力能把 mᵢ的局部特征和 z 的全局特征结合起来
- 如果只看局部特征,可能把 "正常组织的细胞核" 误判为 "肿瘤组织的细胞核",交叉注意力让提示 "不脱离全局语境"
- 结果:密集提示gi(g1是组织提示,g2是细胞核提示)------ 此时的gi是 "既有局部结构细节,又有全局图像语境" 的精准提示
理解2
所提出的协同分割范式需要在组织分割与细胞核分割任务之间建立隐式依赖关系。
- 没有 RP-Encoder 时,两个任务的初步结果是 "孤立、粗糙" 的,无法直接互相参考;
- 有了 RP-Encoder 后,初步结果被转化为 "有结构、有语境、可复用" 的提示 g₁和 g₂,让后续 MP-Decoder 能实现 "组织提示引导细胞核分割,细胞核提示引导组织分割",真正建立起两个任务的隐式依赖
- 显式依赖:手动设定规则 "若某区域细胞核密度> 阈值,则该区域为肿瘤组织",模型直接执行该规则建立关联;
- 隐式依赖:不设定任何手动规则,让模型通过学习大量病理图像数据,自动发现 "细胞核的形态 / 密度" 与 "组织类型" 的内在关联 ------ 这种关联不会以 "规则" 形式存在,而是藏在特征向量的数值变化、注意力权重的分配中。
RP-Encoder 建立任务间依赖的核心逻辑是 "数据驱动的特征交互",而非 "人工定义的显式约束",具体体现在 3 点:
- 依赖关系不是 "手动设定",而是 "数据训练习得"
RP-Encoder 的目标是 "捕捉任务特定的目标区域作为先验约束",但它从未手动定义 "组织和细胞核该如何关联"------ 比如没有规定 "哪些细胞核特征对应肿瘤组织""哪些组织区域必须包含细胞核"。
相反,这种关联是模型通过训练自动学到的:
- 训练时,模型接触大量病理图像数据(如 PUMA 数据集),其中组织类型(如肿瘤 / 正常组织)与细胞核特性(如形态、密度、分布)天然存在统计关联;
- RP-Encoder 通过掩码提示(μ₁、μ₂)和注意力机制,将这种 "数据中天然存在但未被标注的关联",自动编码到提示 g₁(组织提示)和 g₂(细胞核提示)的特征向量中;
- 比如模型可能学到 "某类紧凑排列的细胞核特征(蕴含在 g₂中)" 与 "黑色素瘤组织特征(蕴含在 g₁中)" 高度相关,但这种关联是模型从数据中总结的,而非人类提前写好的规则。
- 依赖关系不是 "直观外露",而是 "蕴含在特征 / 参数中"
RP-Encoder 建立的依赖关系,不会以 "可解读的形式" 呈现 ------ 你无法直接从 g₁或 g₂的特征向量中,看出 "组织和细胞核的依赖是什么"(比如无法像看规则一样读到 "细胞核密度高→组织为肿瘤")。
这种依赖是 "藏在细节里" 的:
- 它体现为注意力权重的分配(比如 g₂的细胞核特征在与 g₁交互时,某类特征的注意力权重更高);
- 体现为特征向量的数值模式(比如肿瘤组织的 g₁向量,与对应区域细胞核的 g₂向量在高维空间中距离更近);
- 这些模式只有模型能 "理解" 并用于协作分割,人类无法直接解读为明确的关联规则 ------ 这就是 "隐式" 的核心:依赖关系存在,但不直观、不外露。
- 依赖关系通过 "特征交互" 自动传递,而非 "显式通道映射"
RP-Encoder 与 MP-Decoder 的协作中,任务间的依赖传递是通过 "交叉注意力" 实现的,而非 "手动指定的特征通道映射":
- 比如 MP-Decoder 中,组织分割任务的查询 q₁通过 CrossAttention 与 z⊕g₂(细胞核提示 + 全局特征)交互,此时细胞核对组织的约束,是通过高维特征的加权融合自动实现的,而非 "将 g₂的某几个通道直接映射到 q₁的特定通道";
- 这种传递过程没有明确的 "依赖路径"(比如 "g₂的第 10 维特征对应 g₁的第 5 维特征"),而是由模型根据训练数据动态调整 ------ 依赖的传递方式也是隐式的。
互提示掩码解码器
Mutual Prompt Mask Decoder, MP-Decoder
遵循协同分割范式,我们提出了相互提示掩码解码器,以强制组织分割与细胞核分割之间的双向交互,确保它们相互精炼而不是被当作孤立任务处理,如图2(c)所示。
具体而言,MP-Decoder包含组织和细胞核头,它们利用两组查询嵌入:q₁ 和 q₂ 来保存两个任务的解码信息。为了利用任务间的互补效应,我们首先对每个查询执行自注意力操作,然后与另一个任务的提示进行交叉注意力操作,如下所示:
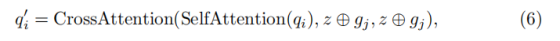
其中 ⊕ 表示逐元素相加。随后应用MLP以确保精炼后的查询嵌入具有表现力。MP-Decoder进一步应用反向交叉注意力来生成任务特定的图像嵌入 h₁ 和 h₂,如下所示:

在此基础上,细胞核的上下文提示可提升组织分割的一致性,而组织的边界反馈则为细胞核的区分提供先验约束。参考标准 SAM [15] 的设计,每个解码头均采用像素解码器(Pixel Decoder)对优化后的任务专属图像嵌入进行上采样。最终,MP-Decoder 通过计算上采样后的图像嵌入与对应任务查询之间的点积(dot product),生成组织分割与细胞核分割的预测结果。
综上,所提出的 MP-Decoder 通过充分利用组织分割与细胞核分割信息的双向交互,实现了 Co-Seg 框架分割掩码质量的相互提升。
理解1
MP-Decoder包含组织和细胞核头,它们利用两组查询嵌入:q₁ 和 q₂ 来保存两个任务的解码信息。为了利用任务间的互补效应,我们首先对每个查询执行自注意力操作,然后与另一个任务的提示进行交叉注意力操作,如下所示:
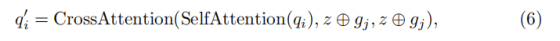
其中 ⊕ 表示逐元素相加。随后应用MLP以确保精炼后的查询嵌入具有表现力。
- 组织头 / 细胞核头:MP-Decoder 的两个 "专属解码器"------ 组织头负责输出组织分割结果,细胞核头负责输出细胞核分割结果,各自有独立的解码逻辑,但需要互相协作。
- 查询嵌入 q₁/q₂:相当于两个任务的 "解码草稿本"------
- q₁:组织头的查询嵌入,存储 "组织分割的解码信息"(比如 "哪里可能是肿瘤组织""边界在哪里" 的初步线索);
- q₂:细胞核头的查询嵌入,存储 "细胞核分割的解码信息"(比如 "哪里有细胞核""哪些属于同一个细胞核" 的初步线索)。
- 它们不是最终结果,是解码过程中可优化的 "中间信息载体"。
- 另一个任务的提示:就是 RP-Encoder 生成的 "精准导航图"------
- 对组织头(用 q₁解码)来说,"另一个任务的提示" 是 g₂(细胞核提示,包含细胞核的位置、密度等信息);
- 对细胞核头(用 q₂解码)来说,"另一个任务的提示" 是 g₁(组织提示,包含组织的范围、类型等信息)。
MP-Decoder 中每个查询嵌入(q₁或 q₂)的优化流程,本质是 "先让自己的解码信息更聚焦,再借鉴对方任务的有用线索",分两步走:
第一步:对每个查询执行 "自注意力" 操作
操作对象:单独对 q₁(组织查询)或 q₂(细胞核查询)执行自注意力,不涉及另一个任务。
具体作用:提纯自身的解码信息,让 "草稿本" 更清晰、更聚焦核心目标。
- 对 q₁(组织分割):自注意力会梳理 q₁中存储的 "组织分割线索",比如 "强化肿瘤组织的核心区域特征""弱化背景噪声的干扰",明确 "我要分割的组织重点在哪里";
- 对 q₂(细胞核分割):自注意力会梳理 q₂中存储的 "细胞核分割线索",比如 "区分单个细胞核和集群""突出细胞核的边界特征",明确 "我要分割的细胞核重点在哪里"。
第二步:与 "另一个任务的提示" 做 "交叉注意力" 操作
操作对象:经过自注意力优化后的查询,和 "另一个任务的提示",再加上共享图像嵌入 z

是把全局图像特征 z 和对方任务的提示"按元素相加",相当于 "把对方的提示融入全局语境",避免借鉴的信息脱离图像背景。
具体作用:让当前任务的解码信息,借鉴另一个任务的 "精准线索",弥补自身不足。
交叉注意力的本质是 "用一个任务的查询(Q),去匹配另一个信息源的键(K)和值(V)",其固定输入格式为:CrossAttention(Q, K, V)
- Q(Query):当前任务的 "待优化查询"(这里是经过自注意力提纯后的SelfAttention(q_i));
- K(Key):用于 "匹配查询" 的特征(需与 Q 的维度一致,提供 "检索线索");
- V(Value):用于 "补充查询" 的特征(与 K 一一对应,提供 "有用信息")。
在绝大多数场景中,为了让 "检索线索" 和 "补充信息" 完全对齐,会直接让K=V
虽然K=V=z⊕g_j,但二者在 CrossAttention 中承担不同角色,共同支撑跨任务协作:
- 第一个z ⊕ g_j(作为 K):负责 "与 Q 匹配"------ 告诉当前任务的查询(Q)"哪里有有用的跨任务信息"。比如组织分割的 Q(SelfAttention(q₁))要找 "能优化边界的线索",K(z⊕g₂)会通过特征匹配,定位到 "细胞核密集的区域"(这些区域的组织边界更需要收紧);
- 第二个z ⊕ g_j(作为 V):负责 "给 Q 补充信息"------ 在 K 找到匹配位置后,将 "全局语境 + 跨任务线索" 的融合信息传递给 Q。比如上述匹配到 "细胞核密集区域" 后,V(z⊕g₂)会把 "该区域是肿瘤组织内的细胞核集群" 的信息补充给 Q,让 Q 调整组织分割的边界。
理解2
MP-Decoder进一步应用反向交叉注意力来生成任务特定的图像嵌入 h₁ 和 h₂,如下所示:
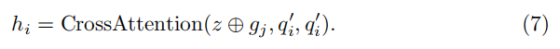
注意力方向(Q→K/V):
- 当前任务查询(qᵢ)→ 对方提示 + 全局特征(z⊕gⱼ):让查询 "主动借鉴" 对方的互补线索,"我(当前任务)去学你的好东西"
- 对方提示 + 全局特征(z⊕gⱼ)→ 当前任务优化后查询(qᵢ'):让对方线索 "被动适配" 当前任务需求,"把你的好东西按我的需求加工,变成我能用的专属资源"
理解3
在此基础上,细胞核的上下文提示可提升组织分割的一致性,而组织的边界反馈则为细胞核的区分提供先验约束。参考标准 SAM [15] 的设计,每个解码头均采用像素解码器(Pixel Decoder)对优化后的任务专属图像嵌入进行上采样。最终,MP-Decoder 通过计算上采样后的图像嵌入与对应任务查询之间的点积(dot product),生成组织分割与细胞核分割的预测结果。
综上,所提出的 MP-Decoder 通过充分利用组织分割与细胞核分割信息的双向交互,实现了 Co-Seg 框架分割掩码质量的相互提升。
参考 SAM 的设计,每个解码头(组织头、细胞核头)都有 "像素解码器",核心作用是上采样:
- 之前生成的任务专属图像嵌入(h₁、h₂)是 "低分辨率特征图"(比如 64×64),而原始病理图像是高分辨率(比如 1024×1024)------ 如果直接用低分辨率特征图输出结果,没法对应原图的每个像素,分割结果会模糊、粗糙。
- 像素解码器的工作:把 h₁(组织专属嵌入)、h₂(细胞核专属嵌入)从低分辨率 "放大" 到和原始图像一样的尺寸,同时保留优化后的精准特征(比如组织的边界细节、细胞核的位置信息)。
上采样后的图像嵌入(高分辨率、优化后),和对应任务的查询做 "点积",就能输出最终的分割掩码,具体逻辑:
输入:
- 上采样后的图像嵌入(比如 h₁上采样后):高分辨率特征图,每个像素都包含 "该位置是否属于组织目标" 的精准特征(比如 "属于肿瘤组织的概率线索");
- 对应任务的查询:组织分割的 "核心需求载体",包含 "我要分割肿瘤组织、明确边界" 的目标信息(相当于模型的 "任务指令")。
点积的作用:计算 "匹配度",输出概率
- 点积是一种数学运算,本质是 "计算两个向量的相似性"------ 在这里,就是计算 "上采样嵌入中每个像素的特征" 和 "查询的任务指令" 的匹配度。
- 匹配度高→该像素属于 "目标区域"(比如肿瘤组织、细胞核)的概率高;
- 匹配度低→该像素属于 "背景区域" 的概率高。
- 最终,点积会输出一张和原图同尺寸的 "概率图",每个像素都有一个 "属于目标的概率值",再经过简单阈值判断(比如概率 > 0.5 判定为目标),就生成了最终的分割掩码(白色为目标,黑色为背景)。
优化流程
Optimization Pipeline
为构建 Co-Seg 框架,我们首先采用 Hiera ViT [27] 作为组织分割与细胞核分割的共享图像编码器,以确保特征学习的一致性。
具体而言,我们加载预训练的 SAM2-L [25] 模型来初始化相应参数,并冻结这些权重以保留预训练知识。此外,我们将 LoRA [12] 和 Adapter [11] 插入注意力层与前馈神经网络(FFN)层,实现从自然图像领域到病理图像领域的参数高效微调。
基于协同分割范式,Co-Seg 包含两次解码前向传播过程,且 MP-Decoder 的组织解码头与细胞核解码头在这两个阶段共享参数。具体流程如下:
- MP-Decoder 利用图像编码器生成的图像嵌入,在无需任何提示的情况下,计算出两个任务的二值分割掩码(y_1')(组织分割初步掩码)和(y_2')(细胞核分割初步掩码);
- 将这两个初步掩码传递至 RP-Encoder,生成组织提示(g_1)和细胞核提示(g_2);
- 在第二次前向传播中,这些提示将直接作为先验约束反馈给 MP-Decoder;
- 最终,MP-Decoder 通过交叉引导协同生成组织分割与细胞核分割的最终掩码。
Co-Seg 的训练过程包含两部分:
(1)组织提示(g_1)和细胞核提示(g_2)的先验约束优化;
(2)组织分割与细胞核分割的联合优化。
整体损失函数定义如下:
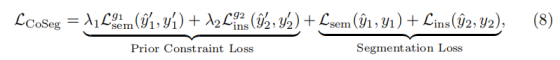

理解1
梯度运算(公式 2)是协同分割范式的理论原理 ,解释了 "组织 / 细胞核分割如何通过梯度更新实现双向依赖";
损失函数(公式 8)是该原理的工程落地,量化了 "协同优化的目标",将双向依赖转化为可最小化的误差指标
- 传统多任务梯度:仅考虑自身任务损失;
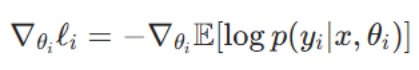
- Co-Seg 的梯度:在传统梯度基础上,新增了任务间依赖的隐式梯度 (第二项)和互信息梯度(第三项)。
将梯度运算的理论逻辑,转化为可计算、可优化的误差指标:
- 先验约束损失:对应梯度运算的 "任务间依赖隐式梯度",确保 RP-Encoder 生成的(g_1/g_2)(先验约束)足够精准,为双向协作提供可靠线索;
- 分割损失:对应梯度运算的 "单任务损失梯度",确保最终分割结果与真实标签一致,不偏离核心任务目标;
- 系数:调节先验约束的重要性,让模型在 "协作线索质量" 和 "最终分割精度" 之间找到平衡。
最终模型训练时,通过最小化(L_{CoSeg}),间接实现了公式 2 的梯度协同逻辑,既保证了任务间的双向依赖,又确保了分割结果的精准性。
理解2
第一次前向传播MP-Decoder 直接解码,生成初步二值掩码 y₁'(组织)、y₂'(细胞核),为 RP-Encoder 提供生成 "精准提示" 的原始素材
第二次前向传播MP-Decoder 通过交叉引导,协同解码,生成最终的组织 / 细胞核分割掩码
若没有第一次传播,RP-Encoder 就没有输入(掩码日志 μᵢ本质就是 y₁'/y₂' 的特征形式),无法生成 g₁/g₂,后续的双向协作就成了 "无的放矢"。
理解3
FPN Neck Layer 特征金字塔网络颈部层 ,整合编码器输出的多尺度特征图,生成兼具 "高分辨率细节" 和 "低分辨率全局语境" 的融合特征,为后续解码提供更全面的特征支撑。
中间枢纽,上接编码器,下接解码器,负责特征的 "提纯与融合"
"FPN(特征金字塔)":核心设计是构建多尺度特征金字塔,打破单一尺度特征的局限性 ------ 编码器输出的特征图通常是 "下采样逐步缩小" 的(比如 1024×1024→512×512→256×256),不同尺度特征各有价值:
- 高分辨率特征(如 512×512):包含细粒度细节(如细胞核的微小边界、组织的局部纹理);
- 低分辨率特征(如 256×256):包含全局语境(如组织的整体范围、肿瘤区域的宏观分布)。
实验
实验设置
为验证所提 Co-Seg 框架的有效性,我们采用黑色素瘤专用病理数据集 PUMA [28],用于组织语义分割和细胞核实例分割任务。该数据集包含 206 张分辨率为 1024×1024 的病理图像,训练集、验证集和测试集的划分比例为 7:1:2。
所有实验均在搭载 NVIDIA A5000 GPU 的设备上基于 PyTorch 框架完成。为保证对比的公平性,所有组织分割和细胞核分割方法均采用相同的训练设置与配置。对于基于医学 SAM 的基线模型,我们采用预训练的 SAM ViT-H [15] 结构作为图像编码器。
优化器选用 Adam,初始学习率设为 1×10⁻⁴,并采用指数衰减策略调整学习率,衰减系数为 0.98。批量大小(batch size)设为 16,训练轮次(epochs)设为 300。
组织分割任务采用交叉熵损失(cross-entropy loss)与 Dice 损失的组合进行监督;细胞核分割任务则遵循标准损失组合方式,采用 Focal 损失、Dice 损失、均方误差损失(MSE loss)和 MSGE 损失 [6,10]。损失系数 λ₁和 λ₂分别设为 2 和 1。
在微调过程中,相比基线模型,我们的 Co-Seg 框架新增了 9.1% 的可学习参数(见表 4)。
理解1
对于基于医学 SAM 的基线模型,我们采用预训练的 SAM ViT-H [15] 结构作为图像编码器。
让所有 "基于 SAM 的基线模型" 和 Co-Seg 的 "编码器基础" 一致 ------Co-Seg 的编码器是 Hiera ViT(加载了 SAM2-L 预训练参数),而对比的 SAM 类基线用 SAM ViT-H,避免 "因为编码器本身性能差异" 导致对比不公平。
理解2
采用指数衰减策略调整学习率,衰减系数为 0.98。
指数衰减策略调整学习率,本质是让模型训练时的学习率随训练轮次(epochs)逐渐缩小,衰减系数 0.98 意味着每训练一轮,当前学习率就变为上一轮的 98%
学习率的更新公式为「当前轮学习率 = 上一轮学习率 × 衰减系数(0.98)」,按指数规律逐步下降
- Co-Seg 的初始学习率是 1×10⁻⁴(0.0001),衰减系数 0.98,训练 300 轮。
- 第 1 轮学习率:1×10⁻⁴(初始值);
- 第 2 轮学习率:1×10⁻⁴ × 0.98 = 9.8×10⁻⁵;
- 第 10 轮学习率:1×10⁻⁴ × 0.98⁹ ≈ 8.3×10⁻⁵;
- 第 100 轮学习率:1×10⁻⁴ × 0.98⁹⁹ ≈ 1.3×10⁻⁵;
- 越往后,学习率缩小的幅度越平缓,但始终在缓慢下降。
理解3
组织分割任务采用交叉熵损失(cross-entropy loss)与 Dice 损失的组合进行监督;细胞核分割任务则遵循标准损失组合方式,采用 Focal 损失、Dice 损失、均方误差损失(MSE loss)和 MSGE 损失 [6,10]。损失系数 λ₁和 λ₂分别设为 2 和 1。
组织分割:交叉熵损失 + Dice 损失
交叉熵损失(Cross-Entropy Loss)
- 核心作用:擅长 "像素级分类",快速优化模型对 "目标(组织)/ 背景" 的区分能力。
- 优势:梯度计算稳定,训练初期能快速让模型学到 "哪里是组织、哪里是背景" 的基础规律,避免模型初期发散。
- 局限:对类别不平衡敏感 ------ 如果背景像素远多于组织像素,模型会偏向预测背景,导致组织区域漏检。
Dice 损失(Dice Loss)
- 核心作用:聚焦 "分割区域的重叠度",专门解决类别不平衡和边界模糊问题。
- 计算逻辑:通过衡量模型预测区域与真实组织区域的 "交并比"(重叠程度)计算误差,重叠度越低,损失越大。
- 优势:对少量组织区域更敏感 ------ 即使组织像素少,也会强制模型关注,避免漏检;同时能强化边界对齐,让组织分割的区域更精准。
细胞核分割:Focal 损失 + Dice 损失 + MSE 损失 + MSGE 损失
Focal 损失(Focal Loss)
- 核心作用:解决 "难易样本不平衡"------ 细胞核密集重叠时,部分细胞核(小核、重叠核)难检测(难样本),部分细胞核(大核、孤立核)易检测(易样本)。
- 优势:通过降低易样本的权重、提高难样本的权重,让模型重点学习难样本,避免 "只捡软柿子捏",减少小核漏检和重叠核误判。
Dice 损失(与组织分割一致)
核心作用:保证单个细胞核的 "区域重叠度",避免细胞核分割过大或过小,确保每个细胞核的范围精准。
均方误差损失(MSE Loss)
- 核心作用:优化细胞核的 "边界平滑度" 和 "像素值连续性"。
- 逻辑:计算预测掩码与真实掩码的像素值平方差,惩罚边界处的剧烈波动,让细胞核的边界更平滑,避免分割出 "锯齿状" 的不规则边界。
MSGE 损失(Mean Squared Gaussian Error Loss)
- 核心作用:针对细胞核的 "形态和分布" 优化 ------ 尤其适合密集细胞核的实例区分。
- 优势:通过高斯分布建模细胞核的形态特征(比如核的大小、圆度),让模型学习到 "正常细胞核的形态规律",减少将组织纹理误判为细胞核(假阳性),同时提升密集核的分离效果。
损失系数 λ₁=2、λ₂=1:调节 "先验约束损失" 的权重
这两个系数对应公式(8)中的 "先验约束损失"(λ₁Lg1^sem + λ₂Lg2^ins),核心是控制 "提示质量" 对模型训练的影响程度:
系数含义
- λ₁=2:组织提示 g₁的先验约束损失权重为 2------ 意味着模型训练时,会更重视组织提示的质量(g₁越精准,损失越小)。
- λ₂=1:细胞核提示 g₂的先验约束损失权重为 1------ 细胞核提示的质量同样重要,但权重略低于组织提示。
- 组织分割的 "区域范围" 是细胞核分割的基础(比如肿瘤组织内的细胞核需要重点区分),组织提示 g₁的质量对双向协作影响更大 ------ 给 λ₁更高权重,能强制 RP-Encoder 生成更精准的组织提示,为细胞核分割提供更可靠的约束。
- 系数平衡了 "组织提示" 和 "细胞核提示" 的重要性,避免某一方的提示质量不足导致协作失效。
与现有最优方法的性能对比
组织语义分割
我们首先评估了所有模型在组织语义分割任务中的性能。如表 1 所示,经典语义分割方法的性能劣于基于医学 SAM 的模型 ------ 例如,H-SAM [5] 的 Dice 系数提升 0.51%,超越了 EMCAD [24]。值得注意的是,我们的 Co-Seg 取得了最优性能,P 值小于 0.005,Dice 系数达 92.51%,且豪斯多夫距离(HD)为 206.72(最低值),表明其具备精准的组织边界定位能力。
表一
表 1 组织语义分割任务与现有最优方法的性能对比

细胞核实例分割
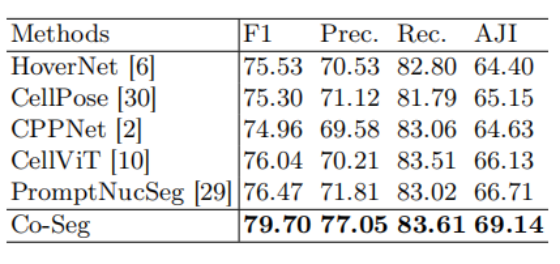
细胞核实例分割的对比结果如表 2 所示。我们的 Co-Seg 框架在四项评价指标中均展现出压倒性性能优势。具体而言,相较于次优方法 PromptNucSeg [29],Co-Seg 的性能提升显著:P 值小于 0.001,F1 分数提升 3.23%,调整后联合交集(AJI)提升 2.43%。
表二
表 2 细胞核实例分割任务与现有最优方法的性能对比
病理全景分割
我们进一步采用全景分割协议,对 Co-Seg 的综合性能进行全面评估。
图三
图 3 组织语义分割与细胞核实例分割三项评价协议的定性对比结果。得益于语义分割与实例分割任务的相互强化,我们的 Co-Seg 能够精准勾勒组织区域、分割出准确的细胞核,且假阳性更少。

表三
表 3 病理全景分割任务与现有最优方法的性能对比

如表 3 所示,Co-Seg 受益于所提出的新型协同分割范式,在全部六项指标中均取得最优性能:组织分割和细胞核分割的全景分割质量(PQ)分别显著提升 1.51% 和 2.74%。
消融实验
Ablation Study
为验证所提出的协同分割范式(C)、区域感知提示编码器(RP-Encoder,P)及互提示掩码解码器(MP-Decoder,D)的有效性,我们在 PUMA 数据集的组织语义分割与细胞核实例分割任务上开展了全面的消融实验,结果如表 4 所示。
通过从 Co-Seg 中移除定制模块,我们构建了两个独立的、基于 Hiera ViT 的语义分割和实例分割网络作为消融实验基线。引入 RP-Encoder 后,组织语义分割的 Dice 系数提升 0.25%,细胞核实例分割的 F1 分数提升 0.66%。
此外,我们探究了 RP-Encoder 与 MP-Decoder 组合使用的效果,结果显示性能显著提升:组织语义分割 Dice 系数达 91.76%,细胞核实例分割 F1 分数达 78.19%。
最后,我们采用所提出的协同分割范式构建完整框架。实验结果表明,该设计能显著提升语义分割和实例分割性能。综上,这些消融实验验证了 Co-Seg 框架中 RP-Encoder、MP-Decoder 及协同分割范式的有效性。
表四
表 4 Co-Seg 在 PUMA 数据集上的组织分割与细胞核分割消融实验
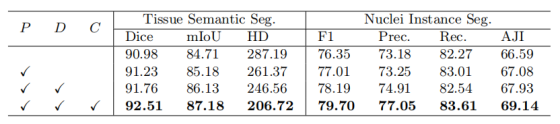
(P=RP-Encoder,D=MP-Decoder,C = 协同分割范式)
第 1 组(基线):无 P、无 D、无 C;
第 2 组:加入 P(仅 RP-Encoder):验证 "区域感知提示编码器" 的作用 ------ 是否能通过生成先验提示提升分割精度
基础解码器先用 z 生成 y₁'/y₂',再通过 P 生成 g1/g2,最后结合 z 和 g1/g2 解码,输出分割结果。
第 3 组:加入 P+D(RP-Encoder+MP-Decoder):验证 "提示编码器 + 互提示解码器" 的协同作用 ------ 是否能通过双向注意力交互进一步优化性能
共享编码器输出图像嵌入 z,输入 MP-Decoder(替代第二组的基础解码器),无提示盲解码生成 y₁'(组织初步掩码)、y₂'(细胞核初步掩码)
y₁'/y₂' 转化为掩码日志 μᵢ,输入 RP-Encoder(P),通过卷积、自注意力、交叉注意力(公式 4-5)生成 g1(组织提示)、g2(细胞核提示)
MP-Decoder 接收 z 和 g1/g2,通过 "自注意力→交叉注意力→反向交叉注意力" 实现双向交互
遵循 SAM 设计,通过像素解码器上采样 h1/h2,再与优化后的查询 q₁'/q₂' 做点积,生成最终分割掩码。
第 4 组(完整 Co-Seg):加入 P+D+C(RP-Encoder+MP-Decoder + 协同分割范式):验证 "协同分割范式" 的核心价值 ------ 是否能通过梯度层面的双向依赖,实现最终性能突破
- 生成初步掩码 y₁'/y₂'(同第三组,MP-Decoder 生成);
- 生成精准提示 g1/g2(同第三组,P 处理 μᵢ生成);
- 双向协作解码(同第三组,MP-Decoder 通过公式 6-7 生成 h1/h2,上采样后点积生成分割结果);
- 核心升级:梯度层面双向协同(新增 C 的作用):
- 计算整体损失 LCoSeg(公式 8),包含 "先验约束损失(优化 g1/g2 质量)+ 分割损失(优化最终结果)";
- 反向传播时,遵循公式(2)的梯度更新规则:
- 更新组织分割参数 θ₁时,梯度不仅包含组织自身损失,还纳入 "组织对细胞核的影响"(任务间依赖隐式梯度)和 "双任务共享特征质量"(互信息梯度);
- 更新细胞核分割参数 θ₂时同理,实现 θ₁和 θ₂的 "互相绑定优化"。
- 迭代训练:通过学习率指数衰减(0.98)优化参数,最终输出最优分割结果。
第三组仅在 "解码阶段" 通过特征交互协作,第四组在 "训练阶段" 通过梯度绑定让双任务参数优化 "互相促进"
结论
本研究明确了病理图像中组织分割与细胞核分割之间的潜在关联,提出了一种新型协同分割范式,并构建了 Co-Seg 框架,以实现细胞核分割与组织分割的相互促进。该框架包含两个核心模块:区域感知提示编码器(RP-Encoder)通过感知目标区域,生成任务专属的区域提示;互提示掩码解码器(MP-Decoder)采用交叉引导机制,协同生成组织分割与细胞核分割掩码。
在黑色素瘤组织与细胞核实例分割数据集上开展的大量实验表明,Co-Seg 框架显著优于现有方法,性能提升幅度明显。