文章目录
介绍
空间转录组学(ST)极大地改变了我们对组织结构的理解,但构建全面的三维(3D)细胞图谱仍面临技术限制和高昂成本的挑战。目前的方法通常只能捕获稀疏采样的二维切片,留下了大量空白区域,限制了我们对连续器官组织结构的理解。在此,我们提出了 SpatialZ,这是一个计算框架,通过在实验测量的切片之间生成虚拟切片来填补这些空白,从而能够从平面 ST 数据中构建密集的 3D 细胞图谱。SpatialZ 旨在以单细胞分辨率运行,并且不受特定空间技术固有的基因覆盖限制的影响。全面的验证表明,SpatialZ 能准确地保留细胞身份、基因表达模式和空间关系。利用 BRAIN 倡议细胞普查网络数据,我们构建了一个包含超过 3800 万个细胞的 3D 半球图谱。这套密集的地图集具备多种新功能,包括在任意角度进行虚拟切片、对三维体积和表面的基因表达进行探索、对查询组织切片进行三维绘制,以及通过新的合成视角发现三维空间中的分子结构。为了展示其在转录组学之外的扩展性,我们将 SpatialZ 应用于人类乳腺癌的成像质谱数据,成功解析了肿瘤微环境中三维空间的梯度。我们的方法生成了细胞图谱,提供了前所未有的三维空间分子景观分辨率。
空间转录组学(ST)已成为现代生物学中的一项变革性技术,它使研究人员能够绘制基因表达模式的同时保留组织内的关键空间信息1、2、3。通过同时获取转录组数据和空间背景信息,这些技术极大地改变了我们对组织结构、细胞相互作用以及基因调控网络在其自然环境中的理解4、5、6、7。
空间转录组学(ST)的最新进展主要集中在二维(2D)组织分析方面8、9、10、11、12、13,其中对单个平面组织切片进行处理,以生成包含转录组学和二维空间信息的细胞图谱4、14、15、16、17。尽管一些实验室试图利用厚组织块将这些能力扩展到三维分析中18、19、20,但目前的方法存在局限性。现有的三维 ST 技术仅限于相对较薄的组织切片(在最近的进展中可达 200 微米18),这使得器官级别的三维映射变得不切实际。这些方法复杂的实验流程和资源密集型的特点进一步限制了它们在更广泛的研究社区中的应用。
为应对这些挑战,研究人员开发了计算方法来将一系列二维切片在统一的三维坐标系中进行对齐21、22、23、24。这种方法使得能够整合大量的二维扫描技术数据集,包括各种器官细胞图谱、癌症研究以及由"大脑倡议细胞普查网络"(BICCN)生成的脑细胞图谱25。然而,这些方法只是朝着三维方向迈出的初步一步,因为它们实质上是创建了一组二维切片的堆叠,而非一个三维体积,从而在相邻切片之间留下了较大的间隔(例如 100 微米)未进行测量。这些间隔导致空间信息出现不连续性,并阻碍了对基因表达模式和器官级结构的全面三维分析。
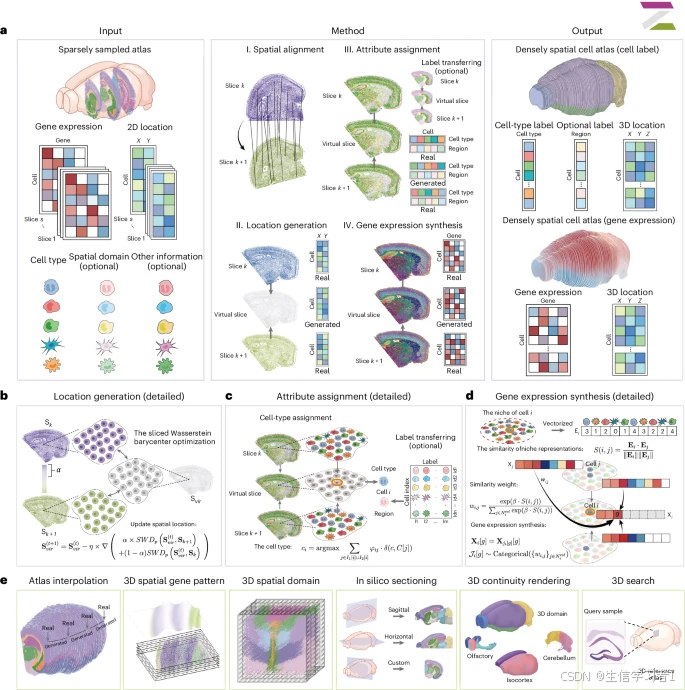
对于弥合二维和三维单细胞转录组(ST)之间的差距而言,理想的解决方案应满足以下关键要求:(1)生成涵盖器官大部分区域的全面三维器官图谱,(2)不受特定 ST 技术的基因覆盖限制的影响,(3)具备单细胞分辨率的能力,(4)在表面和体积上准确呈现三维基因表达模式,(5)识别三维组织区域和感兴趣的空间域,以及(6)能够进行计算机模拟切片以生成任何所需角度或厚度的额外二维切片。
在本研究中,我们引入了 SpatialZ 这一计算框架,它能够从稀疏采样的二维组织切片中构建出一个"伪三维图谱"。该框架借鉴了单细胞分析中的"伪时间"概念------即通过基因表达的连续性而非实际的时间测量来推断时间------我们的"伪三维图谱"基于不同切片之间生态位转变的连续性来重建三维组织结构。尽管当前的技术限制使得无法生成真实的实验性三维图谱,但 SpatialZ 通过在已测量的二维切片之间进行插值来填补这一空白,从而生成"虚拟切片",最终构建出一个密集且三维的空间细胞图谱。
代码
https://github.com/senlin-lin/SpatialZ/
参考
- Bridging the dimensional gap from planar spatial transcriptomics to 3D cell atlases
- https://github.com/senlin-lin/SpatialZ/