在实践中,使用诸如ARI之类的指标有一个很大的问题。在应用聚类算法时,通常没有真实值来比较结果。如果我们知道了数据的正确聚类,那么可以使用这一信息构建一个监督模型(比如分类器)。因此,使用类似ARI和NMI的指标通常仅有助于开发算法,但对评估应用是否成功没有帮助。
有一些聚类的评分指标不需要真实值,比如轮廓系数(silhouette coeffcient)。但它们在实践中的效果并不好。轮廓系数计算一个簇的紧致度,其值越大越好,最高分数为1。虽然紧致的簇很好,但紧致度不允许复杂的形状。
下面利用轮廓分数在two_moons数据集上比较k均值、凝聚聚类和DBSCAN:
python
import matplotlib.pyplot as plt
import mglearn
import numpy as np
from sklearn.metrics.cluster import adjusted_rand_score
from sklearn.datasets import make_moons,make_blobs
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
from sklearn.decomposition import NMF,PCA
from sklearn.cluster import AgglomerativeClustering
from sklearn.cluster import DBSCAN
from sklearn.metrics.cluster import silhouette_score
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
X,y=make_moons(n_samples=200,noise=0.05,random_state=0)
#将数据缩放成平均值为0、方差为1
scaler=StandardScaler()
scaler.fit(X)
X_scaler=scaler.transform(X)
fig,axes=plt.subplots(1,4,figsize=(15,3),subplot_kw={'xticks':(),'yticks':()})
#创建一个随机的簇分配,作为参考
random_state=np.random.RandomState(seed=0)
random_clusters=random_state.randint(low=0,high=2,size=len(X))
axes[0].scatter(X_scaler[:,0],X_scaler[:,1],c=random_clusters,cmap=mglearn.cm3,s=60)
axes[0].set_title('随机分配-轮廓系数:{:.2f}'.format(silhouette_score(X_scaler,random_clusters)))
#列出要用的算法
algorithms=[KMeans(n_clusters=2),AgglomerativeClustering(n_clusters=2),DBSCAN()]
for ax,algorithm in zip(axes[1:],algorithms):
clusters=algorithm.fit_predict(X_scaler)
ax.scatter(X_scaler[:,0],X_scaler[:,1],c=clusters,cmap=mglearn.cm3,s=60)
ax.set_title('{}:{:.2f}'.format(algorithm.__class__.__name__,silhouette_score(X,clusters)))
plt.show()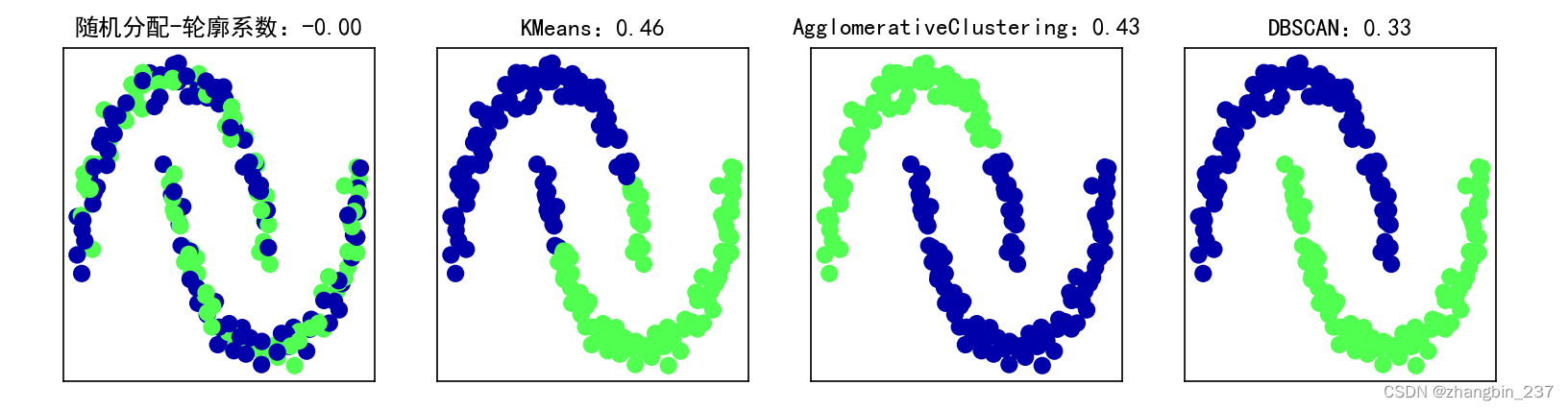
如上图,k均值的轮廓分数最高,尽管我们可能更喜欢DBSCAN的结果。但对于评估聚类,稍好的策略是使用基于鲁棒性的聚类指标。这种指标先向数据中添加一些噪声,或者使用不同的参数设定,然后运行算法,并对结果进行比较。其思想是:如果许多算法参数和许多数据扰动返回相同的结果,那么它是可信的。