
关键词:基准与方法研究;基因测序;变异检测;
文献简介
- 标题(英文):DNAscope: High accuracy small variant calling using machine learning
- 标题(中文) :DNAscope:使用
的机器学习高精度小变异调用 - 发表期刊:bioRxiv
- 作者单位:Sentieon公司
- 发表年份:2022
- 文章地址:https://doi.org/10.1101/2022.05.20.492556

图1 文献简介
当前的小变异检测技术,尤其是GATK的HaplotypeCaller,在大多数情况下表现优秀。然而,在复杂基因组区域的检测准确性仍有提升空间。随着测序技术在临床应用中的普及,提高这些区域的检测准确性变得越来越重要。传统方法主要依赖专家构建的模型和手动调整的过滤器,而机器学习方法显示出通过学习更复杂的变异特征关系来提高检测准确性的潜力。
测序流程
DNAscope作为GATK HaplotypeCaller的进阶版本,巧妙地融合了成熟的基于单倍型的变异检测方法和先进的机器学习技术,以提升变异检测的准确性。它在保留原有逻辑架构的同时,优化了活跃区域检测和局部组装过程,特别增强了在复杂基因组区域的表现。DNAscope通过为候选变异添加额外信息注释,并结合机器学习模型进行变异基因型分析,显著提高了整体准确度。此外,DNAscope还可与贝叶斯基因型分析模型配合使用,使其在非哺乳动物物种的重测序分析中同样发挥优势,体现了其广泛的适用性和卓越的性能。
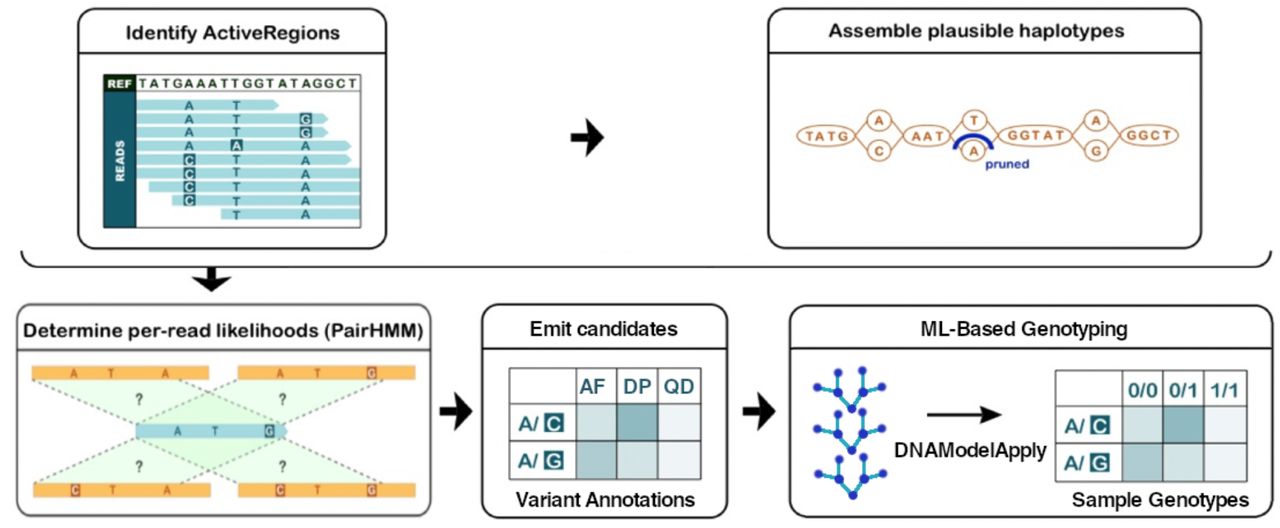
图2 DNAscope方法概述
为了评估 DNAscope 在不同个体中的变异调用准确性,研究者使用 Sentieon 的 DNAscope 和 DNAseq(符合 GATK 种系最佳实践)管道,使用来自三个 GIAB 样本的公开数据来调用变异:HG002、HG003 和 HG004。
测试涵盖了不同测序深度(15x至36x),并以NIST GIAB高置信度调用v4.2.1为基准。结果显示,DNAscope在所有样本和测序深度下的SNP和INDEL检测性能均优于DNAseq,特别是在30x HG002样本中,SNP和INDEL的F1分数分别达到99.57%和99.46%,总体错误率降低了一半以上。这种在多个样本中的卓越表现证明了DNAscope模型的泛化能力,而非过拟合于训练样本。此外,DNAscope还展现了适应新测序技术的潜力,如之前研究中开发的MGI模型所示,进一步凸显了其在变异检测领域的先进性和灵活性。
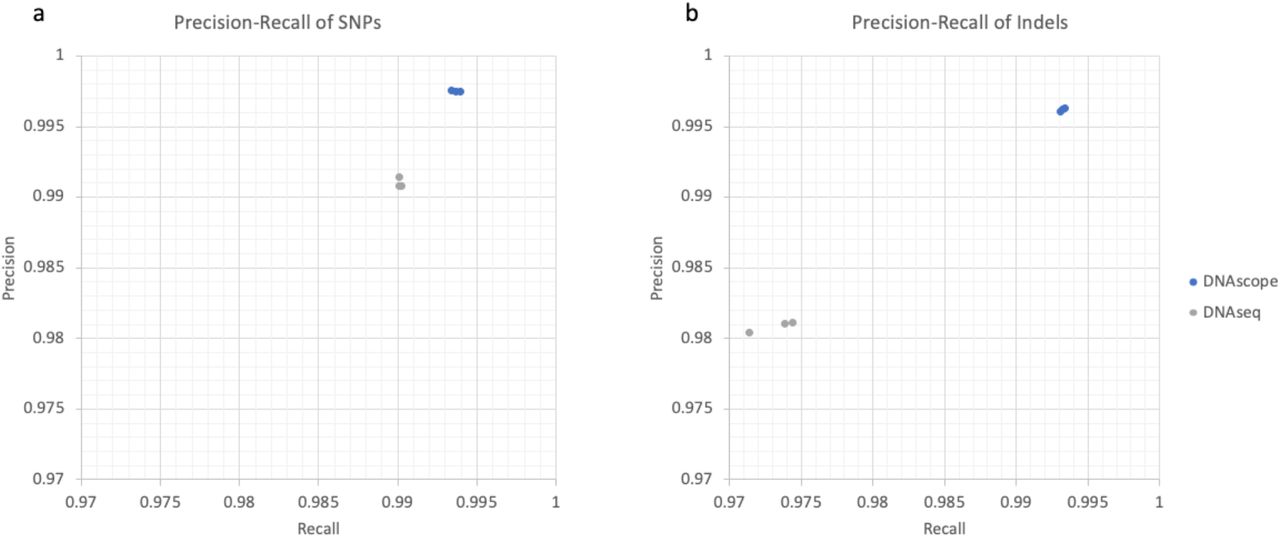
图3 DNAscope 和 DNAseq 的精确召回曲线
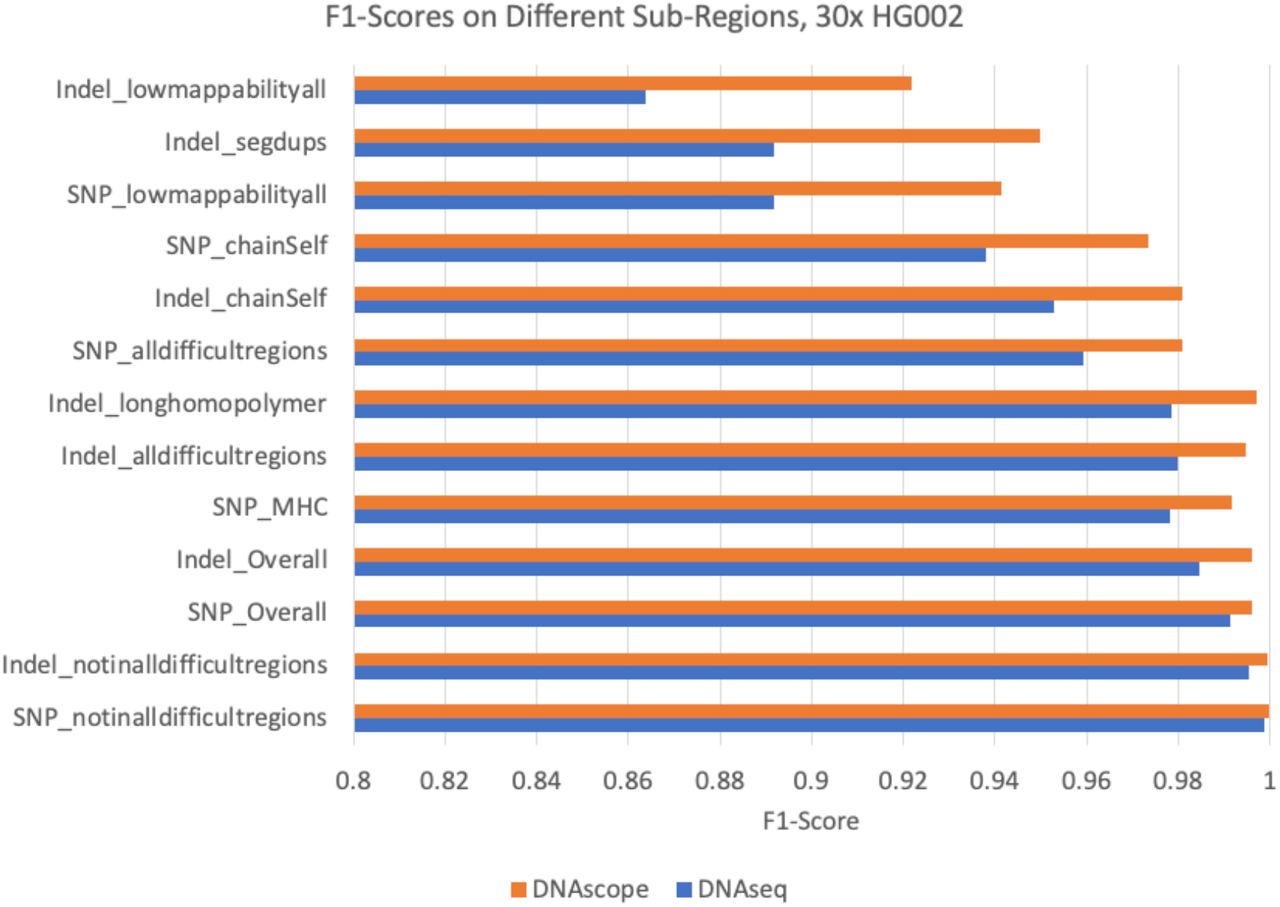
图4 对整个 GA4GH 分层区域进行评估,HG002 深度为 30×
为深入评估变异检测工具的性能,研究组利用GA4GH的分层区域进行了详细分析。这些区域包括低可映射性、分段重复、自链区域、MHC以及综合多种复杂因素的"全难度"区域。结果显示,DNAscope在读数映射困难的区域,如低可映射性、分段重复和自链区域,均明显优于DNAseq。特别是在MHC区域的SNP检测和长同聚物区域的INDEL检测中,DNAscope表现更为出色。这些优势共同导致DNAscope在复杂基因组区域的整体表现优于DNAseq。值得注意的是,即使在相对简单的区域,DNAscope在INDEL检测方面仍保持领先,而在SNP检测方面与DNAseq旗鼓相当。这一全面的分层分析凸显了DNAscope在处理各种复杂基因组区域时的强大能力和灵活性。
研究团队通过对HG002、HG003和HG004的36x测序数据进行抽样,创建了5个不同深度的数据集,以评估变异检测工具在不同测序覆盖度下的性能。结果显示,尽管变异检测准确性通常随覆盖度降低而下降,但DNAscope在低覆盖度条件下仍然保持了优于DNAseq的高准确性。特别值得注意的是,DNAscope在20x覆盖度下的表现始终优于DNAseq在36x覆盖度下的表现。这一发现突显了DNAscope改进的架构和机器学习模型过滤在低覆盖度条件下的显著优势,为高效且经济的变异检测提供了新的可能性。
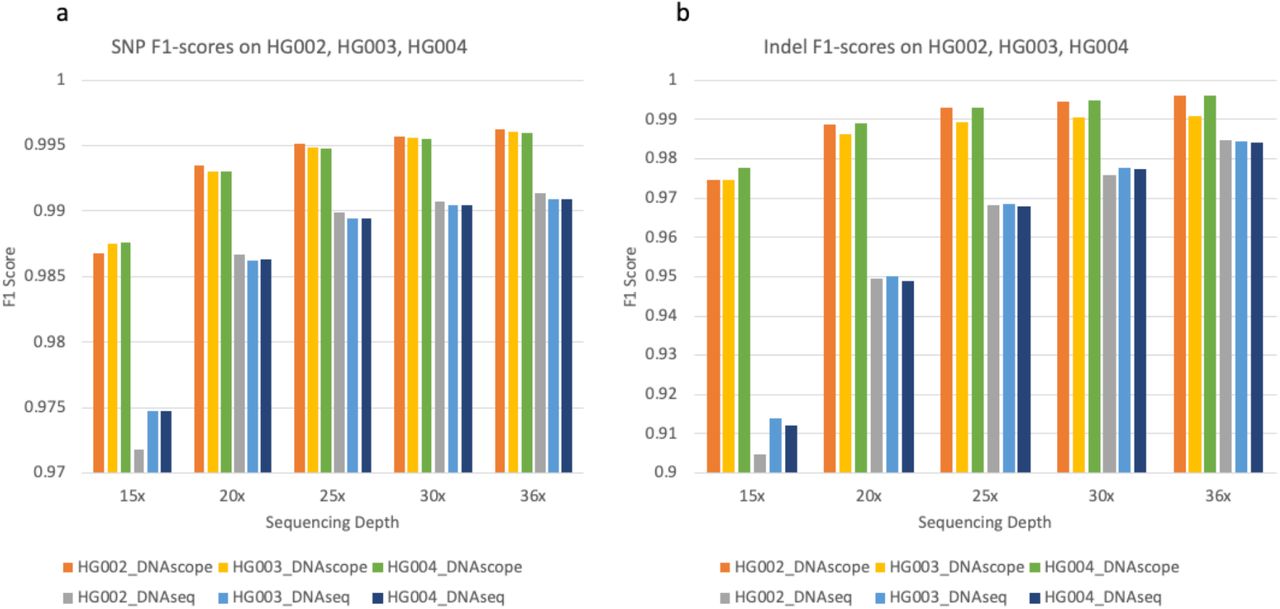
图5 对瓶中基因组样本 HG002、HG003、HG004 进行多深度测序评估
研究探讨了DNAscope贝叶斯模型在非人类和多倍体样本上的表现,特别关注15x覆盖度下的性能。结果显示,尽管整体准确性低于其机器学习模型,DNAscope的贝叶斯模型在INDEL检测方面仍优于DNAseq,而在SNP检测方面两者相当。这表明DNAscope在处理非标准样本时仍具有一定优势,尤其是在INDEL检测方面。
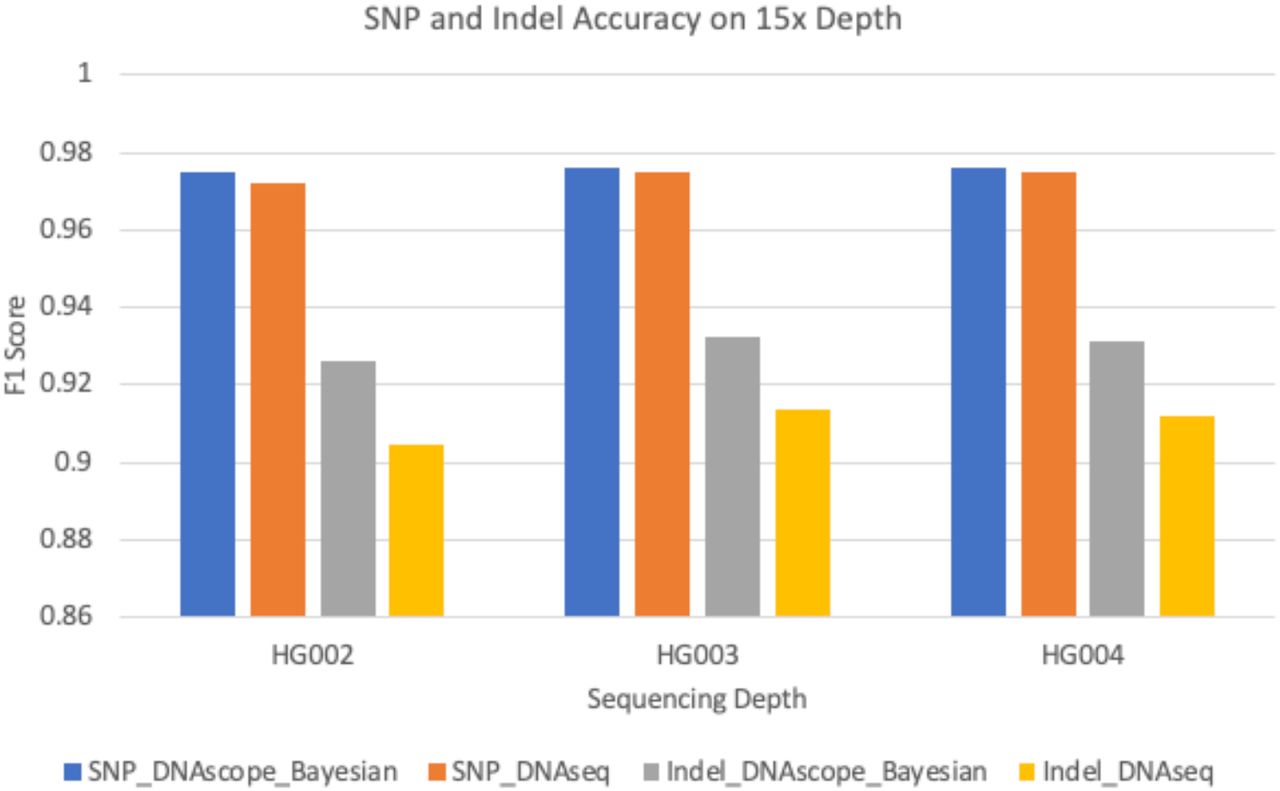
图6 对Genome in a Bottle样本HG002、HG003和HG004在15x测序深度下的评估
在标准化的AWS环境中,对DNAscope进行了性能测试。结果显示,使用96+vCPU处理30x全基因组测序样本时,DNAscope的运行时间不到1小时,与DNAseq相当,比BWA/GATK快5倍。测试还表明DNAscope具有良好的可扩展性,运行时间与线程数几乎呈线性关系。
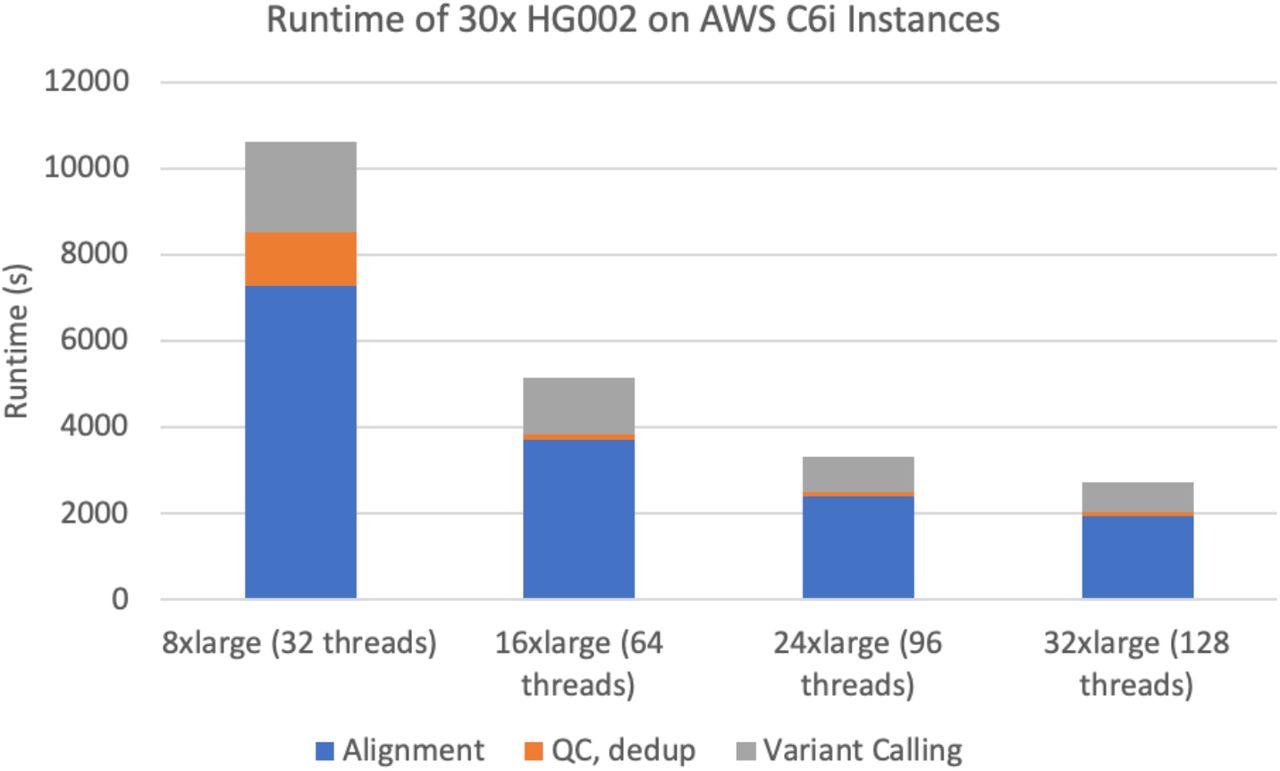
图7 DNAscope 在多个 AWS C6i 实例上的运行时
总结
在这项研究中,研究组证明了DNAscope在不同样本和不同覆盖度水平下都能达到比DNAseq更高的准确性。使用GA4GH分层区域进行的分层分析,能够确认DNAscope在大多数分层区域中都具有高准确性,并突显了DNAscope在插入缺失(indels)和包含变异检测较困难的基因组区域的分层中具有更高的准确性。DNAscope结合了GATK's HaplotypeCaller中使用的成熟数学和统计模型,以及用于变异基因型分析的机器学习方法,在保持计算效率的同时实现了卓越的准确性。