2025.12.24
这篇文章发表于《Medical Image Analysis》期刊(2026 年第 109 卷),由莫纳什大学、悉尼大学等机构学者联合撰写,聚焦脑内血管周围间隙(PVS)的自动分割问题,提出一种基于 nnUNet 的通用型分割工具 PINGU。
Title 题目
01
Perivascular space identification nnUNet for generalised usage (PINGU)
泛用型nnUNet脑血管周围间隙识别系统(PINGU)
Aastract摘要
02
脑血管周围间隙(PVSs)是脑部废物清除系统------淋巴系统的重要组成部分,当其扩大时可在MRI上观察到,其形态与衰老和神经系统疾病相关。手动量化PVS耗时且主观。尽管已有多种深度学习方法用于PVS的自动化分割,但大多数算法是在同质数据集和高分辨率扫描上开发和评估的,这可能限制了它们在临床和研究环境中各种图像质量下的适用性。本研究开发了一个名为PINGU(泛用型nnUNet脑血管周围间隙识别系统)的nnUNet模型,该模型是一种顶尖的、任务驱动型生物医学图像分割深度学习算法。PINGU在来自7个不同数据集、6台不同扫描仪的异构手动分割MRI样本上进行了训练和验证,这些数据集涵盖了各种图像质量和分辨率。PINGU模型在白质(WM)中实现了0.50(标准差=0.15)的体素级Dice分数和0.63(0.17)的聚类级Dice分数,在基底神经节(BG)中实现了0.54(0.11)的体素级Dice分数和0.66(0.17)的聚类级Dice分数。尽管在未见过的"外部"站点数据上的表现有所下降,但PINGU显著优于其他公开可用的算法,尤其在基底神经节区域表现突出。PINGU作为一种广泛使用的PVS分割工具脱颖而出,在与血管疾病和病理高度相关的基底神经节区域具有特别的优势。
Method 方法
03
本研究的数据来源于4项澳大利亚神经影像研究和3个公开的美国数据集(ADNI、HCP、NKI-RS),涵盖了广泛的人群、6种不同扫描仪、1.5T-3T的磁场强度以及多样的图像分辨率。7名经过培训的评估员使用ITKSNAP软件进行PVS的手动分割,并由资深神经放射科医生进行复核。图像预处理采用nnUNet的默认设置,但将目标重采样分辨率统一设为0.8mm3,以保留PVS的精细结构。模型训练采用nnUNet v1.7.0,通过5折交叉验证进行内部评估,并通过"留一站点交叉验证"(PINGU-LOSO)进行外部评估,以全面衡量模型的泛化能力。后处理包括将T1w图像非线性配准到MNI模板空间,以在白质(WM)和基底神经节(BG)区域内计算PVS体积和数量。性能评估指标包括体素级和聚类级的Dice分数、敏感度、阳性预测值以及与手动分割的关联度。此外,还进行了定性视觉评估,并将PINGU的性能与两个公开可用的3D PVS分割算法(SHIVA-PVS和WPSS)进行了比较。
Discussion讨论
04
本研究提出的基于nnUNet的PINGU模型,在临床T1w MRI图像上实现了对PVS的有效分割,在WM和BG中的Dice分数分别为0.50和0.54。该算法对未见过的MRI图像表现出优于其他公开可用分割算法的泛化能力,尤其是在基底神经节区域。研究结果表明PINGU能够发现约一半的手动标记PVS(聚类敏感度为0.55-0.56),且体素和聚类测量精度适中(PPV=0.74-0.77),其分割结果与手动分割结果高度相关,因此能准确反映扩大的PVS负荷,尽管绝对体积可能被低估。我们认为PINGU可适用于各种疾病状态和图像属性的数据集,尤其是在仅有T1图像的情况下。PINGU的优越性能归因于多样化的训练数据、nnUNet的自配置特性和强大的数据增强策略。外部验证中Dice分数的下降突显了深度学习算法泛化到新数据特征的挑战,也进一步凸显了PINGU的相对优势。基底神经节区域PVS分割性能的提升尤为重要,因为该区域的PVS与血管病理学密切相关。本研究的局限性包括手动PVS分割固有的困难和不确定性、各数据集样本量不均以及高数据方差下模型可能欠拟合。然而,PINGU生成完整的3D PVS分割,在未见过数据上表现良好,仅需T1w图像,且对基底神经节的优化分割,都突显了其独特的优势。
Conclusion结论
05
本研究成功开发并验证了PINGU,一个基于nnUNet的3D脑血管周围间隙(PVS)分割算法。PINGU通过在包含异构数据集和多种扫描仪数据的广泛训练集上进行训练,显著提升了模型的泛化能力。实验结果表明,PINGU在未见过的外部数据集上表现出强大的性能,并且在基底神经节区域的分割效果明显优于其他现有的公开算法。这使得PINGU成为一个具有广泛临床和研究应用价值的PVS分割工具,尤其对于研究神经血管病理学中的淋巴系统功能具有重要意义。
Results结果
06
在内部验证(5折交叉验证)中,PINGU在白质(WM)中的平均体素级Dice分数为0.50(标准差=0.15),在基底神经节(BG)中为0.54(0.10)。相应的聚类级Dice分数在WM中为0.63(0.17),在BG中为0.66(0.17)。敏感度和精确度均表现良好,手动分割与PINGU分割之间存在强相关性(r=0.69-0.84),表明PINGU能准确反映PVS负荷。视觉评估显示,PINGU分割的精确度很高(WM为94.7%,BG为88.7%),提示PINGU可能检测到了一些手动评估员遗漏的真实PVS,尽管它可能略微低估了PVS的实际范围。在外部验证(留一站点交叉验证)中,尽管未见过的站点数据上的Dice分数显著降低(WM体素级Dice为0.38,BG体素级Dice为0.36),但PINGU的性能仍远优于SHIVA-PVS(WM体素级Dice为0.18,BG体素级Dice为0.10)和WPSS(WM体素级Dice为0.30,BG体素级Dice为0.20),特别是在基底神经节区域。对模型参数的变体测试表明,nnUNet v2版本、自动设置体素间距以及1mm体素间距的性能均有所下降。
Figure 图
07
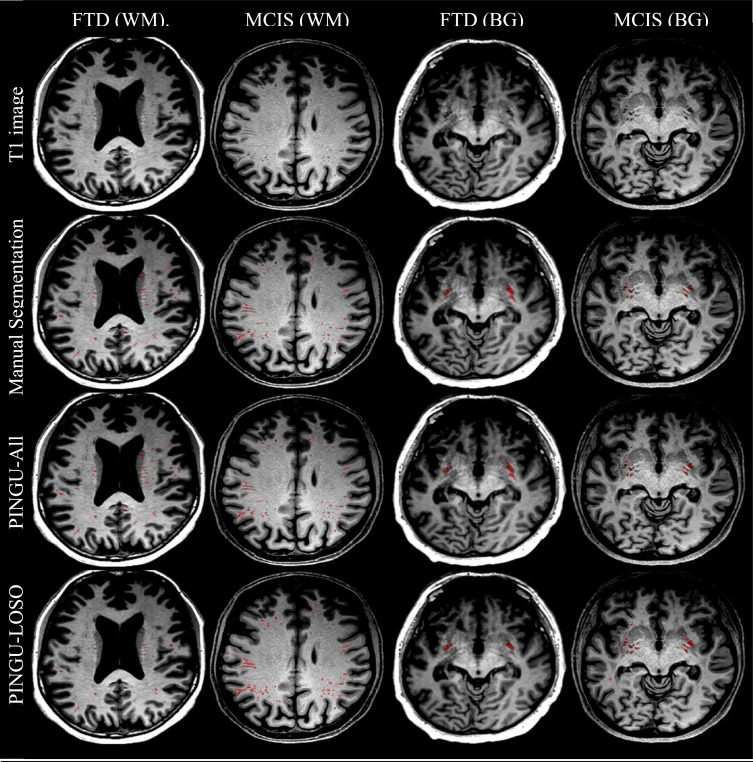
图1.PINGU-All和PINGU-LOSO在T1图像上的叠加,并与"高质量"数据集(MCIS)和"低质量"数据集(FTD)的手动分割进行比较。显示切片是根据手动分割中白质(WM)和基底神经节(BG)中PVS体积最大的切片自动选择的。
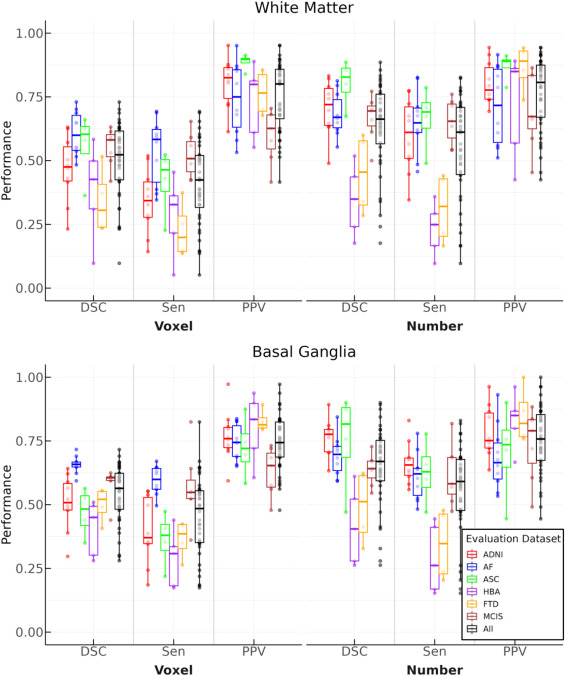
图2.内部验证。PINGU在所有站点数据上训练和评估的性能,包括白质(上)和基底神经节(下),DSC:Dice分数,Sen:敏感度,PPV:阳性预测值/精确度,vox:体素级别指标,num:聚类级别指标。
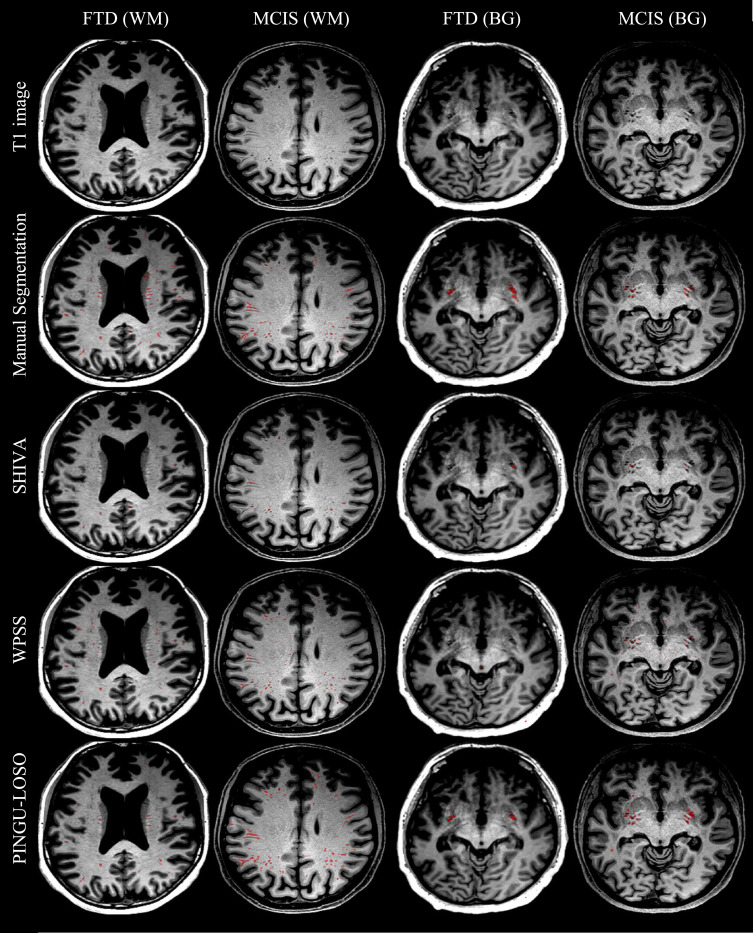
图3.三种深度学习算法分割结果在T1图像上的叠加,并与"高质量"数据集MCIS和"低质量"数据集FTD的手动分割进行比较。显示切片是根据白质(WM)和基底神经节(BG)中PVS体积最大的切片自动选择的。
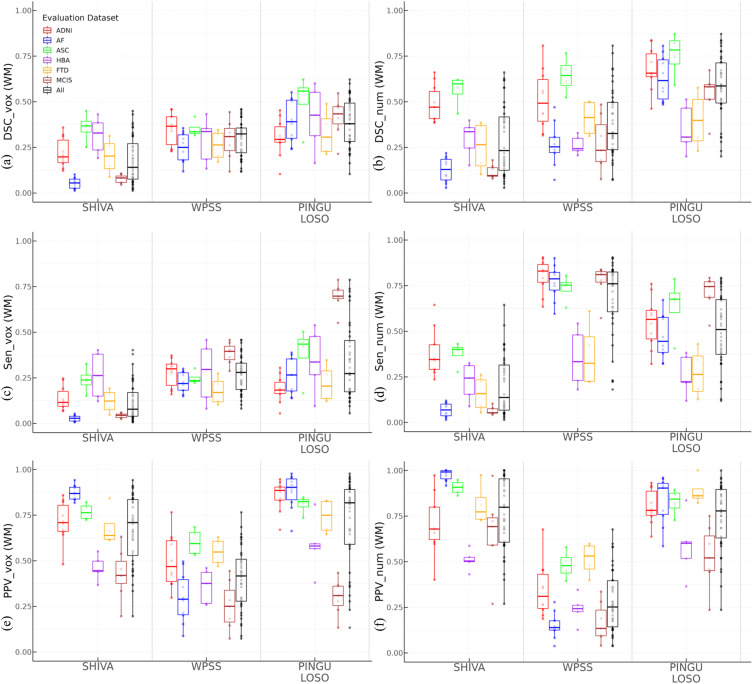
图4.外部验证,算法比较(白质(WM)):在未见过站点数据上评估的算法性能。DSC:Dice分数,Sen:敏感度,PPV:阳性预测值/精确度,vox:体素级别指标,num:聚类级别指标。
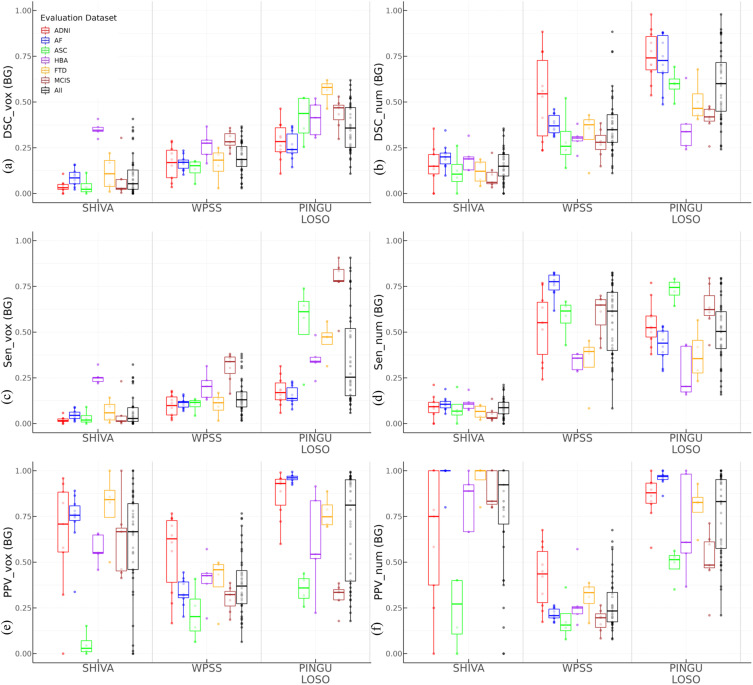
图5. 外部验证,算法比较(基底神经节(BG)):在未见过站点数据上评估的算法性能。DSC:Dice分数,Sen:敏感度,PPV:阳性预测值/精确度,vox:体素级别指标,num:聚类级别指标。